第一作者联系方式: E-mail: muchuansanshui@163.com
自然衰老提高了植物对环境的适应性, 是其生长发育的重要生命历程, 但在农业生产中, 叶片一旦早衰, 将极大影响作物的产量和品质。为探索水稻叶片衰老的分子机制, 我们对EMS诱变获得的一个早衰突变体 esl6进行了研究。田间种植情况下, 四叶期之前, esl6与野生型无明显差异, 之后心叶发育成完整叶后叶尖黄化, 叶基部保持正常绿色, 一直持续到开花期; 在灌浆期, esl6的所有叶片均不同程度地黄化早衰, 且叶片上部的衰老程度明显严重于叶片基部。衰老部位细胞结构异常, 主要表现为细胞膜破裂、液泡变大和细胞器不完整等, 叶绿体中基质类囊体破裂, 含有较多的淀粉粒。与野生型相比, esl6叶尖衰老部位的SOD、CAT和POD活性以及超氧阴离子O2܋、H2O2和羟自由基·OH含量均极显著升高。早衰不仅导致 esl6叶片光合色素含量和净光合速率极显著降低, 还引起 esl6的植株变矮和叶片变短, 倒一和倒二节间极显著变短是导致 esl6植株矮化的主要原因。遗传分析表明该性状受一对隐性核基因调控, 利用西大1A/ esl6的F2分离群体, 最终将调控基因定位在第9染色体203 kb的物理范围内, 为下一步基因的克隆和功能研究奠定了基础, 有利于水稻叶片衰老分子机制的阐释。
As an essential process in life, natural senescence is necessary to adapt plant to environment diversity, while earlier senescence could reduce yield per unit and cause inferior quality in crop production. Therefore, it is significant to elucidate senescence molecular mechanism in plant. Here, we reported a novel rice mutant esl6derived from the progeny of EMS-induced restorer line Jinhui 10, which senescent peculiarity was observed at the early stage of life. In detail, cultivated under the paddy field, the esl6 had no obvious difference with the wild type before the 4-leaf stage, while after that the whole leaf blade of esl6 displayed chlorosis in the tip and kept normal green in the base until the flowering stage. Subsequently, all leaf blades in the esl6 demonstrated chlorosis and senescence, still more severe at the upper position. Observation by scanning electron microscope showed that cell structures in the senescent location of esl6 leaf blade were abnormal and filled with ruptured cell membranes, enlarged vacuoles and broken organelles such as the chloroplasts containing incomplete stroma thylakoids and excessive starch grains. Meanwhile, early senescence significantly lessened photosynthetic pigment contents and photosynthetic rate. The activities of SOD, CAT, and POD raised and the contents of O2܋, H2O2, and ·OH increased in the esl6 leaf tip, and all of the differences led to the extremely significant level compared with those of the wild type. Additionally, the mutational plant showed semi-dwarfism and shorter leaf blades, the first and second internodes decreased to the extremely significant level in statistics. Genetic analysis suggested that the mutational traits were controlled by a recessive nuclear gene. The gene was finally mapped on chromosome 9 with 203 kb physical distances between Indel markers Sind09-3 and Sind09-4 on the basis of F2 generation of Xida1A/ esl6. All of these provide a foundation for ESL6 cloning and function analysis and then are beneficial to ascertaining the molecular mechanism of senescence in Oryza sativaL.
衰老是植物生长发育的最后一个阶段, 受遗传和外界环境因子的共同影响, 在衰老过程中, 叶片等器官中的营养物质转移到生殖器官, 是植物长期进化过程中的一种自我保护形式[1]。水稻是世界上最重要的粮食作物之一, 也是单子叶模式植物, 叶片一旦早衰, 将极大影响其生长发育, 进而影响产量和品质, 因此, 研究叶片早衰机理不仅可以从分子水平阐释植物叶片的生长发育, 而且对培育高产、优质水稻新品种具有重要的应用价值。如研究发现, 水稻生长发育过程中95%的物质来自光合作用[2], 灌浆过程中, 叶片适度推迟1 d衰老, 水稻产量理论上可增加2%, 实际增产1%左右[3]。
植物衰老的主要表现特征是叶片黄化死亡, 其内RNA、蛋白质和叶绿体降解。全基因组分析发现, 叶片衰老相关的调控基因众多, 如在拟南芥自然衰老过程中, 有2491个基因在转录水平改变[4], 在水稻剑叶衰老过程中, 则检测到815个表达上调的EST[5]; 截至2014年, 在LSD2.0网站中(http://www. eplantsenescence.org/)收录了3745个拟南芥、882个香蕉、256个小麦、132个水稻等衰老相关基因[6]。MicroRNAs (miRNAs)是真核生物中调控细胞衰老的重要因子, 在水稻叶片衰老过程中, 检测到一系列miRNAs与其靶基因的表达变化, 表明miRNAs也参与了水稻叶片的衰老调控[7]。这些结果为研究植物衰老基因之间的网络调控提供了便利。
目前研究表明植物衰老是一个复杂的生物学过程, 涉及叶绿素、激素、自由基等的合成与代谢。通过基因克隆和全基因组分析等技术手段, 已构建了植物衰老分子调控网络的部分结构, 但机理还远不清晰[8]。部分研究者认为植物衰老受QTL调控, 如Abdelkhalik等[9]利用籼粳交群体在第6染色体定位到1个、在第9染色体定位到2个主效QTL; Yoo等[10]则在第7和第9染色体上分别定位到1个和2个延迟衰老的QTL。但多数研究者将衰老当质量性状研究, 已利用突变体定位了ospse1[11]、lmes[12]、sms1[13]、Psl1[14]、psl2[15]、Pse(t)[16]、Psl3[17]、es-t[18]、esl2[19]等十几个早衰基因。我们从缙恢10号的EMS诱变库中鉴定到一个新的早衰突变体, 暂命名为esl6 (early senescence leaf 6, esl6), 本文对其进行了表型鉴定、理化分析和基因定位等研究。
利用EMS诱变水稻籼型恢复系缙恢10号, 从其后代中鉴定得到一个稳定遗传的早衰突变体esl6, 连续种植5代, 突变性状均稳定遗传。配制西大1A/esl6的杂交组合, 利用F1和F2群体进行遗传鉴定和基因定位。西大1A是西南大学水稻研究所选育的一个三系不育系, 表型正常。灌浆期从种植小区中间选取10株突变体和10株野生型, 调查株高和各节间长, 同时调查倒一、倒二和倒三叶的叶片长和宽, 成熟期调查农艺性状。利用Statistical Package for the Social Sciences 19.0 (SPSS)软件分析显著性。
孕穗期, 晴天上午9:30— 10:30, 利用LI-6400型便携式光合作用测定仪分别测定长势相对一致的esl6及野生型倒二叶的叶片基部(持绿)和尖部(早衰)的光合速率。测量时, 使用红、蓝光源, 光强恒定为1200 μ mol m-2 s-1, 温度为30℃, CO2浓度为空气中的浓度, 湿度为大气中的湿度[20]。然后取相应的基部和尖部叶片, 利用95%乙醇提取叶绿素, 分光光度计测量波长为470、646和663 nm下的光吸收值, 参照Wellburn[21]的方法计算叶绿体色素含量。每个材料测10株, 取平均值。
抽穗期, 取突变体esl6和野生型叶片衰老与绿色部位, 经戊二醛和锇酸双重固定后, 利用不同梯度的乙醇逐级脱水, 再置换和包埋, 制超薄切片后, 以醋酸双氧铀和柠檬酸铅液双重染色, H600型透射电镜观察叶片细胞超微结构[17]。
抽穗期, 取突变体esl6和野生型长势相对一致的倒二叶衰老和绿色部位, 利用南京建成生物工程研究所生产的试剂盒, 参照说明书测定过氧化氢(H2O2)、超氧阴离子(O2܋)、羟自由基(· OH)的含量以及过氧化氢酶(CAT)、超氧化物歧化酶(SOD)和过氧化物酶(POD)的活性, 各重复3次。
参照Michelmore等[22]的BSA法筛选连锁标记, 从F2植株中分别选取10株正常和10株突变株剪取等量叶片, 构建正常基因池和突变基因池, 采用改良的CTAB法[23]提取亲本、基因池和F2突变单株的基因组DNA。
参照http://www.gramene.org/microsat合成SSR引物, Indel标记根据高通量测序获得的西大1A和缙恢10号序列差异, 利用NTI vector 11.0软件设计开发。PCR总体系12.6 μ L, 包含1.25 μ L的10× PCR buffer、0.75 μ L的25 mmol L-1 MgC12、0.5 μ L的2.5 mmol L-1 dNTPs、8.0 μ L的ddH2O、1.0 μ L的10 μ mol L-1引物、1.0 μ L的模板DNA和0.1 μ L的5 U μ L-1Taq DNA聚合酶。PCR程序为94℃预变性5 min, 然后94℃变性30 s、55℃退火30 s、72℃延伸1 min, 35个循环后再72℃充分延伸10 min。PCR产物经10%的非变性聚丙烯酰胺凝胶电泳后, 快速银染法染色观察[24]。
西大1A× esl6的F2作图群体中, 具有西大1A带型的单株记为A, 具有esl6带型的单株记为B, 具有F1带型的单株记为H。用MapMaker3.0分析数据和作图, 用Kosambi函数将重组率转化为遗传距离。
田间种植条件下, 一叶期至四叶期, 突变体esl6与野生型缙恢10号无明显差异; 四叶期之后,
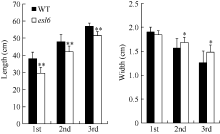
esl6的叶片发育完整后叶尖部即黄化早衰, 基部则保持正常的绿色, 该现象一直持续到开花期, 之后, esl6的整个叶片均黄化早衰, 且一张叶片的尖部比基部更为严重, 野生型植株在相同时期内则一直保持绿色(图1)。此外, 野生型的株高为106.84 cm, 突变体的则为94.60 cm, 与野生型相比, esl6的株高极显著降低。进一步分析发现, esl6的穗长由野生型的25.58 cm下降为22.62 cm, 达到显著差异水平; 倒一和倒二节间极显著降低, 分别下降了39.95%和82.78%; 倒三和倒四节间则显著升高(图1-E)。
开花期, 剑叶、倒二叶和倒三叶的叶片长度均显著变短, 分别下降了28.96%、14.18%和10.61%, 叶片宽度除剑叶变化不显著外倒二叶和倒三叶均显著变宽, 分别增宽了7.69%和17.46% (图2)。农艺性状分析发现, esl6的穗实粒数、一次枝梗数和二次枝梗数极显著低于野生型, 有效穗数、结实率和千粒重则无显著变化(表1)。
| 表1 突变体esl6和野生型(WT)之间的农艺性状分析 Table 1 Agronomic analysis of the esl6 and wild type (WT) |
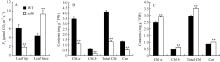
孕穗期, 叶尖衰老部位, esl6的净光合速率为1.86 μ mol CO2 m-2 s-1, 极显著低于野生型的6.14 μ mol CO2 m-2 s-1; 而在叶片基部, esl6的净光合速率为9.31 μ mol CO2 m-2 s-1, 极显著高于野生型的4.42 μ mol CO2 m-2 s-1(图3-A)。光合色素含量分析发现, 叶尖衰老部位esl6的光合色素含量极显著降低(图3-B); 叶片基部, esl6的光合色素含量则极显著升高(图3-C), 与野生型相比, 叶绿素a、叶绿素b、总叶绿素和类胡萝卜素含量分别升高了16.04%、36.96%、19.32%和18.82%。光合色素含量变化与净光合速率变化趋势一致。
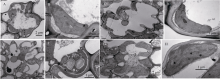
透射电镜观察发现, 突变体esl6的叶片基部细胞(图4-A)和野生型(图4-C)相比无明显差异, 均具有完整的细胞核、线粒体、叶绿体等细胞器; 叶绿体内, esl6尽管也含有完整的基质类囊体(图4-B), 但与野生型(图4-D)相比, 基粒片层的堆积较不规则。叶尖部位, esl6的细胞形状与野生型相比无差异, 但细胞结构遭到严重破坏, 主要表现为细胞膜破裂、液泡变大和细胞器不完整(图4-E, G); 在叶绿体内, esl6含有较多的淀粉粒, 基质类囊体已破裂, 无完整的基粒片层(图4-F), 而野生型的叶绿体(图4-H)则表现正常。
抽穗期, 和野生型相比, esl6叶尖部和叶基部的H2O2、O2܋、· OH都极显著升高(图5-A, B, C), 其中叶尖部分别升高了99.67%、56.20%、119.33%; 叶基部分别升高了37.54%、34.17%、46.45%。叶基部升高的幅度比叶尖部升高的幅度低的原因可能是叶基部衰老程度没有叶尖部严重。esl6叶尖部CAT、SOD、POD的活性分别升高了50.28%、199.83%、54.48% (图5-D, E, F), 差异均达到极显著的水平; esl6叶基部CAT、POD的活性分别下降了17.50%、45.57% (图5-D, F), 差异分别达到显著和极显著水平; esl6叶基部SOD的活性则没有变化(图5-E)。
突变体esl6和不育系西大1A的杂交组合, F1植株表型正常, F2世代中出现叶色正常和叶尖黄化早衰2种植株类型。调查分析发现, 1207个西大1A/esl6杂交组合的F2单株中, 有921株叶色正常, 286株叶
片早衰, χ 2检测表明正常和突变株符合3︰1的分离比[χ 2= 1.03< 3.84 (χ 2(0.05, 1))], 暗示esl6的叶片早衰性状受1对隐性核基因调控。
用西南大学水稻研究所保存的96对均匀分布在水稻12条染色体上且在西大1A和缙恢10号之间呈现多态性的核心SSR和Indel标记进行连锁分析, 发现位于第9染色体上的SSR标记RM201和Indel标记YI09-5在正常池和突变池之间也存在明显的条带差异, 暗示可能存在连锁关系。对F2定位群体中的10株正常株和24株突变株的检验进一步确定了连锁关系, 并将ESL6初步定位在RM201和YI09-5之间, 遗传距离分别为15.7 cM和8.7 cM。
在初步定位区间内, 进一步设计了18对Indel标记, 其中5对在亲本西大1A和esl6之间呈现多态性, 分别命名为Sind09-1、Sind09-2、Sind09-3、Sind09-4和Sind09-5 (表2)。利用这5对多态性引物进一步分析286株F2隐性单株发现, 前3个标记的交换株数量分别为12、6和4个, 后2个标记的交换株数量分别为1和5个, 且前3个和后2个标记的交换株不同, 从而最终将ESL6定位在第9染色体Indel标记Sind09-3和Sind09-4之间203 kb的物理范围内(图6)。
| 表2 在亲本西大1A和esl6之间具有多态性的Indel引物序列 Table 2 Primer sequences of Indel markers with polymorphisms between the parents of Xida 1A and esl6 |
水稻叶片早衰突变体植株表型一般为叶片黄化, 叶绿素含量降低。除主要特征外, 部分早衰突变体叶片还出现褐色斑点、花粉育性降低等, 且早衰时期和表型强弱各异。本研究中的esl6突变体, 1~3叶断乳期植株表型正常, 四叶期之后叶尖部位逐渐黄化, 灌浆期之后尤为严重(图1), 黄化部位的细胞膜破裂、液泡变大、细胞器不完整(图4), 表明esl6是一个叶片早衰突变体。进一步分析发现, 叶片早衰导致esl6的株高极显著变矮, 叶片长度极显著变短(图1-E和图2)。穗长、倒一节间和倒二节间显著或极显著变短是导致esl6株高变矮的主要原因。
植物白天形成的光合产物大部分以淀粉的形式储存在叶绿体中, 晚上则分解供应植物的生长发育。阻断淀粉合成或分解途径可影响植物的正常生长发育[25]。淀粉粒降解途径可进一步分为淀粉粒降解的起始、支链和直链淀粉分解成低聚麦芽糖及衍生物2个阶段[26]。而这一过程受昼夜循环、淀粉磷酸化和酶活性等的调节, 尽管已经克隆了一些调控基因, 但其遗传机制还很不清晰[27]。细胞超微结构观察发现, 在突变体esl6的叶尖衰老部位, 叶绿体中有大量的淀粉粒积累, 而绿色部位和相应的野生型部位则没有观察到该现象(图4), 暗示esl6的叶片早衰可能与淀粉的降解途径障碍相关。
在植物中, 目前已克隆的叶片衰老相关基因, 多参与过氧化反应、叶绿素/体降解、逆境胁迫、激素合成及信号传导等途径。如, NOE1编码一个水稻过氧化氢酶OsCATC, 功能缺失突变体内过氧化氢(H2O2)含量增加, 进而诱导叶片细胞死亡[28]。NYC1编码叶绿素b还原酶亚基, 功能缺失后叶绿素b、捕光复合体II以及类囊体基粒的降解被抑制, 从而延缓叶片衰老[29]。OsDos编码CCCH-型锌指蛋白, 负调控水稻叶片的衰老[30]。OsNAP受到脱落酸(ABA)的特异性诱导, 通过直接调控叶绿素降解、营养再转运及其他衰老相关基因的表达调控叶片的衰老进程[31]。NAC是植物特有的一类转录因子, 在拟南芥中已经报道了多个正向或负向调控叶片衰老的基因。近来研究发现, 水稻中的NAC转录因子ONAC106负调控叶片衰老, 过量表达植株不仅延缓了叶片早衰, 而且提高了植株对盐胁迫的耐受性[32]。
植物叶片衰老既受光、温、盐、干旱等各种环境胁迫的影响, 也受乙烯、脱落酸、邻苯酚甲酸、细胞分裂素等植物自身的调节, 是一个复杂的生命历程[33]。根据水稻基因组注释计划(RGAP)和Gramene等网站, 在ESL6基因203 kb的物理定位范围内, 有45个注释基因, 其中, 有2个编码C2H2型锌指蛋白、1个编码钾离子转运蛋白、1个编码E3泛素连接酶、1个编码NADPH还原酶。生物信息学分析发现, 这些基因中, 尚未有调控叶片衰老的相关报道。已有研究表明, C2H2型锌指蛋白和钾离子转运蛋白可参与盐胁迫应答反应, 也调控细胞器的发育[34]。E3泛素连接酶的功能具有多样性, 如TUD1编码的U-box家族的E3泛素连接酶, 仅参与油菜素内酯的应答[35], OsSRFP1编码的E3泛素连接酶, RNAi植株增强了对盐、低温及氧化胁迫的耐性[36]。在水稻中, OsPORA和OsPORB分别编码NADPH:原叶绿素酸酯氧化还原酶A和B, OsPORB/FGL功能缺失突变体的叶片出现黄白花斑和坏死病斑[37]。而拟南芥中的NADPH还原酶编码基因NTRC受光周期诱导, 其功能缺失突变体叶片表现黄化且淀粉粒含量增高[38]。因此推测, NADPH还原酶编码基因最可能是ESL6的候选基因, 但尚需进一步研究。
四叶期之前, esl6与野生型植株无明显差异, 之后, esl6的叶片一旦发育完整, 叶尖即出现黄化早衰, 叶基部保持正常的绿色, 该现象一直持续到开花期。在灌浆期, esl6的整个叶片均出现不同程度的黄化早衰, 且叶片上部的衰老程度明显比叶片下部严重。透射电镜观察发现, 衰老部位的细胞结构异常, 主要表现为细胞膜破裂、液泡变大和细胞器的不完整, 叶绿体内含有较多的淀粉粒, 基质类囊体破裂。此外, esl6还表现为植株变矮和叶片变短, 倒一和倒二节间极显著变短是导致esl6植株矮化的主要原因。该性状受单隐性核基因调控, 利用F2分离群体, 本文已将调控基因定位在第9染色体203 kb的物理范围内, 为下一步基因的克隆和功能研究奠定了基础。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|