第一作者联系方式: E-mail:xingzhang.1989@163.com
小麦高分子量麦谷蛋白亚基(high molecular weight glutenin subunit, HMW-GS)由 Glu-A1、 Glu-B1、 Glu-D1位点中含有的复等位基因编码, 评价和优化 Glu-1位点组合是认识与改良HMW-GS表达与功能的重要途径。本研究创制了以小偃81为背景的HMW-GS基因完全缺失突变体DLGlu1。将DLGlu1与加拿大优质强筋小麦品种Glenlea杂交, 结合后代幼胚培养与分子标记辅助选择技术, 在BC3F3种子中快速鉴定出来自Glenlea的 Glu-A1a、 Glu-B1al和 Glu-D1d位点不同组合的7种渗入系材料, 可进一步发展成一套完整的 Glu-1位点有差异的近等渗入系。本研究表明, DLGlu1可用于 Glu-1位点近等渗入系的快速创制, 对 Glu-1位点功能研究和改良具有重要价值。
Wheat ( Triticum aestivum L., AABBDD) high molecular weight glutenin subunits (HMW-GS) were encoded by the genes located in Glu-A1, Glu-B1, and Glu-D1 loci. Evaluation and optimization of the combination of HMW-GS are very important to understand Glu-1 functions. In this study, we constructed a HMW-GS deletion mutant, DLGlu1 with Xiaoyan 81 background, and crossed it with Glenlea, a Canada elite wheat variety with superior end-use quality. Combining the technologies of wheat embryo culture and molecular marker-assisted selection (MAS), we obtained seven introgression lines containing Glenlea Glu-A1a, Glu-B1al, and Glu-D1d loci, which can be developed as a complete set of near-isogenic introgression lines possessing Glenlea different HMW-GS genes. Our study indicated that the Glu-1 deletion mutant DLGlu1 is of great value in the fast development of Glu-1 near-isogenic introgression lines and the study and utility of wheat Glu-1.
随着测序技术的发展, 一些模式植物和重要农作物基因组相继被测序, 并绘制出基因组精细图[1, 2]。小麦(Triticum aestivum L., 2n = 42, AABBDD)基因组十分庞大(17 Gb, 约为水稻基因组的40倍), 基因组重复序列比重较高, 同时携带A、B和D三套亚基因组, 因此小麦基因组测序和功能基因组研究一直是极具挑战性的工作[3]。最近已经通过测序获得普通小麦及其近缘物种的基因组草图[4, 5, 6], 为进一步认识小麦功能基因和加快小麦产量和品质育种进程提供了新的机遇。
创制基因缺失突变体是研究基因功能的重要方法之一。采用T-DNA插入和EMS诱变方法, 已得到拟南芥和水稻几乎每个基因的缺失突变体, 这些突变体是用来研究相应基因功能缺失效应的重要材料[7, 8]。普通小麦是异源六倍体, 对大多数小麦基因而言, 同时存在6个同源拷贝, 每2个拷贝来自同一亚基因组, 理论上只有把每个基因的6个拷贝同时突变才可以造成功能缺失, 这种基因组冗余对创制小麦基因功能缺失突变体是个巨大挑战。然而, 基因组冗余特性使小麦基因组可以耐受更高频率的突变, 即可以在一个较小的突变群体内检测到较多基因的突变, 同时也使得小麦基因组可以耐受更大片段的缺失[9]。如Fitzgerald等[10]通过检测4500份小麦离子束辐射诱变M2材料, 共检测出9个TaPFT1基因缺失突变体, 缺失区间达到数十兆碱基。Yang等[11]检测了5600份小麦离子束辐射诱变M2材料, 共发现7个Glu-1缺失突变体, 缺失范围对应水稻156~345 kb的区间。这些结果说明采用物理诱变方法可以快速创制大片段缺失突变体用于小麦功能基因研究。
相对于玉米和水稻等作物, 小麦面粉可以制作成面包、馒头、面条、饼干和糕点等多种食品, 主要是因为小麦面粉含有独特面筋成分, 赋予面团黏弹性和延展性。小麦面筋主要由醇溶蛋白(gliadin)和谷蛋白(glutenin)组成, 其中谷蛋白又可以分为高分子量麦谷蛋白(high molecular weight glutenin subunit, HMW-GS)和低分子量麦谷蛋白(low molecular weight glutenin subunit, LMW-GS)两大类[12]。醇溶蛋白对形成面团的延伸性起主要作用, 而麦谷蛋白是影响面团弹性的重要因素, 决定面包烘烤品质, 其中HMW-GS与小麦面包烘烤品质的关系尤为密切[12]。HMW-GS的编码基因位于普通小麦第一部分同源群1A、1B、1D长臂的Glu-1位点, 分别称为Glu-A1、Glu-B1和Glu-D1[13]。每个Glu-1位点编码2个在遗传上紧密连锁的HMW-GS亚基, 一个为分子量较大的x-型亚基, 一个为分子量较小的y-型亚基。x-型和y-型亚基蛋白序列均由信号肽、N-端结构域、中间重复区和C-端结构域组成[12, 13]。HMW-GS在不同小麦品种中存在着丰富的变异, 这些等位变异不仅造成其表达及在SDS-PAGE凝胶电泳的迁移率差异, 也造成它们功能上的差异。如在Glu-D1位点, Glu-D1d (表达Dx5和Dy10亚基)功能显著优于Glu-D1a(表达Dx2和Dy12亚基)[14]。
本实验室在前期研究中创制了普通小麦品种小偃81的离子束辐射诱变群体, 筛选到Glu-A1、Glu-B1和Glu-D1的单缺失突变体, 并在同一遗传背景下评价了3个同源位点的功能差异[11]。在此基础上, 我们采用聚合育种技术获得了小偃81的Glu-1完全缺失突变体DLGlu1。本研究旨在评价DLGlu1作为材料基础快速获得不同Glu-1等位渗入系, 并用于Glu-1功能研究的前景。
将普通小麦品种小偃81 (1, 14+15, 2+12)分别缺失Glu-A1a、Glu-B1h及Glu-D1a的突变体DLGluA1、DLGluB1和DLGluD1 [11]相互杂交, 获得Glu-1完全缺失突变体DLGlu1。Glenlea (2* , 7OE+8, 5+10)是加拿大优质强筋小麦品种, 携带Glu-A1b、Glu-B1al和Glu-D1d优异等位变异[15]。普通小麦中国春及其缺体-四体材料[16]用于分子标记的定位验证。
2014年将Glenlea (父本)和突变体DLGlu1 (母本)种植于中国科学院遗传与发育生物学研究所温室(北京), 并进行杂交, 利用分子标记筛选目标单株, 与轮回亲本DLGlu1回交至BC3F1代, 再自交一次; 结合幼胚培养技术[17], 于2015年获得BC3F2群体; 随机选取3个位点均为Glenlea基因型(Glu-A1b、Glu-Bal1和Glu-D1d)的3份材料(BC3F2-6、BC3F2-16、BC3F2-56)自交, 产生BC3F3种子, 用于基因分型分析。
利用NCBI网站(http://www.ncbi.nlm.nih.gov/) Glu-A1序列信息和普通小麦中国春基因组序列信息(http://plants. ensembl.org/Triticum_aestivum/Info/Index), 根据Glu-A1转座子插入多态性开发Glu-A1特异分子标记Xrj5, 并采用Primer Premier 5.0设计引物。Glu-B1特异分子标记为BxMAR, 位于Bx基因上游750 bp核基质结合区(matrix attachment region, MAR) [18]; Glu-D1特异分子标记为Xrj2, 位于Glu-D1座位1Dx和1Dy基因之间[19]。Glu-A1、Glu-B1和Glu-D1特异分子标记引物序列及在不同小麦品种中预期扩增产物如表1所示。
| 表1 本研究所用的分子标记 Table 1 Molecular marker primers used in this study |
从每个系取30粒BC3F3代种子, 用解剖刀切取有胚端的半粒放入营养钵发苗, 在小麦一叶一心期剪取第1片叶, 用CTAB法提取基因组DNA[20]。
PCR总体积为20 μ L, 含2× PCR Mix (全式金公司) 10 μ L、引物各0.5 μ L、DNA模板1 μ L、双蒸水8 μ L。PCR程序为94 ° C预变性5 min; 94℃变性30 s, 60℃退火30 s, 72℃延伸50 s, 35个循环; 72℃延伸5 min。用1%琼脂糖凝胶在1× TAE缓冲液中电泳分离PCR产物。所使用DNA分子标量为Trans2k Plus DNA Marker (全式金公司)。
用SPSS 18.0软件对分子标记基因型分离比进行卡方检验。
取BC3F3种子无胚端的半粒, 用锤子砸碎置1.5 mL离心管中。参照Wan等[21]描述的方法提取HMW-GS, 在10%分离胶上进行SDS-PAGE电泳。
Glu-1特异分子标记扩增谱带(图1)与预期(表1)一致, 其中标记Xrj5在含Glu-A1b的Glenlea中扩增出882 bp谱带, 在小偃81和中国春中的扩增谱带分别为594 bp和719 bp; 标记BxMAR在含Glu-B1al的Glenlea中扩增出563 bp谱带, 在小偃81和中国春中分别扩增出800 bp和520 bp谱带; 标记Xrj2在含Glu-D1d的Glenlea中的扩增谱带为428 bp, 中国春和小偃81携带Glu-D1a, 扩增产物为1085 bp。
利用中国春缺体-四体系, 将Xrj5、BxMAR和Xrj2分别定位于小麦染色体1A、1B和1D上(图1)。3个分子标记均为共显性标记, 且在Glenlea和小偃81间存在明显多态性(图1)说明这3个分子标记可以用于Glenlea和小偃81 Glu-1位点的基因型分析。
由于DLGlu1同时缺失了Glu-A1、Glu-B1和Glu-D1位点, 因此3个分子标记检测DLGlu1均为阴性(图2), 在DLGlu1 × Glenlea杂交后代中, 该3个标记扩增出的条带为来自Glenlea的Glu-A1b、Glu-B1al和Glu-D1d等位位点。
用标记Xrj2和Xrj5检测BC3F2-6单株后代, 均检测出与Glenlea相同的带型, 说明Glu-A1b和Glu-D1d在BC3F2已经纯合, 而标记BxMAR检测结果存在分离, 因此在BC3F2-6单株后代中共发现Glu-A1b+Glu-D1d和Glu-A1b+Glu-B1al+Glu-D1d两种基因型(表2)。对BC3F2-16单株后代的检测结果表明, 共有4种基因型, 分别是Glu-D1d、Glu-A1b+Glu-D1d、Glu-B1al+Glu-D1d和Glu-A1b+Glu-B1al+Glu-D1d (表2)。
对BC3F2-56单株后代分两批检测, 共60粒种子, 3个分子标记均检测到分离, 说明Glu-A1、Glu-B1和Glu-D1位点都是杂合状态。检测第1批30粒种子发现5种基因型, 分别是Glu-A1b、Glu-A1b+Glu-D1d、Glu-A1b+Glu- B1al、Glu-B1al+Glu-D1d和Glu-A1b+Glu-B1al+Glu-D1d, 但是没有发现Glu-B1al基因型; 从第2批30粒种子中检测到2个Glu-B1al基因型个体, 但没有Glu-A1b、Glu- B1al、Glu-D1d全部为阴性的材料(表2)。
综上认为, DLGlu1× Glenlea BC3F3群体中存在以小偃81为背景、携带GlenleaGlu-1位点的7种渗入系(图2)。对BC3F2-6和BC3F2-16单株后代分离比的卡方检验表明, Glenlea的Glu-1遗传行为遵从孟德尔基因独立分配和自由组合规律(表2); 由于BC3F2-56后代个体观测数较小, 未进行卡方检验。
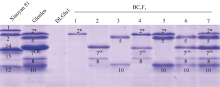
为了进一步验证Glenlea的Glu-1在所筛选材料的表达, 提取所检测材料籽粒谷蛋白, 采用SDS-PAGE方法检测发现所有材料均有相应Glu-1亚基的表达(图3)。同时发现, Glu-A1b+Glu-B1al基因型(BC3F3-4)的7OE表达量略低于2* 。
| 表2 以分子标记检测3个BC3F2材料后代基因型分布 Table 2 Genotype distribution in the progenies of three BC3F2 lines identified by molecular markers |
HMW-GS的组成是影响小麦加工品质的重要因素之一[13]。HMW-GS功能研究的主要手段有构建重组自交系或近等基因系研究同一位点不同等位亚基的功能差异[22, 23], 创制单位点缺失突变体研究单个位点的缺失效应[11], 及采用基因沉默或过表达手段研究单个HMW-GS亚基的功能[24, 25]。这些研究为解析HMW-GS功能提供了重要资料, 但是由于每个Glu-1位点均存在多种等位变异, 需要创制多套材料才能一一对其开展研究, 同时受环境及其他遗传位点(如LMW-GS或醇溶蛋白)影响, 来自不同材料的研究有时会得到截然相反的结论[26, 27]。有研究表明, 不同Glu-1位点编码的HMW-GS之间存在一定的上位效应[28], 通过构建近等基因系或转基因等实验材料难以消除这些影响。目前, 还没有一个较好的用于研究每个Glu-1位点及每个Glu-1位点不同等位变异功能的系统。本实验室采用离子束诱变方法获得Glu-A1、Glu-B1、Glu-D1单缺失突变体, 并证明了不同位点缺失在功能效应上存在显著差异[11]。本研究在此基础上, 采用杂交方法创制了不携带任何Glu-1位点的材料DLGlu1。理论上可以通过杂交或者转基因方法将任何Glu-1位点或HMW-GS亚基编码基因导入到DLGlu1背景进行相关基因的功能研究。
为进一步检验DLGlu1作为材料载体用于HMW-GS功能研究的潜力和创制不同HMW-GS渗入系的效率, 2014年我们以超强筋小麦Glenlea为父本, DLGlu1为轮回亲本开展了回交转育工作, 对每个世代的杂交材料均进行分子标记辅助选择, 大大减少了工作量。所有杂交种均以幼胚培养获得下一代材料, 两年时间即得到BC3F3材料。从这些材料中检测到携带Glenlea的Glu-A1b、Glu-B1al、Glu-D1d、Glu-A1b+Glu-B1al、Glu-A1b+Glu- D1d、Glu-B1al+Glu-D1d、Glu-A1b+Glu-B1al+Glu-D1d共7种基因型, 即获得了一整套以小偃81为遗传背景、携带Glenlea的Glu-1不同亚基组合的渗入系(图2和图3)。利用这些材料可以对2* 、7OE+8和5+10功能展开研究, 也可对HMW-GS全部或者部分缺失在培育低筋小麦的应用前景上展开研究。
通过计算Glenlea× DLGlu1的BC3F3材料分离比(表2), 发现Glu-1的遗传行为遵从孟德尔基因独立分配和自由组合规律。SDS-PAGE分析发现Glenlea的Glu-1编码的x-型和y-型亚基作为一个孟德尔单位共遗传(图3)。有意思的是, 发现携带Glenlea的Glu-A1b+Glu-B1al材料中2* 亚基表达量略高于7OE (图3), 推测这是由于该材料在Glu-A1位点纯合, Glu-B1位点杂合造成的剂量效应。李保云等[29]曾报道, 采用SDS-PAGE方法发现在正交和反交杂种F1中HMW-GS表达双亲所有的全部亚基, 但表达量不同, 母本谱带的表达量多, 父本谱带的表达量少。这种现象与小麦的双受精和三倍体胚乳形成时来源于母本和父本的遗传物质比例不同(母本占2/3, 父本占1/3)所造成的基因剂量效应有关[29]。本研究中携带Glu-1位点的材料作父本, 如果Glu-A1b纯合, 而Glu-B1al杂合, 则7OE的理论表达量为2* 的1/3, 这可能是SDS-PAGE凝胶中7OE的谱带略浅于2* 的原因之一。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|