第一作者联系方式: E-mail:baina0919@sina.cn
穗行数是影响玉米产量的重要因素之一, 其遗传机制解析和关键基因精细定位对开展分子育种具有重要的意义。本研究以穗行数仅有4行的“四路糯系选”和多穗行自交系“农531” (18~22行)为亲本, 构建了高代回交群体和次级定位群体(四路糯选系为供体亲本, 农531为轮回亲本)。通过对不同类型试验群体的多环境表型鉴定和基因型鉴定, 利用完备区间作图法(ICIM)进行穗行数主效QTL定位分析, 将穗行数主效位点 qKRN5.04定位到第5染色体136.3~140.0 Mb的区间之内; 遗传效应分析发现, 该位点在不同环境条件下最大可解释的表型变异为21.76%, 效应值为0.80~1.76行。通过次级分离群体重组事件分析可将其进一步定位到~300 kb区间内。本研究结果不仅为分子标记辅助选择提供了实用的InDel标记, 而且为玉米穗行数主效位点 qKRN5.04的图位克隆和候选基因挖掘奠定了重要的基础。
Kernel row number (KRN) is one of the important factors of maize yield. The genetic basis dissection and fine mapping of crucial gene would be greatly beneficial to maize molecular breeding. In present study, series of advanced backcross population and secondary segregation population were developed from a backcross of the derived line of Silunuo (four kernel rows, as the donor parent) and Nong 531 (18-22 kernel rows, as the recurrent parent). The QTL mapping for KRN was conducted by the inclusive complete interval mapping (ICIM) method in multiple environments. And a major KRN QTL, qKRN5.04, was mapped to the interval of 136.3-140.0 Mb on chromosome 5, with the largest phenotypic variation of 21.76% and the effect value of 0.80-1.76 row. Furthermore, according to recombinant analysis of secondary population, qKRN5.04 was fine mapped to the region of about 300 kb, which provided both practical InDel markers for marker-assisted selection and sufficient supports for the map-based cloning and candidate gene mining of the target locus.
玉米产量是复杂的数量性状, 受微效多基因控制, 其主要构成要素包括单位面积穗数、穗粒数和百粒重, 而穗粒数由穗行数和行粒数组成。玉米穗行数具有较高的遗传力, 且与产量密切相关, 是重要的产量相关性状之一[1]。因此, 深入研究玉米穗行数关键调控基因及遗传效应, 对促进玉米高产育种理论与应用的发展具有重要的意义[2]。
近年来, 在玉米穗行数遗传基础研究方面, 借助QTL作图, 已开展了大量工作。已报道的研究结果表明, 玉米穗行数遗传区域主要分布在bin2.03-2.04、bin4.05、bin4.08-4.09、bin5.04、bin7.02-7.03、bin8.05、bin9.03、bin10.03-10.04等染色体区段[3, 4, 5]。基因挖掘方面, 利用人工创制的突变体, 确定了一些重要的玉米穗行数调控基因。例如, 当barren inflorescence2 (bif2)、barren stalk1 (ba1)、sparse inflorescence1 (spi1)、Barren inflorescence1 (Bif1)、barren stalk1 (ba1)、Suppressor of sessile spikelets1 (Sos1)等基因突变后, 会引起小穗分枝减少, 从而导致穗行数减少[6, 7, 8]。而另一些基因的突变则会引起穗行数的增加, 这些基因包括FASCIATEDEAR2(FEA2)、td1、ramosa1 (ra1)、ramosa2 (ra2)、branched silkless1 (Bd1)、rgo1、Zfl1、Zfl2等[9, 10, 11, 12, 13, 14]。但人工突变体与自然群体中存在的变异类型差别较大, 难以直接应用于育种实践。在此条件下, 充分挖掘自然群体中的既有变异类型, 基于图位克隆方法挖掘穗行数相关基因, 对解析玉米穗行数遗传调控机制和开展分子标记辅助选择具有重要参考价值。
基于分子标记辅助选择, 利用高代回交群体构建导入系进行QTL分析, 已经成为研究数量性状遗传机制的有效方法。导入系群体遗传背景基本一致, 极大程度地降低了遗传背景对表型的干扰, 从而有效提高目标表型性状鉴定的准确性; 同时, 由于导入系含有的外源片段数较少, 任何表型变化都可认为由导入片段而引起[15], 因此, 可实现主效位点遗传效应的准确估计。胡立宗等[16]基于玉米BC2F2群体, 采用复合区间作图法定位到了3个穗行数QTL; 李卫华等[17]利用单片段代换系群体结合显著性检验的方法检测到3个穗行数QTL; 齐欢欢等[18]利用导入系群体对玉米产量及产量相关性状定位, 两个环境下一致性地检测到2个穗行数QTL; Li等[19]利用130个染色体片段代换系(CSSLs)共检测到11个穗行数QTL; Tian等[20]利用BC5F2:3把一个穗行数的主效QTL qKRN10定位到标记ssr1430和umc1077之间。截至目前, 虽然利用自然变异的分离群体定位到一些穗行数主效QTL, 但真正实现克隆的穗行数主效位点仍然很少, 仅Liu等[21]利用近等基因系, 将一个穗行数主效位点KRN4定位到了1.2 kb的区间内, 并成功验证水稻的同源基因Unbranched3 (UB3)是KRN4的关键调控基因。
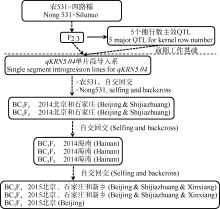
本研究基础材料为四路糯选系和农531。四路糯是起源于云南勐海、西双版纳等地的一个原始地方品种, 具有多分蘖、多果穗、少穗行、半有稃等一系列玉米原始性状特征; 农531是一个多穗行数玉米自交系, 最高可达22行。以上2份材料的穗行数性状差异极其显著。在本实验室前期的研究工作中, 利用穗行数只有4行的四路糯选系和多穗行亲本农531为亲本, 构建了F2:3家系分离群体, 借助多环境表型数据和基因型数据, 在基因组范围内共定位到5个穗行数主效QTL。其中, 位于第5染色体上的主效位点qKRN5.04单环境下最大可以解释13.55%的表型变异, 联合环境分析可以解释9.83%的表型变异[22], 且该位点位于bin5.04穗行数QTL富集区段之内。因此, 本研究以该位点为关键目标区段, 借助分子标记辅助选择, 通过构建一系列高代回交群体和大规模次级分离群体, 进行穗行数主效位点qKRN5.04的精细定位和遗传效应分析, 以期为该位点的图位克隆和候选基因挖掘提供参考和支持。
以四路糯选系为供体亲本, 农531为轮回亲本, 从BC1代开始, 借助分子标记辅助选择, 筛选仅包含qKRN5.04导入的单株(去掉含有其他穗行数主效位点的基因型), 并通过连续的回交和自交, 获得了主效位点定位的相关试验群体(图1)。
2014年夏, 在北京昌平(BJ, 116.20° N, 40.22° E)和河北石家庄(SJZ, 114.26° N, 38.03° E)同时种植BC3F2和BC4F2群体, 进行单株分离群体的表型和基因型鉴定, 并全部自交以获得BC3F3和BC4F3群体的种子。当年冬天, 在海南三亚(HN, 109.30° N, 18.15° E), 根据北京和河北石家庄QTL定位结果, 一方面, 挑选主效区段纯合型的BC3F2和BC4F2自交后代的种子, 进行基于BC3F2:3和BC4F2:3家系的表型评价, 同时种植目标区段杂合的BC3F3和BC4F3分离群体, 进行表型和基因型鉴定, 并全部自交以获得BC3F4和BC4F4分离群体; 另一方面, 种植目标区段杂合的BC5F2群体, 进行单株表型和基因型鉴定, 并全部自交, 用于主效位点的进一步验证和精细定位群体的构建; 2015年, 在北京昌平种植BC3F4、BC4F4和BC5F3群体, 以获得更多的交换单株, 用于QTL效应检测和重组事件分析, 完成主效位点qKRN5.04精细定位。单行种植试验群体, 1次重复, 行长3.0 m, 行距0.6 m, 每行定植12株。各试验点田间管理与当地大田管理相同, 成熟后收获, 以玉米穗轴中部穗行数为准调查表型。
基因型鉴定的分子标记, 一部分来自MaizeGDB (http://www.maizegdb.org/)公布的SSR标记, 该部分标记主要用于单片段主效QTL (qKRN5.04)导入片段的分子标记辅助选择。同时, 为了提高作图精度, 实现主效位点精细定位, 利用Illumina HiSeq 2000测序平台, 进行亲本四路糯和农531深度测序(20X), 利用Primer5.0设计开发针对qKRN5.04遗传区段的多态性分子标记, 以增加目标区段标记密度。
在幼苗长到6片叶取单株新鲜叶片, 采用CTAB法[23]提取植物基因组DNA。利用筛选到的多态性标记, 通过PCR扩增、8.0%非变性聚丙烯酰胺凝胶电泳、0.1% AgNO3染色、甲醛和NaOH显色, 鉴定所有群体基因型。
利用QTL IciMapping V3.3完备区间作图法(ICIM, inclusive composite interval mapping), 进行QTL检测, 共进行1000次的排列测验(permutation test)以确定QTL的LOD阈值, 同时计算QTL的遗传效应及其对表型的贡献率[24]。利用SAS9.2进行数据的方差分析(Proc ANOVA)、单标记分析(Proc GLM)及多重比较(Proc GLM/DUNCAN, α = 0.05)。利用t测验(Proc TTEST)检验不同交换类型与受体亲本间的表型差异。按Stuber等[25]标准判定基因作用方式, 当显性效应值与加性效应值比值的绝对值为0~0.2时认为是加性(additive, A), 0.2~0.8为部分显性(partial dominance, PD), 0.8~1.2为显性(dominance, D), 大于1.2为超显性(over dominance, OD)。利用一般线性模型(GLM)检测主效位点与环境间的互作效应, 模型为Y = G + E + G × E + error, 其中, Y代表家系穗行平均数, G代表家系基因型, E代表环境, error代表残差。
四路糯选系在不同环境下穗行数稳定为4行, 农531在14BJ、14SJZ、14HN、15BJ、15SJZ、15XX (河南新乡) 6个环境下穗行数均值分别为18.28、18.15、18.38、18.60、17.07和18.02, 变化范围均值在17~19行之间; 双亲之间存在极显著差异。不同环境下, 不同世代回交群体的穗行数均值为15.38~ 16.81行, 群体内部穗行数表型多样性高(表1)。
| 表1 不同回交群体及其亲本的穗行数 Table 1 Kernel row number for parents and backcross populations with qKRN5.04 |
依据穗行数主效位点qKRN5.04在染色体上的物理位置, 筛选多态性引物并开发标记。首先, 在MaizeGDB上查找SSR引物序列, 筛选多态性引物; 同时参考亲本四路糯和农531全基因组重测序结果, 设计InDel扩增引物, 共计230对, 其中筛选到24对引物扩增稳定、条带清晰, 用于高代回交群体的基因型鉴定(部分多态性引物见表2)。
| 表2 目标区段内开发的多态性InDel标记 Table 2 Polymorphic InDel markers within the target QTL |
利用qKRN5.04区段内的最显著SSR分子标记(基于F2:3群体定位结果), 进行各回交群体的基因型鉴定, 然后结合表型数据, 利用单标记分析(single marker analysis, SMA)方法, 对BC3F2、BC4F2、BC3F3、BC4F3、BC5F2等分离群体, 进行不同基因型材料间穗行数的显著性测验(表3和图2-A, B, C)。结果表明, 基因作用方式均呈现部分显性; 除2014年海南三亚的BC3F3群体外, 农531和四路糯基因纯合型的穗行数均达到显著性差异。为了减小偶然因素造成的影响, 还进行了基于家系表型和基因型的主效位点遗传效应分析。结果表明, 在2014年海南三亚, 农531纯合型BC3F2:3家系(N531/N531)和四路糯纯合家系(SLN/SLN), 穗行数均值分别为16.70、15.24, 遗传效应为1.46行, 且两种基因型间穗行数差异达到极显著水平(P = 5.40E-09) (图2-D)。
| 表3 玉米穗行数主效位点qKRN5.04的多群体验证和遗传效应分析 Table 3 Effect validation and genetic analysis of multiple populations for major locusqKRN5.04 on kernel row number |
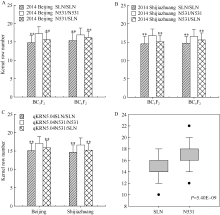 | 图2 玉米穗行数主效位点qKRN5.04在不同环境条件下的遗传效应比较分析 A, B分别代表2014年北京和河北石家庄环境下的主效位点qKRN5.04在BC3F2和BC4F2群体内的遗传效应比较; C代表2014年北京和河北石家庄2个环境下(群体合并计算) qKRN5.04的遗传效应比较; D代表2014年海南三亚环境下不同基因型BC3F2:3家系间的穗行数差异比较, * * 在代表水平P< 0.01差异极显著; N531、SLN分别是农531和四路糯选系的缩写。Fig. 2 Genetic effect comparisons for kernel row number related major QTL (qKRN5.04) under different environments A, B represent the genetic effects analysis ofqKRN5.04(BC3F2 and BC4F2 population) in 2014 Beijing and 2014 Shijiazhuang, respectively; C represents the genetic effects analysis ofqKRN5.04in 2014 Beijing and 2014 Shijiazhuang (total populations); D represents the KRN comparison of different genotypes of BC3F2:3 family in 2014 Hainan, * * represents significant difference at P< 0.01; N531 and SLN are the abbreviation of Nong 531 and Silunuo, respectively. |
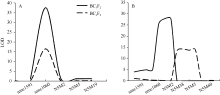
在2014北京, 用BC3F2和BC4F2群体可同时将qKRN5.04定位在136.3~138.5 Mb的染色体区段之内, LOD值分别达37.56和16.68, 可以分别解释21.76%和8.68%的表型变异(表4和图3-A)。在2014石家庄, 利用同样的BC3F2和BC4F2定位群体, 可以在紧密相邻的区域范围之内, 将该QTL定位于138.5~140.0 Mb之内, 可以解释的表型变异分别为15.29%和6.44% (表4和图3-B)。遗传效应分析表明, 减效等位变异同样来自供体亲本四路糯, 效应值为0.80~1.76行。2015年, 在北京还种植了BC3F4、BC4F4、BC5F3分离群体, 其中用BC3F4和BC5F3群体可同时将qKRN5.04定位至相同染色体的139.7~140.0 Mb区段之内。同时发现, 在不同环境下该主效位点的基因作用方式以加性和部分显性为主。总之, 本研究定位结果与前期基于F2:3分离群体结果高度一致, 表明该位点能够在多个高代回交群体稳定地表达, 为重要的玉米穗行数主效位点。
利用一般线性模型分析主效位点与环境的互作效应(71个BC3F2:3家系、30个BC3F3:4家系和56个BC4F3:4家系, 同时种植于北京、河北石家庄和河南新乡3个环境), 没有检测到显著的互作效应, 表明qKRN5.04能够在不同环境中稳定表达。
| 表4 不同回交群体玉米穗行数主效位点qKRN5.04的多环境定位分析 Table 4 QTL mapping for qKRN5.04using multiple backcross population in different environments |
在利用多个回交群体进行主效QTL验证定位的工作基础之上, 还进一步构建了包含398个单株的BC5F3群体, 结合标记加密, 可将该主效位点定位至3.8 Mb的染色体区段之内(umc1060-N5M5) (图4-C)。在此基础上, 通过目标区段内多态性标记的进一步加密, 进行了不同重组类型与轮回亲本间的表型差异分析, 以实现穗行数主效位点qKRN5.04的精细定位(图4)。在umc1060-N5M5区段内, 可进一步筛选出3个多态性标记, 可将BC5F3群体分为12种重组类型(图4-D)。以受体亲本农531为对照(P2), 进行了各重组类型与对照组的t检验分析, 结果表明, 标记区段N5M98-N5M136内含四路糯等位基因的重组类型(R1、R6-R8), 其穗行数显著高于受体亲本类型(P2), 标记N5M98-N5M136之间含农531等位基因的交换类型(R2-R5, R9-R12), 穗行数与受体亲本类型之间无显著差异(图4-D)。因此, 推测调控玉米穗行数的关键基因位点位于标记N5M98-N5M136之间约300 kb的区间之内。
本研究的目标区段qKRN5.04初定位于81~150 Mb约69 Mb的区间, 最大可以解释13.55%的表型变异[22]。该定位结果与之前很多研究得出的结果区段重叠。例如, Yan等[5]、谭巍巍等[26]利用F2:3家系在第5染色体上分别定位到穗行数QTL rn5(80.8~ 151.6 Mb)、QTL qKRE1-5-2 (80.4~142.3 Mb); 吕学高等[27]利用大小为400的F2群体, 也在bin5.04检测到1个穗行数主效QTL RN3, 位于标记bnlg1660- phil09188之间, 贡献率为15.1%。周强等[3]通过meta-QTL分析, 在第5染色体定位到4个穗行数一致性QTL区间, 其中包括本研究的qKRN5.04区段。以上证据表明, qKRN5.04所在区段对玉米穗行数调控具有重要作用, 可作为穗行数遗传基础研究和候选基因挖掘的重要热点区域。
基因型与环境的互作对数量性状遗传定位具有重要的影响[28], 甚至可导致定位结果的偏差。但整体上, 主效QTL位点更容易在不同环境中被稳定地检测到[29]。本研究中, 在2014年北京, 利用BC3F2和BC4F2群体, 可同时将qKRN5.04定位在136.3~ 138.5 Mb之间; 在石家庄, 利用相同群体, 可将该位点定位在138.5~140.0 Mb之间, 与北京试验点定位区间紧密相邻。另外, 无法检测到显著的主效位点与环境间的互作效应。进一步表明, qKRN5.04能够在不同环境中稳定表达, 为环境不敏感型玉米穗行数主效位点。
初定位的区间较大, 不利于候选基因的挖掘和图位克隆, 利用回交构建导入系等次级作图群体是数量性状主效位点挖掘基因的有效策略之一[30]。导入系的独特优势在于, 除特定的染色体片段来源于供体外, 其他遗传组成与受体完全相同, 消除了遗传背景的干扰, 固此可通过对导入系与表型性状的显著性分析把目标性状基因或QTL定位于较小的区段内, 实现精细定位[31]。Zhang等[32]利用导入系BC4F3将控制穗粒数的主效位点qKN缩小到480 kb的范围内。Teng等[33]用单片段导入系(SIL)将株高主效位点qPH3.1定位到12.6 kb区间内。Liu等[21]利用近等基因系将控制穗行数的主效位点KRN4缩小到1.2 kb的区间内。本研究在已有初定位基础上, 利用分子标记辅助选择构建了针对主效区段的多个高代回交群体和次级分离群体, 在准确验证原有定位结果的同时, 利用重组事件分析将玉米穗行数主效位点qKRN5.04定位到约300 kb的染色体区段之内, 为下一步的候选基因挖掘提供了重要的工作支持。
在本研究定位到的~300 kb区间之内, 以玉米B73基因组为参考, 发现该区段内共存在10个基因, 其中7个能够在玉米雌穗中得以表达, 但仅有2个基因有功能注释, 分别为GRMZM2G300841和GRMZM2G060253。序列分析发现, GRMZM2G 300841在拟南芥中的同源基因是At3g24315, 即编码磷脂酰肌醇转运蛋白(PITPs)的成员之一SEC20转运蛋白。研究发现AtSec20在拟南芥花器官中高强度地特异表达[34, 35]。GRMZM2G060253在拟南芥中存在同源基因At4g23800, 编码HMG转录因子。研究表明, HMG转录因子具有保守的HMG-box结构域, 该结构域与生物生殖发育关系密切, 且有证据表明该基因与玉米雌雄花序发育具有一定的关系[36]。另外一个基因GRMZM5G897364功能尚未验证, 但是表达谱分析发现, 该基因在发育的玉米雌穗中表达量最高[35]。总之, 依据目前定位结果尚不足以确定候选基因, 仍需依据交换事件分析进一步缩小目标区段, 在此基础上克隆基因, 并通过转基因及互补试验验证功能, 挖掘并深入分析玉米穗行数主效位点qKRN5.04的关键调控基因。
以“ 四路糯” 选系和“ 农531” 分别为供体亲本和轮回亲本, 针对玉米穗行数主效位点qKRN5.04, 构建多个高代回交群体, 将其定位到第5染色体的136.3~140.0 Mb区间之内。同时, 通过次级分离群体重组事件分析, 将其进一步缩小到约300 kb的染色体区间范围内, 从而为目标位点的图位克隆提供了重要的工作基础。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|