第一作者联系方式: E-mail: 781692734@qq.com, Tel: 17698308361
SRO是植物特有的类RCD-ONE蛋白家族, 在植物抵御各种生物和非生物胁迫过程中具有重要的作用。本研究利用生物信息学方法从陆地棉遗传标准系TM-1 ( Gossypium hirsutum L. acc. TM-1)基因组中鉴定到12个 SRO基因家族成员。多重序列比对及进化树分析表明, 12个陆地棉SRO均含有PARP和RST结构域, 聚为3类, 即A、B和C亚家族。转录组数据分析表明, TM-1全基因组中的 Gh_D12G1442在茎中优势表达, Gh_D05G2064在萼片和雄蕊中优势表达; Gh_A05G3788在雄蕊中优势表达, Gh_A12G1318在雄蕊和纤维中优势表达; Gh_A12G2480、 Gh_D12G2608、 Gh_A05G2257和 Gh_D05G2516在纤维中优势表达; Gh_A08G1390、 Gh_D08G1685、 Gh_A12G2663和 Gh_D12G2054具有较为广泛的组织表达。逆境胁迫处理试验表明, Gh_A08G1390明显受冷处理和盐处理的诱导, Gh_D12G2054、 Gh_D08G1685、 Gh_A12G2663基因在不同的胁迫处理下受到不同程度的诱导。
RCD-ONE proteins family (SRO) is a kind of plant-specific proteins, which plays an important role in plants in response to different abiotic stress. In this study, 12 SRO genes were preliminary identified in Gossypium hirsutum L. standard line TM-1 by bioinformatics analysis. Multiple sequence alignment and phylogenetic tree analysis showed that these SROs contain PARP and RST domains, and could be divided into A, B, and C subfamilies. Transcriptome data analysis showed that in the whole genome of TM-1, Gh_D12G1442 advantageously expressed in the stem, Gh_D05G2064 in the sepals and stamens, Gh_A05G3788advantage expression in stamens, Gh_A12G1318 in stamens and fiber, Gh_A12G2480, Gh_D12G2608, Gh_A05G2257, Gh_D05G2516 advantage expression in fiber; Gh_A08G1390, Gh_D08G1685, Gh_A12G2663, and Gh_D12G2054 in wider organs. In stress treatment showed that Gh_A08G1390was obviously induced by cold and salt treatments, Gh_D12G2054, Gh_D08G1685, Gh_A12G2663 gene were induced try different stresses to a different degrees.
植物适应外界高温、干旱等环境的变化是由自身特有的一些转录因子所调控, 一些转录因子在基因组水平上已经被分离, 例如NAC (NAM、ATAF和CUC), AP2 (APETALA2)/ERF (ethylene-responsive element binding factor), WRKY, Dof (DNA-binding with one finger)和ARF (auxin response factor)[1]。许多植物特有的转录因子家族成员参与植物特殊的发育过程, 参与并调控植物对外界环境的应激反应[2, 3, 4]。在这些植物特有的转录因子家族中, SRO (SIMILAR- TO-RCD-ONE)是最近被鉴定出的一类小蛋白家族, 在植物逆境反应中具有重要的作用。它包含植物所特有的PARP (Poly ADP-ribose polymerase)催化中心和C末端的RST [RCD-SRO-TAF4 (TATA-box- binding protein-associated factor 4)]结构域[5, 6]。
在拟南芥中, 共鉴定出SRO家族的6个同源基因, 分别为AtCEO1、AtSRO1、AtSRO2、AtSRO3、AtSRO4 和AtSRO5。AtCEO1能提高野生型酵母和Yap1突变酵母对氧化胁迫的抵抗能力[7]。其功能缺失能够引起拟南芥中超过500个基因的表达受到不同程度的调控, 其突变能够引起植物细胞的快速死亡, 所以又被称之为RCD1 (Radical-induce cell death 1), 其突变体表现出提前开花, 能够引起叶绿体ROS抗性、盐敏感性、改变NO、激素反应等多种发育及胁迫反应表型[8, 9, 10]。因此, AtCEO1在拟南芥生长发育和对环境胁迫应答机制中发挥着重要的作用。研究表明, AtSRO1与 AtRCD1之间存在功能上的部分冗余, AtSRO1也参与到非生物胁迫反应当中, 但相应的突变sro1-1对渗透、氧化等胁迫具有较野生型强的抵抗能力[11, 12, 13], AtSRO5的表达受到盐胁迫的诱导, 为氧化胁迫反应所需, AtSRO5之前被报道作为公共的胁迫反应因子发挥作用[14], AtSRO2、AtSRO3和AtSRO5也显示出在强光、盐处理及O3胁迫下转录子水平的变化, AtSRO4没有明确的类似功能[15]。
对于SRO的功能研究除了模式植物拟南芥外, 水稻、小麦、番茄等植物上也有报道[16]。水稻RCD1在一定的条件下有助于蛋白质DREB2A的稳定, 敲除RCD1基因或缺失RCD1结构域的一部分, DREB2A蛋白功能将缺失[17]。在水稻中, SRO水稻蛋白OsSRO1c, 与水稻干旱转录因子SNAC1 和DST直接作用[18], 在干旱和氧化应激反应中通过气孔作用扮演双重身份。AtSRO其他成员在研究中较少涉及。上述研究显示, SRO家族可能是一类转录调节因子参与了植物对多种胁迫的应答[19, 20, 21]。
棉花是重要的纤维作物, 迄今, 陆地棉中已克隆到了2个SRO基因, 分别为GhSRO04 (GenBank登录号为KR534896)和GhSRO08 (GenBank登录号为KR534895), 这2个基因受高盐、干旱和黄萎病菌处理后诱导表达, 表明SRO在棉花生长发育过程中应对病菌、盐和干旱胁迫起着重要的调节作用, 对棉花种质的抗逆性遗传改良具有潜在的利用价值[22]。棉花中对SRO家族的系统研究还未见报道, 为了探究棉花SRO在陆地棉中纤维发育及抵御逆境过程中扮演的角色, 本研究利用6个拟南芥SRO蛋白为查询序列, 鉴定编码棉花SRO蛋白基因, 并对其进行生物信息学和表达模式分析, 为进一步明确SRO蛋白基因在棉花抗逆生长发育中的功能奠定基础。
陆地棉(Gossypium hirsutumL.)遗传标准系TM-1由中国农业科学院棉花研究所中熟课题提供。种植在中国农业科学院棉花研究所东场试验站。在盛花期挂牌标记当日花, 采集当日胚珠(0 DPA)及3、6、9、12、15、18、21、24、27和30 DPA (days post-anthesis)纤维, 迅速置液氮中冷冻并保存在-80℃冰箱, 取TM-1种子, 剥去种皮后用0.1% HgCl2浸泡10 min, 灭菌水冲洗3~4次, 播种于1/2MS培养基上, 28℃暗培养4 d, 水培2周后取幼苗的根、茎、叶, 液氮处理后-80℃保存。选取生长一致15 d苗龄的棉苗, 将根部的蛭石经流水小心冲干净, 快速置吸水纸上吸干, 分别进行各种处理, 以正常生长的幼苗为对照。高盐处理是将根部浸在400 mmol L-1 NaCl溶液中; 干旱胁迫是将苗置20%PEG-600溶液中, 在相对湿度45%、连续光照条件下处理; 冷处理是将棉苗置于盛有水(已预冷到4℃)的大烧杯中, 于4℃培养箱, 持续光照; 热处理是将棉苗置盛有水(已预冷到38℃)的大烧杯中, 于38℃培养箱, 持续光照; PEG、NaCl处理在25± 2℃培养箱中进行[23]。分别于0、1、3、6和12 h采集各处理真叶叶片, 迅速置于液氮, -80℃保存, 用于总RNA提取。
大肠杆菌(Escherichia coli) Trans1-T1 Phage Resistant Chemically Competent Cell、DNase I、Reverse Transcriptase M-MLV (RNase H-)反转录酶等购自宝生物工程(大连)有限公司; SYBR Premix Ex Taq试剂盒购于TaKaRa公司, 其他试剂均为国产分析纯
以TAIR数据库(http://www.arabidopsis.org/ index.jsp)中6个拟南芥SRO蛋白序列为探针, 与四倍体陆地棉的基因组数据库(http://www.cottongen. org/)进行Blast比对, 鉴定获得陆地棉全基因组中SRO家族的所有成员及其CDS和氨基酸序列。运用EBL-EBI (http://www.ebi.ac.uk/interpro/search/sequence- search)和NCBI的Blast P分析氨基酸序列的蛋白质保守区; 以IBS 2.0软件绘制蛋白结构域图。
利用四倍体陆地棉的基因组数据库(http://www. cottongen.org/)获得染色体长度及棉花SRO家族成员在亚基因组上的位置分布; 用MapInspect软件绘制SRO基因家族的染色体物理分布图。
利用WOLF PSORT (http://psort.hgc.jp/)和TMHMM (http://www.cbs.dtu.dk/services/TMHMM/)软件进行信号肽、跨膜区及亚细胞定位分析。
利用Clustal W软件对拟南芥和陆地棉SRO蛋白序列进行多重序列比对, 参数选为默认值。利用MEGA 6.0软件邻接法(Neighbor-Join, NJ)构建系统进化树, BootStrap值设定为1000。
用EASYspin Plus植物RNA快速提取试剂盒提取棉花总RNA, 经DNase I处理后, 以ND1000紫外分光光度计测OD260/OD280值, 计算RNA的浓度与纯度。用1.2%琼脂糖凝胶电泳检测RNA的完整性, 保存于-80℃备用。按照大连宝生物工程有限公司Reverse Transcriptase M-MLV (RNase H-)的说明书合成cDNA第1链。
采用Tophat和Cufflinks软件包分析陆地棉(TM-1)转录组数据库, 检索SRO家族不同组织及不同胁迫处理的RNA-Seq数据库, 获得SRO基因表达量的FPKM (fragments per kilobase of exon per millon fragments mapped, FPKM)值。用层次聚类算法[24]分析SRO在陆地棉不同组织及不同胁迫处理的表达数据, 用HemI软件绘制SRO基因不同组织及不同胁迫处理下的表达模式图。用HemI 1.0软件分析数据, 获得SRO在不同组织及不同逆境处理下的数据信息, 明确SRO基因在不同组织及不同逆境中的表达情况。
为验证并进一步了解棉花SRO基因家族在棉花不同组织中的表达模式, 参照RNA-Seq数据库, 选取Gh_A05G2257和Gh_D05G2516基因作为探究SRO基因家族在棉花纤维不同时期的表达模式的研究对象, 选取Gh_A05G3788、Gh_D12G2054和Gh_D12G1442基因作为探究SRO基因家族在棉花不同组织中的表达模式的研究对象, 分别设计5对特异性实时荧光定量引物(表1), 以棉花根、茎、叶、花和不同发育时期的棉纤维cDNA为模板进行qPCR扩增。
为明确棉花SRO基因家族在棉花中对逆境胁迫的应答机制, 参照RNA-Seq数据库, 选取Gh_ A12G2663、Gh_D12G2054、Gh_A08G1390和Gh_ D08G1685基因为研究对象, 分别设计4对特异性实时荧光定量引物(表1), 以棉花根、茎、叶、花和不同发育时期的棉纤维cDNA为模板进行qPCR扩增。在罗氏Light Cycler 480实时荧光定量PCR仪上进行qPCR, 反应体系含 SYBR Premix ExTaq (2× ) (Tli RNaseH Plus) 5 μ L、10 μ mol L-1上下游引物各0.4 μ L, cDNA模板1 μ L, 补dd H2O至10 μ L。反应程序为 94℃1 min; 95℃ 15 s, 52~56℃ 20 s, 72℃ 30 s, 45个循环。3次重复, 每次试验设置3个重复, 按照2-∆ ∆ Ct法分析结果。以上实时荧光定量部分都以Action作为内参引物。
| 表1 本研究所用的引物 Table 1 Primers used in the study |
以6个拟南芥SRO蛋白(AtRCD1及AtSRO1-5)为探针, 从陆地棉基因组数据库(http://www. cottongen.org/)中鉴定出12个SRO家族基因, 其编码区长度为993~1890 bp, 编码蛋白序列长度为330~629个氨基酸(表2)。
| 表2 陆地棉SRO基因家族序列信息 Table 2 Sequence information of GhSRO family genes |
在陆地棉A和D亚基因组分别分布有6个SRO家族基因, 分别位于A05、DO5、A08、D08、A12和D12上。在A亚组和D亚组有GhSRO基因分布的对应亚组间, 都存在成对的平行进化同源基因, 在A12和D12这1对亚基因组中有3对平行进化同源基因, 在A05和D05有2对平行进化同源基因, 在A08和D08亚基因组中只有1对平行进化同源基因(图1)。
利用EBL-EBI在线分析, 结合NCBI中Blast P比对, 发现12个棉花SRO蛋白成员均具有PARP (Poly ADP-ribose polymerase)催化中心和C-末端的RST [RCD-SRO-TAF4 (TATA-box-binding protein- associated factor 4)]结构域, 有的成员还含有WWE结构域(图2)。根据SRO蛋白序列中所包含的保守功能区域, 将SRO分为3种亚家族, A类SRO亚家族包含PARP和RST结构域, 分别位于第259~477氨基酸之间和第532~587氨基酸之间; B类SRO亚家族包含WWE、PARP和RST 3个结构域, 分别位于第38~173氨基酸之间、第178~400氨基酸之间和第330~517氨基酸之间; C类SRO亚家族包含PARP和RST结构域, 分别位于第25~222氨基酸之间和第240~332氨基酸。PARP和RST结构域是植物特有的, 由于棉花SRO蛋白质氨基酸序列的长度不同, 棉花SRO蛋白中PARP和RST结构域出现的位置和长度也不同, 推测其功能也不相同。
陆地棉SRO蛋白与拟南芥SRO蛋白的多重序列比较表明, 其同源区域主要集中在PARP-RST结构域(图3)。
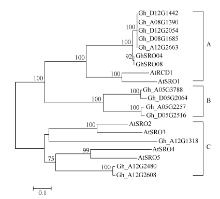
系统进化树表明, 12个陆地棉SRO蛋白中Gh_ D08G1685、Gh_D12G2054、Gh_D12G1442、Gh_ A08G1390、Gh_A12G2663聚为一类, 属于A类SRO亚家族; Gh_A05G3788、Gh_A05G2257、Gh_ D05G2064、Gh_D05G2516聚为一类, 属于B类类SRO亚家族、Gh_A12G2480、Gh_D12G2608、Gh_ A12G1318聚为一类, 属于C类SRO亚家族(图4)。
12个陆地棉SRO家族蛋白均没有跨膜区和信号肽存在; 其细胞功能定位并不相同, Gh_ D08G1685、Gh_D12G2054、Gh_D12G1442、Gh_ A08G1390、Gh_A12G2663和Gh_D05G2516定位于细胞核, 其他成员除了定位于细胞核外, 还定位于细胞质、微体(过氧化氢酶体)和叶绿体(表3)。因此, 推测SRO蛋白具有不同的功能。
| 表3 陆地棉SRO基因家族蛋白信号肽、跨膜区及亚细胞定位预测 Table 3 Prediction of signal peptide, transmembrane structure, and subcellular localization for SRO proteins |
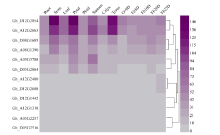
图5表明, 12个GhSRO基因在不同组织中均有表达。根据其表达模式, 可分为5种类型: (1)组成型表达的包Gh_A08G1390、Gh_D08G1685、Gh_ A12G2663和Gh_D12G2054; (2)茎中优势表达的是Gh_D12G1442; (3)萼片和雄蕊中优势表达的是Gh_ D05G2064; (4)在雄蕊中优势表达的是Gh_ A05G3788; (5)在雄蕊和纤维中优势表达的是Gh_ A12G1318。其中Gh_A12G2480、Gh_D12G2608、Gh_A05G2257、Gh_D05G2516在纤维中优势表达。
为了验证并进一步了解棉花SRO基因家族在棉花不同组织中的表达模式, 参照陆地棉(TM-1)不同组织及不同发育阶段纤维的RNA-Seq数据(图5), Gh_A05G3788、Gh_D12G2054、Gh_A05G2257、Gh_D05G2516和Gh_D12G1442基因实时荧光定量分析表明, 这5个基因在棉花不同组织及纤维各个发育时期均有表达, 其中, Gh_D12G2054的相对表达量在根、茎、叶、花中均高于在纤维中, 且在雄蕊中优势表达, 在纤维中各个时期均有表达, 但0DPA表达量高于其他纤维时期, 说明Gh_ D12G2054可能参与纤维起始阶段的发育。Gh_ A05G3788在雄蕊中优势表达, 与RNA-Seq数据分析结果一致(图6)。Gh_D12G1442在纤维中优势表达, Gh_A05G2257和Gh_D05G2516在纤维中特异表达, 这3个基因具有进一步研究的价值, 可将分子生物学和遗传学方法运用到解析棉花纤维生长发育调控机制研究中, 并可将其选为目标基因转化棉花改良棉花纤维的品质。
12个陆地棉SRO对逆境胁迫均存在应答响应; 随着处理时间的延长, Gh_D08G1685、Gh_ D12G2054、Gh_A12G2663和Gh_A08G1390基因的表达趋势都不相同, 表达量发生了明显的变化(图7)。qPCR的结果表明, Gh_D08G1685、Gh_ D12G2054、Gh_A12G2663和Gh_A08G1390对于4种胁迫都存在明显的响应, 变化趋势不尽相同(图8)。
如图8所示, Gh_A08G1390在正常生长状况下, 0 h表达量很低, 其表达模式在0~1 h之间没有明显变化, 在1 h后, 表达量急剧增加, 在3 h达最高值, 从3~6 h表达量又迅速下降, 在6 h时RNA转录水平和1 h时基本一致, 12 h时RNA转录水恢复到0 h。而在不同逆境处理后, Gh_A08G1390表达模式发生了明显的变化, 特别是热处理和PEG处理后, 在处理期间表达量基本没有变化, 而在NaCl和冷处理后, 表达模式和CK基本一致, 不同的是, NaCl处理后1 h时达到高峰, 且此时RNA转录水平远高于CK 3 h时的最高值, NaCl处理后1~3 h表达量迅速下降, 12 h RNA转录水恢复到0 h。Gh_A08G1390在冷处理后的表达模式和NaCl处理一致, 但在1 h的转录水平远低于NaCl处理后的最高值。陆地棉Gh_A08G1390在不同逆境环境下的应答反应说明该基因很可能在参与植物响应逆境胁迫过程, 并起重要作用。
Gh_D12G2054在正常生长条件下呈现先升高后降低且在3 h达到最大值的表达模式, NaCl处理和热处理后的表达模式与CK一致, 不同的是NaCl处理后1 h达到最高值, 热处理后6 h达到最高值。而在PEG处理后其表达量在1 h达到最高值, 1~6 h之间先降低后升高, 在3 h达到最小值, 6~12 h表达量缓慢下降。在冷处理后, 表达量先降低后升高, 然后再3 h后又开始下降, 在1 h后表达模式与CK一致。Gh_D12G2054基因在不同的胁迫处理下表现出的表达模式, 可能参与不同的胁迫调控途径, 与RNA-Seq数据库中基因响应胁迫应答相一致。
Gh_D08G1685在四种胁迫下与CK相比, 其表达模式也存在一定的差异, 在PEG, 热处理及冷处理情况下, 表达模式和CK相同, 呈现先升高后降低的趋势, 不同的是该基因在不同处理下转录水平达到最高值的时间不同, CK在3 h达到最高值, PEG处理后在6 h达到最高值。热处理后在6 h达到最高值。在冷处理后1 h就达到了最高值。而在NaCl处理情况下, 表达模式为先升高后降低, 再升高再降低, 表达量1 h达到最高值。
Gh_A12G2663在4种胁迫下与CK相比, 其表达模式基本一致, 即先升高后降低, 但RNA的转录水平达到最高值的时间不同, CK在6 h, PEG、NaCl和热处理在3 h达到最高值, 而冷处理和CK一样, 在6 h时达到最高, 且在1~6 h之间表达量有一个缓慢的上升过程。
由于全球气候变化和土壤盐渍化, 使得非生物胁迫成为限制棉花生长和产量的主要环境因子[23], 挖掘一些重要的抗逆基因显得十分重要, 而陆地棉全基因组数据库完成为我们提供了有利的工具。SRO是植物中特有的一类小蛋白家族, 在植物逆境反应中具有重要的作用。拟南芥中共有6个SRO家族成员, 以拟南芥6个SRO家族成员蛋白序列为探针, 共鉴定出12个陆地棉SRO家族成员。这12个SRO蛋白成员和马骏骏等[22]从抗枯萎病耐黄萎病的棉花品种鲁棉32中克隆获得的2个SRO基因GhSRO04和GhSRO08不同, 这可能是因为不同棉种之间序列存在多态性。蛋白质进化关系表明, 12个陆地棉SRO蛋白中Gh_D08G1685、Gh_D12G2054、Gh_D12G1442、Gh_A08G1390和Gh_A12G2663聚为一类, 属于A类SRO亚家族; Gh_A05G3788、Gh_A05G2257、Gh_D05G2064和Gh_D05G2516聚为一类, 属于B类SRO亚家族, Gh_A12G2480、Gh_D12G2608和Gh_A12G1318聚为一类, 属于C类SRO亚家族。B类SRO亚家族具有WWE、PARP和RST 3个结构域, WWE结构域与酵母 Yap1中的WW基序具有一定的同源性[13, 24], 目前该区域被认为是介导蛋白与蛋白之间相互作用的特异功能区域[25, 26, 27]。这些研究进一步明确了陆地棉SRO家族在植物SRO家族中的类别。
四倍体陆地棉基因组属于AADD型, 研究发现A亚组和D亚组的基因存在很高的共线性[28], 本研究鉴定出的陆地棉12个SRO基因在A亚组和D亚组中的分布就很好地体现了这一特性, 通过对陆地棉12个SRO家族成员的物理定位分析表明, 12个SRO基因家族在陆地棉A和D亚基因组对称分布, 分别位于A05、D05、A08、D08和A12、D12, 且都存在成对的平行进化同源基因。蛋白质跨膜结构域及信号肽分析表明陆地棉SRO蛋白属于定点蛋白, 在一些固定位置如细胞核和叶绿体、微体上与其他蛋白相互作用, 参与棉花的发育及各种胁迫反应。
转录组数据分析表明, 部分家族成员存在组织特异性表达。定量RT-PCR结果也验证了部分基因的特异表达模式。特别对Gh_A05G3788、Gh_ D12G2054、Gh_A05G2257、Gh_D05G2516和Gh_ D12G144在棉花不同组织中的表达模式分析表明, Gh_A05G3788在雄蕊中优势表达, 与RNA-Seq数据分析结果一致。Gh_D12G1442在纤维中优势表达, Gh_A05G2257和Gh_D05G2516在纤维中特异表达, 这3个基因具有进一步研究的价值, 可将分子生物学和遗传学方法运用到解析棉花纤维生长发育调控机制研究中, 并可将其选为目标基因转化棉花改良棉花纤维的品质。
马骏骏等[22]实时荧光定量 PCR (qRT-PCR)分析基因表达显示, GhSRO04和GhSRO08基因均受黄萎病菌侵染、干旱和高盐诱导表达, 表明GhSRO04和GhSRO08在棉花应对盐、旱和病菌胁迫中可能起着重要的调节作用[17]。参照不同逆境处理RNA-Seq表达数据库, 挖掘4个基因Gh_D08G1685、Gh_ D12G2054、Gh_A12G2663、Gh_A08G1390, 实时荧光定量分析表明, 这4个基因在一定程度上参与棉花的胁迫反应, 特别是基因Gh_A08G1390, 对各种胁迫特别是干旱胁迫, 热胁迫反应比较敏感, 在处理的12 h之间基本不表达, 可能是植物自身调节以对抗外界环境的改变, 更说明Gh_A08G1390受干旱胁迫的诱导。而在冷处理和盐处理的情况下, 在1 h表达量达到最大值, 而后急剧下降, 在6 h RNA反转录水平和未处理前一样, 说明Gh_A08G1390受冷处理和盐处理的诱导。Gh_A08G1390属于A类SRO亚家族, 而在拟南芥中, RCD1和SRO1同属于A类SRO亚家族, 拟南芥RCD1的功能缺失突变体导致对盐、葡萄糖、非原生质体活性氧的敏感性增加[10], RCD1和SRO1的双突变体, 在不同生长发育阶段的生物胁迫应答均受到严重影响[7, 9], 这些影响都与调节氧化还原平衡的改变有关[29]。小麦的SRO1对于提高非生物胁迫抗性和调节体内的氧化还原平衡具有重要的作用[30], 可以显著提高植株对盐害的耐受性。所以推测Gh_A08G1390是棉花胁迫反应过程中的一个重要基因, 可能承担着RCD1和SRO1相似的作用, 有必要对Gh_A08G1390进行深入的研究, 探索其在棉花生物胁迫过程中的作用。
本试验全面分析了SRO基因家族在陆地棉基因组的存在情况, 并找到了棉花发育过程中不同组织特异和优势表达的基因和棉花生物胁迫过程中可能的关键基因。有必要克隆并深入研究这些基因。本文为研究这些基因的分子生物学功能提供了基础理论依据, 同时也为棉花及其他植物中SRO家族的鉴定和功能研究奠定了数据基础。
从陆地棉中鉴定到12个SRO家族基因, 均含有PARP和RST结构域, 聚为3类, A类含 Gh_ D08G1685、Gh_D12G2054、Gh_D12G1442、Gh_ A08G1390和Gh_A12G2663; B 类含 Gh_ A05G3788、Gh_A05G2257、Gh_D05G2064和 Gh_ D05G2516; C类含 Gh_A12G2480、Gh_D12G2608和Gh_A12G1318。它们编码的蛋白在330~629氨基酸之间, TM-1全基因组中的Gh_D12G1442在茎中优势表达, Gh_D05G2064在萼片和雄蕊中优势表达; Gh_A05G3788在雄蕊中优势表达, Gh_A12G1318在雄蕊和纤维中优势表达; Gh_A12G2480、Gh_D12G2608、Gh_A05G2257和Gh_D05G2516在纤维中优势表达; Gh_A08G1390、Gh_D08G1685、Gh_A12G2663和Gh_D12G2054具有较为广泛的组织表达。Gh_A08G1390明显受冷处理和盐处理的诱导, Gh_D12G2054、Gh_D08G1685、Gh_A12G2663基因在不同的胁迫处理下受到不同程度的诱导。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|