第一作者联系方式:E-mail:shencc0805@163.com, Tel:010-82105855
利用多亲本高代互交系(multi-parent advanced generation inter-cross, MAGIC)群体(DC1、DC2和8way)及其复合群体DC12 (DC1+DC2)和RMPRIL (DC1+DC2+8way)进行关联分析定位水稻抽穗期和株高QTL。2015年和2016年分别在江西和深圳收集3个MAGIC群体抽穗期数据, 2016年在两地收集株高数据, 结合Rice 55K SNP芯片进行基因分型, 利用关联分析方法检测到3个影响抽穗期的主效QTL ( qHD3、qHD6和 qHD8), 分别位于第3、第6和第8染色体, 且分别与已知抽穗期基因 DTH3、 Hd3a和 Ghd8在同一区域。检测到5个影响株高的QTL ( qPH1.1、 qPH1.2、 qPH1.3、 qPH4和 qPH6), 其中 qPH1.1和 qPH1.2位于已知基因 Psd1和 sd1附近, 其余3个QTL为影响株高的新位点, 但仅在1个群体和单个环境下被检测到, QTL表达受遗传背景和环境影响大。不同MAGIC群体定位抽穗期和株高的效果不同, 在8亲本MAGIC群体8way及复合群体DC12和RMPRIL分别检测到5、5和6个抽穗期和株高QTL, 明显多于4亲本群体DC1的2个和DC2的4个, 而且作图的精度更高, 表现在定位到的QTL显著水平高和与已知基因距离更近, 尤其是复合群体的联合分析(如DC12和RMPRIL)的作图优势更为明显。
Association analysis was performed to map QTLs for heading date (HD) and plant height (PH) using three multi-parent advanced generation inter-cross (MAGIC) populations including two four-parent- and one eight-parent-derived populations (DC1, DC2, and 8way), and their composite populations (DC12=DC1+DC2, RMPRIL=DC12+8way) derived from eight elite indica lines. Using the phenotypic data of HD collected in the two sites (Jiangxi and Shenzhen) across two years (
抽穗期和株高是水稻重要的适应性性状, 在产量和品质方面起着关键作用。抽穗期的长短决定品种地区适应性和季节适应性[1, 2], 株高直接影响着水稻的抗倒伏能力, 进而影响水稻产量[3, 4]。株高和抽穗期都是由主效基因和微效多基因共同控制[5, 6], 近年来国内外学者已经定位并克隆了许多水稻抽穗期和株高相关的功能基因[7, 8, 9, 10, 11, 12]。随着当前水稻育种目标朝着超高产、直播、机械收获等多样化发展, 需要进一步从种质资源中定位和挖掘更多的抽穗期和株高基因, 通过分子设计聚合不同抽穗期和株高基因, 以期满足不同育种目标对抽穗期和株高的多元化需求。
以往大多数QTL定位采用双亲本衍生群体的遗传连锁分析方法, 相对较低的分辨率和有限的有利等位基因发掘是其存在的主要缺陷[13]。近年来, 关联分析是一种基于自然群体的连锁不平衡定位QTL的方法[14], 与连锁作图相比, 它可以提高定位精度, 节省构建作图群体的时间和成本, 同时可以检测多个复等位基因[14, 15, 16]。但是, 自然群体并不是严格的随机交配, 复杂的育种史和大多野生群体内有限的基因流造成了群体结构分层, 这就使得不相关位点之间产生假阳性关联[17]。
为克服双亲本作图群体和自然群体定位QTL的缺陷, 利用多个优良亲本构建的MAGIC (multi- parent advanced generation inter-cross)群体定位QTL正在兴起[18, 19, 20, 21, 22, 23]。MAGIC群体可以弥补双亲作图和关联分析的不足, 具有以下几方面的优势:(1)由于多个亲本相互杂交, 比双亲作图群体有更丰富的遗传多样性, 比关联分析的种质资源具有更平衡的等位基因频率; (2)几乎不受群体结构的影响, 减少假阳性的概率[18, 24]; (3)通过不同亲本数目构建的群体及其合并群体作图, 可以比较遗传背景对QTL检测的影响, 分析QTL检测的遗传背景效应; (4)累积的重组会提高作图精度, 同时会出现许多优良的重组类型, 增加优良个体选择的机率, 因此可以实现基因定位与育种的有机结合[21, 25, 26]。本文利用从国际水稻研究所引进的8亲本MAGIC群体, 连续两年在两个不同环境考查水稻抽穗期和株高进行, 结合高通量SNP基因型进行全基因组关联分析定位影响株高和抽穗期QTL, 研究结果将为抽穗期和株高有利基因挖掘奠定基础, 同时为分子育种提供材料和有利信息。
本试验所用的MAGIC群体由菲律宾国际水稻研究所构建[27, 28]。利用来自不同国家的8个优良品种(系)为亲本构建3个相互关联的水稻MAGIC群体, 分别是2个4亲本群体和1个8亲本群体。8个亲本的编号、来源和特点见表1。将8个亲本分成ABCD和EFGH两组, 两组4亲本间两两成对杂交(A× B、C× D和E× F、G× H), 产生4对双亲本杂交种, 将这4对双亲本杂交种F1再成对杂交产生两种4亲本杂交种(A/B//C/D和E/F//G/H)。通过单粒传法将4亲本杂交种自交6代, 构建2个4亲本MAGIC群体, 包括221个稳定株系的A/B//C/D群体(记作DC1)和219个稳定株系的E/F//G/H群体(记作DC2)。分别从4亲本杂交种A/B//C/D和E/F//G/H中选25个F1单株, 通过多次互交产生8亲本杂交种(A/B//C/D/// E/F//G/H)。再经单粒传法加代, 构建包含446个稳定株系的8亲本MAGIC群体(记作8way)。本研究在QTL定位时采用联合定位(Joint mapping)的方法, 将DC1和DC2群体合并称为DC12群体, 将DC1、DC2和8way群体合并称为RMPRIL(rice multi- parent recombinant inbred lines)群体。
| 表1 用于构建MAGIC群体的8个亲本的相关信息 Table 1 Description of eight parental lines used for developing MAGIC population |
将群体和亲本材料于2015年和2016年分别种植在江西省萍乡市农业科学研究所试验田(113.85° N, 27.6° E)和中国农业科学院深圳农业基因组研究所试验田(114.07° N, 22.6° E), 分别命名为15JX、15SZ、16JX和16SZ。2015年两地的播种时间分别是5月28日和6月26日, 2016年两地的播种时间分别是5月30日和6月14日。播种后25 d左右单本移栽, 株行距17 cm × 20 cm, 田间按两重复随机区组设计, 每株系每重复种植1行, 每行10株。田间管理按照常规大田生产的基本方法。
自播种至每个株系50%植株抽穗记为抽穗期, 每隔3 d记载一次。齐穗后选取每个株系中间长势一致的5株, 测量植株基部到穗顶部之间的长度(不包括芒), 即为株高。考察了两年两地的抽穗期和2016年两地的株高。
前人已对该MAGIC群体Rice 6K SNP芯片进行了基因分型[27, 28, 29]。为进一步提高标记密度, 于分蘖期从DC1、DC2和8way群体植株摘取幼嫩的叶片提取基因组DNA, 委托北京博奥晶典生物技术有限公司利用Affymetrix Axion Rice 55K Genotyping Array芯片进行基因重新分型。对获得的39 600个高分辨率的SNP基因型数据, 对每个群体进行如下步骤处理:(1)保留该群体亲本间有多态的标记; (2)保留该群体株系间有多态的标记; (3)杂合基因型作为缺失处理; (4)删除缺失率高于10%的标记; (5)删除稀有等位基因频率低于3%的标记。最终DC1、DC2、8way、DC12和RMPRIL群体分别获得22 160、22 020、28 505、28 540和28 531个高质量的SNP位点用于该群体关联分析。
Meng等[27, 28, 29]利用Rice 6K SNP芯片基因型对不同群体进行主成分和邻接进化树分析, 结果表明DC1、DC2和8way都没有明显的群体结构, DC12群体可以明显分为2个亚群, RMPRIL群体可以明显分为3个亚群, 分别与原来合并前的群体对应。8way、DC12和RMPRIL群体的连锁不平衡程度较小, 衰减距离较短, 衰减速度较快, 而4亲本群体DC1和DC2的连锁不平衡表现趋于一致。
由于MAGIC群体生育期和株高遗传变异较大, DC1、DC2和8way群体未收集到所有株系的表型数据。3个群体在两年两点4个环境下同时收集到抽穗期数据的株系数分别是187、200和380份, 2016年2点2个环境下同时收集到株高数据的株系数分别是201、210和384份。采用SAS program version 9.2统计分析上述株系数据, 采用一般线性模型(Proc GLM)分析3个群体基因型、环境及基因型和环境互作方差。
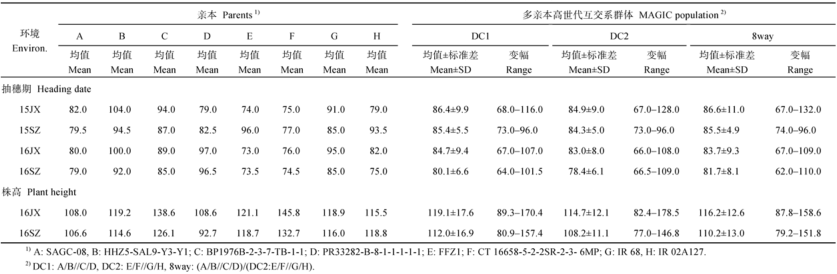
亲本和MAGIC群体在不同环境下抽穗期和株高遗传变异如表2所示。8个亲本在4个环境中, 平均抽穗期以DC2群体的亲本之一的CT16658-5-2- 2SR-2-3-6MP最短, 为75.6 d, DC1群体的亲本之一的HHZ5-SAL9-Y3-Y1最长, 为97.6 d。DC1、DC2和8way 3个群体的抽穗期均值相似, 但以8way群体的变异幅度最大。8个亲本在2个环境中, 植株最矮的是DC1群体的亲本之一PR33282-B-8-1-1-1-1-1, 为100.7 cm, 最高的是DC2群体的亲本之一CT16658-5-2-2SR-2-3-6MP, 为139.3 cm。DC2、8way群体的平均株高略低于DC1, 变幅16JX以DC2群体为最大, 16SZ则以8way群体最大。
根据方差分析的结果, 可以看出DC1、DC2和8way群体抽穗期和株高的基因型效应、环境效应、基因型和环境互作效应均达到极显著水平(表3)。3个群体中抽穗期基因型解释表型变异分别为75.43%、67.88%和63.54%, 环境解释的表型变异分别为9.73%、14.79%和9.94%, 基因型与环境互作的解释表型变异分别为14.35%、16.57%和22.80%。3个群体中株高基因型解释表型变异分别为90.22%、84.19%和86.88%, 环境解释的表型变异分别为5.15%、6.66%和4.27%, 基因型与环境互作的解释表型变异分别为3.39%、6.30%和6.19%, 表明环境对MAGIC群体的抽穗期影响比对株高更大。
| 表2 亲本和MAGIC群体在不同环境下抽穗期和株嵩的表型性状表现 Table 2 Performance of parents and MAGIC populations for heading date plant height in different environments |
| 表3 MAGIC群体不同环境下的抽穗期和株高的方差分析 Table 3 ANOVA analysis of heading date and plant height in the MAGIC populations across different environments |
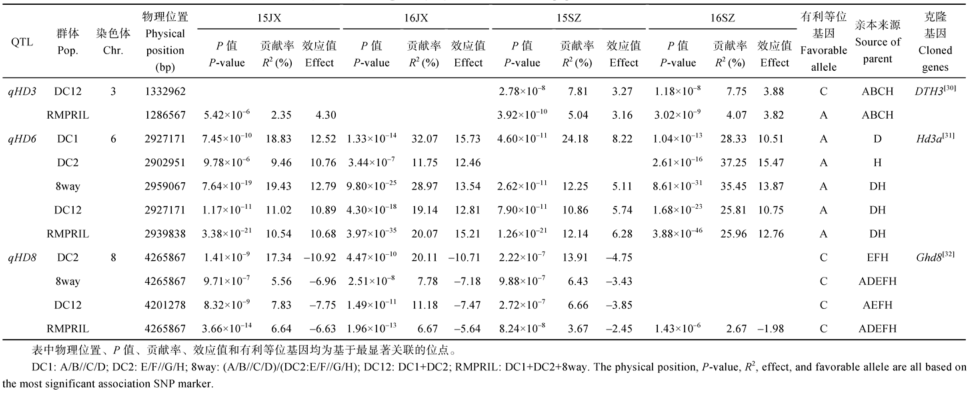
利用DC1、DC2、8way、DC12和RMPRIL群体在4个环境下共定位到3个与抽穗期相关的显著位点, 分别分布在第3、第6和第8染色体上(表4和图1)。qHD3在第3染色体的1.0~1.5 Mb区间有一个峰值, 在DC12深圳2个环境和RMPRIL群体在3个环境中鉴定出qHD3, 解释表型变异幅度从RMPRIL群体15JX的2.35%到DC12群体15SZ的7.81%, 亲本A、B、C和H携带的qHD3等位基因延迟抽穗。qHD6显著峰值在第6染色体的1.8~3.6 Mb区间, 除在DC2群体15SZ未检测到, 在其余群体不同环境下均检测到qHD6, 解释表型变异幅度从DC2群体15JX的9.46%到16SZ的37.25%, 是一个表型效应比较大的位点。亲本D和H携带的qHD6等位基因延迟群体的抽穗期。qHD8显著峰值位于第8染色体的3.9~5.1 Mb区间, 除在RMPRIL群体4个环境下均检测到, 仅在DC2、8way和DC12群体15JX、16JX和15SZ 3个环境检测到, 解释表型变异幅度从RMPRIL群体16SZ的2.67%到DC2群体16JX的20.11%, 亲本A、D、E、F和H携带的qHD3等位基因提早抽穗期。
| 表4 MAGIC群体在4个一坑下检测到影响抽穗期的QTL Table 4 QTLs for heding date (HD) detected in MAGIC population in two environments |
利用DC1、DC2、8way、DC12和RMPRIL群体在16JX和16SZ共定位到5个与株高相关的显著位点, 分别位于第1、第4和第12染色体(表5)。在8way、DC12和RMPRIL群体不同环境中都在第1染色体的35.5-37.0 Mb区域检测到qPH1.1, 解释表型变异幅度从RMPRIL群体16JX环境的3.41%到8way群体16JX环境的11.52%。除在DC2 16SZ环境下未检测到QTL外, 在5个群体不同环境中都在第1染色体的38.1~39.8 Mb区域检测到qPH1.2, 平均解释了22.71%的表型变异, 是所有检测到影响株高QTL解释表型变异最高的位点。携带qPH1.2位点的亲本C和F等位基因增加群体株高, 同时携带qPH1.2位点亲本C和F等位基因的3个8亲本群体株高增加最为显著。在RMPRIL群体16SZ环境下检测到qPH1.3, 位于第1染色体上, 解释表型变异为9.27%。在DC2和8way群体16JX环境分别检测到qPH4和qPH12, 分别解释表型变异12.23%和5.27%。
| 表5 MAGIC群体在2个环境下检测到影响株高的QTL Table 5 QTLs for plant height (PH) detected in MAGIC population in two environments |
4亲本群体DC1和DC2尽管它们的亲本数目相同、群体大小和标记密度相近, 但由于亲本间遗传差异大导致群体结构完全不同。对影响抽穗期的QTL, 在DC1群体只检测出1个, DC2群体检测出2个, 其中qHD6是在2个群体中都被检测到的, 但2个QTL的显著性水平和贡献率差异很大(表5)。同样是8亲本群体, 8way、DC12和RMPRIL群体检测到的QTL数目也不尽相同, 但定位到的QTL数目总体上要比4亲本群体多(表5)。qHD6在5个群体的遗传背景中被检测到, qHD8在4个群体的遗传背景中被检测到, 前者在5个群体4个环境的平均表型贡献率高达20.7%, 后者在4个群体4个环境的平均表型贡献率为9.0%, 均属于不依赖遗传背景或受遗传背景影响较小的稳定表达的2个主效QTL。
同样地, 对影响株高的QTL, 在DC1、DC2和8way群体及2个复合群体DC12和RMPRIL分别检测到1个、2个、3个、2个和3个。总体来看, 在亲本数多的群体要比亲本数少的群体定位到较多的QTL (表5)。qPH1.2在5个群体中稳定地被检测到, 平均解释株高的表型贡献率为22.7%, 是不依赖遗传背景稳定表达的1个主效QTL。qPH1.1在8way、DC12和RMPRIL群体中被同时检测到, 表明该QTL对遗传背景具有一定的依赖性, 而qPH1.3、qPH4和qPH12仅在1个群体背景中被检测到, 具有遗传背景特异性。
比较发现, 影响抽穗期的qHD3被定位在第3染色体的1.0~1.5 Mb区间, 该区间包含已克隆的抽穗期基因DTH3 (LOC_Os03g03070); qHD6被定位在第6染色体包含已克隆抽穗期基因Hd3a (LOC_ Os06g06320)所在的1.8~3.6 Mb区间; qHD8被定位在第8染色体的3.9~5.1 Mb区间, 该区间包含已克隆的抽穗期基因Ghd8 (LOC_Os08g07740)。5个影响株高的QTL中, 第1染色体的qPH1.1和qPH1.2所在区间附近存在已克隆的矮秆基因Psd1 (LOC_ Os01g60740)和半矮秆基因sd1 (LOC_Os01g66100)。在另外3个株高QTL (qPH1.3、qPH4和qPH12)区域没发现有其他株高基因或者QTL的报道, 可能属于新的位点。此外, 以上QTL中的qHD3、qHD6、qPH1.1和qPH1.2同样被该群体采用Rice SNP 6K低密度标记定位到[27, 28]。上述5个QTL (3个抽穗期和2个株高)在不同群体和环境下稳定被检测到, 表明这些位点影响抽穗期和株高不受或很少受环境和遗传背景的影响, 在分子标记辅助选择中具有应用价值。其与已知基因是否属同一基因还有待精细定位和克隆后方能知晓。
本研究中以定位到的抽穗期和株高QTL作总体统计, 利用8way群体定位到5个QTL, 多于DC1定位到的2个和DC2定位到的4个, 这与多亲本产生的后代可以检测到更多QTL的预期一致[35]。在8way群体检测到在DC1和DC2未检测到的qPH1.1和qPH1.2, 但却未检测到在DC2群体的qPH4。在3个群体共同定位到的抽穗期qHD6, 峰值距离与已知基因Hd3a分别为13、28和17 kb, 其中8way群体显著位点的P值最小, 为8.61× 10-31, 显著水平最高。在3个群体共同定位到的株高qPH1.2, 峰值与sd1的距离分别为16、162和0.4 kb, 其中在8way群体检测到的峰值对应P值最小(8.84× 10-20)。综上所述, 用8亲本群体比用2个4亲本群体定位QTL的效果要好, 不仅能检测到较多的QTL, 而且定位的精度较高。因此, 对于将定位到的QTL进一步克隆和直接用于育种, 应选择更多个亲本构建的MAGIC群体[27, 36]。
克服单个双亲本群体等位基因多样性低和作图分辨率低的一个策略是使用联合群体作图, 即多个MAGIC群体联合定位。与单个群体的分析相比, 多群体联合关联分析可以检测到更多QTL, 且QTL的位置更为精确, 同时还可以研究不同遗传背景等位基因替换效应。以在本研究定位到的抽穗期和株高QTL作总体统计, 用DC12群体(检测到5个QTL)和RMPRIL (6个QTL)显然比用DC1 (2个QTL)、DC2 (4个QTL)和8way (5个QTL)定位到的QTL要多。5个群体共同检测到的抽穗期qHD6比在复合群体DC12和在RMPRIL检测到该位点的显著水平高得多, 而且与已知基因Hd3a的距离也以复合群体检测的结果最接近, 其中RMPRIL群体检测的距离最小, 为1 kb左右。对于在5个群体共同检测到的株高qPH1.2, 在DC12群体的显著位点(P值3.23× 10-22)距离sd1仅0.4 kb左右, 在RMPRIL群体的显著位点(P值3.49× 10-32)则位于sd1基因物理位置内, 检测该QTL的显著水平和精度均显著好于4亲本群体。以上均表明联合分析具有更高的作图能力。但是在DC12和RMPRIL群体未检测到qPH4和qPH12, 可能与联合多个群体使得群体结构变得复杂有关。我们采用混合模型的关联分析, 同时将群体结构和亲缘关系作为协同变量, 这样的模型可能过度补偿群体结构和相关性, 并导致假阴性。这对于与亚群相关的性状尤其如此[37], 这也可以部分解释为什么一些QTL未被联合分析鉴定到。
抽穗期和株高是典型的数量遗传性状, 易受到环境条件的影响, 同一个群体在不同的环境条件下, 检测到的QTL数量和效应差别很大, 存在明显的QTL与环境互作效应[38]。本研究中我们利用多个遗传背景群体在不同环境下定位到稳定表达的3个主效QTL, 包括5个群体在4个环境下均检测到的抽穗期qHD6和4个群体在3个环境下检测到qHD8以及5个群体在2个环境下均检测到的株高QTLqPH1.2。qHD6和qHD8分别与2个抽穗期主基因Hd3a和Ghd8定位在一起, Hd3a基因在从长日照转变为短日照时诱导表达, 转录本的水平直接影响抽穗期, 而Ghd8基因在长日条件下抑制水稻特异开花调控基因Ehd1, 又同时抑制水稻成花素基因Hd3a和RFT1的表达, 在短日条件下促进水稻抽穗[39], 这2个抽穗期基因已广泛存在于推广品种中, 对不同地域水稻品种抽穗期调控起关键作用。株高QTLqPH1.2与“ 绿色革命” 基因sd1定位在一起, 该基因是最重要的半矮秆基因。上述3个主效QTL的表型效应大, 在不同遗传背景和环境下稳定被检测到, 属不依赖于遗传背景和环境稳定表达的主效QTL。相比之下, qHD3只在DC12和RMPRIL 2个群体中被检测到, qPH1.1在8way、DC12和RMPRIL群体中被检测到, 表明这2个QTL具有一定的遗传背景效应。而qPH1.3、qPH4和qPH12仅分别在1个群体背景中被检测到, 表明具有强烈的遗传背景特异性。对受遗传背景影响较大的QTL, QTL定位与育种群体最好统一, 才能取得较好的标记辅助选择的应用效果。
利用多样性丰富的8个亲本构建3个相互关联的水稻MAGIC群体及2个复合群体, 两年两点通过关联分析定位到3个抽穗期主效QTL和5个株高QTL。3个抽穗期主效QTL (qHD3、qHD6和qHD8)和其中的2个株高QTL (qPH1.1和qPH1.2)分别与已知基因定位在一起, 属不依赖遗传背景和环境独立表达的主效QTL。另3个株高新位点(qPH1.3、qPH4和qPH6), 其表达受遗传背景和环境影响较大。8亲本群体优于4亲本群体, 复合群体优于单一群体, 表现在检测到较多的QTL和较高的定位精度上。对定位到的QTL进一步克隆和将其应用到标记辅助育种, 应选择更多个亲本构建的MAGIC群体。
致谢:感谢国际水稻研究所叶国友博士和赵向前博士构建和友情提供MAGIC群体, 感谢本实验室曹国亮、朱双兵、刘晨、代明笠等同学帮助田间性状调查。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|