第一作者联系方式: 朱协飞, E-mail: fxx615@njau.edu.cn; 王鹏, E-mail: wp280018@163.com **同等贡献(Contributed equally to this work)
棉花产量分为籽棉产量和皮棉产量, 其中高皮棉产量总是育种的首要目标。皮棉产量由单株铃数、衣分、单铃重等因素组成。其中衣分在各因素中的遗传率最高, 同时也是产量育种中重要的选择指标。育种中利用分离群体对单株铃数、铃重等产量性状选择受环境影响较大。利用染色体片段导入系进行铃数、铃重等产量性状的定位, 定向改良产量性状, 是棉花分子设计育种的有效方法。本研究利用陆地棉TM-1为轮回亲本和海岛棉海7124为非轮回亲本构建了一套陆地棉背景的染色体片段导入系, 并在7个环境的田间试验下, 鉴定了它们的产量表现, 定位了28个与单株铃数、铃重、衣分和籽指相关的QTL。其中, 在Dt亚组染色体上鉴定出的产量性状QTL多于在At亚组染色体上鉴定出的。28个QTL中, 加性效应为正的16个, 加性效应为负的12个, 表明海岛棉不同的导入片段效应不同, 有的片段可以提高陆地棉产量, 有的则降低陆地棉产量。在6个环境下, 导入系IL008 (特征标记NAU2573和NAU3576)的衣分均显著高于轮回亲本TM-1, 因此IL008可以应用于棉花分子育种, 定向改良陆地棉的衣分。
Cotton yield is divided into seed cotton yield and lint yield. High lint yield is always the primary breeding goal in cotton. Lint yield consists of three components, including boll number per plant, lint percentage and boll weight. Of them, lint percentage has the highest heritability and is a most important target in breeding for increasing lint yield. Selection of yield components such as boll number and boll weight is easily affected by environmental factors in temporary segregating populations. It is testified that it is one of efficient methods to map the yield component QTLs and develop the elite lines in molecular breeding by using chromosome segment introgression lines (CSILs). In the present study, we developed a set of CSILs using G. hirsutumacc TM-1 as a recurrent parent and G. barbadense cv. Hai 7124 as a non-recurrent parent through the molecular markers assisted-selection. Here, we identified 28 QTLs for yield components under seven environments. Much more QTLs were enriched on Dt subgenome than on At subgenome. The chromosome segments introgressed from G. barbadense have different effects on yields in G. hirsutumbackground .There were 16 QTLs showing positive additive effects, implying these chromosome segments introgressed from G. barbadense could be used to improve yield components, while 12 QTLs showing negative additive effects, decreasing yield components. Lint percentage in IL008 line anchored with the SSR markers NAU2573 and NAU3576 was significantly higher than that of the recurrent parent TM-1 under six environments. Therefore, the CSIL IL008 could be used in molecular breeding to improve the lint yield in G. hirsutum.
单株铃数、铃重等产量性状是棉花的重要经济性状, 其表现型是基因与环境共同作用的结果, 并且产量性状与品质性状间存在显著的负相关[1]。传统的棉花产量改良, 都是直接以产量性状作为选择依据, 其选择易受环境等外在因素的影响, 选择的准确性较差。分子标记技术不受基因表达强弱、环境条件的影响, 易打破其不良连锁, 实现优良基因的定向转移和多基因聚合。产量性状QTL定位的研究大多是以F2或F3群体为基础, 遗传背景复杂, QTL定位结果除环境对其影响较大外, 还受到非目标QTL的干扰。虽然统计方法在不断改进, 但是这些问题始终无法克服。
陆地棉(Gossypium hirsutum L.)和海岛棉(G. barbadense L.)是棉花的2个四倍体栽培种, 陆地棉产量高, 纤维品质中等, 海岛棉产量低但纤维细强, 可纺100~200支纱, 是纺高支纱的原料。遗传基础狭窄已经成为目前限制陆地棉遗传改良的主要因素。在陆地棉和海岛棉间进行优良基因的高效转移渗透对二者的遗传改良具有极为重要的现实意义。如何将海岛棉中的这些优异基因渐渗到陆地棉栽培品种中, 拓宽陆地棉栽培品种的遗传基础, 创造更多优异种质, 是棉花品种改良必须考虑的问题。传统的海陆种间杂交后代性状疯狂分离, 要想得到稳定纯合的后代往往需要很长时间和巨大的工作量。染色体片段导入系(chromosome segment introgression lines, CSIL)是利用杂交、回交和分子标记辅助选择(marker-assisted selection, MAS)构建的覆盖作物整个基因组的一系列近等基因系, 其基因组内有一个或多个纯合的来自供体亲本的染色体片段, 而基因组的其余部分与轮回亲本相同。染色体片段导入系是进行基因组研究, 特别是QTL定位的理想材料。到目前为止培育了番茄、水稻、玉米、小麦、大麦、油菜等作物各自的染色体片段导入系, 并进行了QTL精细定位研究[2, 3, 4, 5, 6, 7]。本研究以TM-1为受体亲本, 海7124为供体亲本通过回交和自交, 并结合4次SSR标记辅助选择构建了一套棉花种间的染色体片段导入系。该导入系包括174个家系, 每个家系含有1~4个不等的导入片段, 共计298个片段, 导入片段的平均长度为16.7 cM, 覆盖棉花基因组的83.3% [8]。以此导入系为研究材料, 在7个环境下进行田间试验, 调查单株铃数、铃重、衣分和籽指4个产量性状, 筛选产量性状显著优于轮回亲本的导入系, 进一步利用SSR分子标记进行QTL定位, 发掘能提高陆地棉产量的优异基因资源, 为棉花高产分子育种打下坚实的物质基础。
TM-1是陆地棉遗传标准系, 来自美国德州农业部南方平原农业研究中心作物种质资源研究室[9]; 海7124是南京农业大学棉花研究所抗黄萎病遗传研究的一个单株选择后代, 纤维品质优良且高抗黄萎病[10, 11]。CSIL家系为南京农业大学棉花研究所选育[12]。
试验概况及调查性状见表1。每个环境的试验均采用2次重复, 随机区组排列。试验田管理同大田生产一致。在棉花摘顶后调查单株铃数; 从每行选取中间5个单株调查单铃重、衣分、籽指。
| 表1 CSIL家系试验概况及调查性状 Table 1 Field test of the CSILs |
用SPSS 17.0 (SPSS, Chicago, Illinois, America)进行CSIL家系的方差分析和群体平均数等群体特征数据分析。参考Wang等[13, 14]基于逐步回归的极大似然估计算法(A likelihood ratio test based on stepwise regression RSTEP-LRT)来检测多片段导入系中的QTL。其基本原理是: 采用逐步回归分析来选择对性状影响最大的标记(片段); 然后用似然比来估算每个标记的LOD值; RSTEP-LRT方法适用于非理想的 CSSL 群体(即每个家系含一个以上供体亲本插入片段), 对于理想的CSIL群体(每个家系只有一个来自供体亲本的插入片段), 该方法与标准t测验有相同的功效。利用QTL IciMapping 3.0[14](http://www. isbreeding.net/)检测CSIL群体产量相关性状的QTL, 以LOD≥ 3.0作为阈值来判断具有加性效应的QTL是否存在。
参照水稻上常用的方法命名QTL[15], 以小写的“ q” 起, 后接性状的英文字母缩写, 再接染色体或连锁群的编号(2~3个字母和符号), 如果同一染色体上有2个以上的QTL, 则加数字“ 1” 、“ 2” 、“ 3” 等加以区别。
所有性状在不同环境中的表型变异列于表2。亲本和CSIL群体的平均值在不同环境中表现存在差异。尽管CSIL群体在不同环境中的均值更接近于受体亲本TM-1的表型值, 但是有少量家系在不同的产量性状和环境中表现超亲分离。超亲分离的个别家系在不同环境中表现基本一致, 表明在这些家系中存在稳定表达的QTL。
| 表2 亲本及CSIL群体的产量性状 Table 2 Yield related traits of parents and CSIL population |
为了更深入分析CSIL群体中产量差异的来源, 对其基因型、环境效应和基因× 环境的互作效应进行了方差分析。从表3可以发现, 所有性状的基因型、环境效应和基因× 环境互作效应的F值都达到极显著水平, 表明它们受到以上3个方面的显著影响。
| 表3 产量性状的不同环境间的双向方差分析 Table 3 Two way ANOVA analysis of yield related traits |
利用QTL IciMapping 3.0软件在CSIL群体中检测到4个产量性状相关的QTL共28个, 其中铃重相关QTL最多, 为11个; 衣分QTL次之, 为7个; 铃数和籽指QTL都为5个。这28个QTL分别分布在染色体A1、A5、A6、A7、A10、A13、D1、D3、D4、D6、D7、D9、D12和D13上(表4和图1)。
| 表4 产量性状的QTL定位 Table 4 QTL mapping for yield related traits in CSIL population |
检测到28个与产量性状相关的QTL, 其在棉花2个亚组中的数量各不相同。所有性状QTS在At亚组的数量都少于在Dt亚组的数量, 适合性测验结果表明每个性状的QTL数量在2个亚组之间不存在显著差异, 但是QTL总量在2个亚组间差异显著。At亚组检测到的QTL总量显著少于Dt亚组检测到的QTL总量。通过对所有28个QTL的加性效应的方向统计发现, 加性效应为正的QTL有16个, 加性效应为负的QTL有12个(表4和图1)。本研究在14条染色体上检测到QTL, 但是每条染色体上检测到的QTL数目是不同的, 在Chr.D1、D3、D4、D9上检测到的QTL数目都是3个。
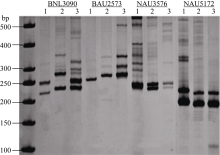
海岛棉产量较陆地棉低, 原因是海岛棉的单铃重低。而从表2可以看出, 海岛棉的单铃重比陆地棉低, 衣分在有些环境比陆地棉高, 有些环境比陆地棉低, 所以有些海岛棉染色体片段导入陆地棉以后, 能增加陆地棉的铃重或衣分。含有海岛棉染色体片段D1-3的导入系IL008 (图2)在6个环境下的衣分为39.22%~45.08%, TM-1在6个环境下的衣分为31.37%~33.58%, 含有海岛棉染色体片段D1-3的导入系IL008 的衣分比轮回亲本TM-1增加23.29%~41.72% (表6)。因此海岛棉染色体片段导入系IL008可以应用于棉花分子育种, 通过追踪IL008的特征标记NAU2573和NAU3576, 能有效地改良陆地棉衣分, 提高棉花的产量和育种效率。
| 表6 导入系IL008在不同年份、不同环境中衣分表现 Table 6 Lint percentage of IL008 in different years and environments (%) |
利用F2群体、回交群体或者重组自交系群体定位的缺点是它们的遗传背景复杂, 受到连锁累赘的影响较大, 从而降低了QTL定位的准确性。染色体片段导入系由于每个家系遗传背景一致, 受到的连锁累赘的影响小, 同时可提供大量的种子进行多年多点的重复试验, 减少环境对试验的影响, 所以导入系的应用必然能够促进对棉花产量性状遗传的研究。
本研究表明, 所有性状的基因型、环境效应和基因× 环境的互作效应的F值都达到极显著水平, 说明4个性状受到以上3个方面的显著影响, 但是环境对各性状的影响大于基因型和互作效应, 环境的差异是各性状表型差异的最重要的来源。由此可见, 在棉花农艺性状的QTL定位中必须考虑环境效应。先前的大量研究采用F2群体为研究材料[16, 17, 18], 无法进行有重复或区组效应的试验及多环境下的性状调查, 因而无法估算环境效应对棉花农艺性状QTL的影响。
本研究发现检测到的4个性状QTL都是分布在At亚组上的少于分布在Dt亚组上的。适合性测验结果表明每个性状的QTL数量在2个亚组之间不存在显著差异, 但是QTL总量在2个亚组间是存在显著差异的。At亚组检测到11个QTL, 极显著少于Dt亚组检测到的17个QTL (P=0.01), 表明QTL在Dt亚组富集。这与Jiang等[1]、Rong等[18]、Paterson等[19]、Wang等[20]的结果一致。
通过对所有28个QTL的加性效应的方向统计发现, 加性效应为正的QTL有16个, 加性效应为负的QTL有12个, 表明海岛棉不同的导入片段效应不同, 有的片段可以提高陆地棉产量, 有的则降低陆地棉产量。
染色体片段导入系可以对复杂产量性状进行定位并且定向改良棉花产量性状。我们调查了单株铃数、铃重、衣分和籽指4个性状的数据并定位了28个相关的QTL。其中含有海岛棉染色体片段D1-3的家系IL008的衣分在多个环境下显著高于轮回亲本TM-1。海岛棉染色体片段D1-3的NAU2573和NAU3576等SSR分子标记能大大提高棉花衣分的选择效率。分子标记辅助目标片段选择具有早期鉴定、快速鉴定及稳定性高等特点, 将加快高产棉花新品种培育与种子产业化进程。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|