第一作者联系方式: E-mail: liukaiyouxiang@163.com
小麦茎秆断裂强度与倒伏特性关系密切, 并对产量有很大影响。本研究旨在解析茎秆断裂强度的遗传机制, 开发与该性状紧密连锁/关联的分子标记。利用山农01-35×藁城9411重组自交系(RIL)群体(含173个F8:9株系)和由205个品种(系)构成的自然群体, 借助90 k小麦SNP基因芯片、DArT芯片及传统分子标记技术, 在2个环境中对两群体的茎秆断裂强度相关性状进行连锁分析和全基因组关联分析。利用已构建的高密度连锁图谱, 在4B染色体的TDURUM_CONTIG63670_287-IACX557和EX_C101685-RAC875_C27536等区段上, 检测到9个控制小麦茎秆断裂强度、株高、茎秆第2节间充实度、茎秆第2节壁厚相关性状的加性QTL, 可解释表型变异9.40%~36.30%。同时, 利用包含24 355个SNP位点的复合遗传图谱, 在自然群体中检测到37个与茎秆断裂强度相关性状( P < 0.0001)的标记, 分别位于1A、1B、2B、2D、3A、3B、4A、4B、5A、5B、5D、6B、7A、7B和7D染色体, 可解释表型变异7.76%~36.36%。在4B染色体上, 以连锁分析检测到控制茎秆断裂强度的RAC875_C27536与关联分析检测到的Tdurum_contig4974_ 355标记, 在复合遗传图谱上的距离为6.7 cM, 说明该区段存在控制小麦茎秆断裂强度的重要基因。
Stem strength has close relationship with lodging character, thereby, affects final yield in wheat. The objectives of this study were to unravel the genetic mechanism of stem-breaking-strength-related traits and find molecular markers closely linked or associated with these traits. We tried to map the stem-breaking-strength-related QTLs through linkage analysis using the RIL population consisting of 173 F8:9 lines derived from Shannong 01-35 × Gaocheng 9411) and association analysis using a nature population consisting of 2015 wheat varieties. Both populations were planted in two environments and genetically screened with the 90 k SNP array, DArT technology, and traditional molecular markers. By means of the existing high-density genetic map, nine additive QTLs were detected in different regions on chromosome 4B, such as DURUM_CONTIG63670_287-IACX557 and EX_C101685-RAC875_C27536, which controlled stem-breaking strength, plant height, filling degree of the second internode and culm wall thickness of the second internode and explained 9.40%-36.30% of the phenotypic variations. By means of a composite map (containing 24 355 SNPs) based on the Illumina Infinium assay, a total of 37 SNPs were found in the natural population to be associated with stem-breaking-strength-related traits ( P < 0.0001). These SNPs were distributed on chromosomes 1B, 2B, 2D, 3A, 3B, 4B, 5A, 5D, 6B, 7A, 7B, and 7D and explained 7.76%-36.36% of the phenotypic variations. The genetic distance between RAC875_C27536 detected through linkage analysis and Tdurum_contig4974_355 detected through genome-wide association analysis was 6.7 cM in the composite map, indicating the presence of important genes controlling stem strength in this region.
倒伏是小麦生产中普遍存在的问题, 不仅导致小麦产量降低、收割不便, 而且造成子粒瘪瘦、容重降低、磨粉品质和最终加工品质变差、小麦商品性下降等问题[1]。小麦倒伏一般发生在茎秆基部10%~30%处[2], 即茎秆基部第2节和第3节。小麦茎秆断裂强度是小麦自身抗倒性的综合指标, 与茎粗和秆壁厚、节间密度、茎秆结构特征以及茎秆化学成分关系密切。前人对小麦茎秆的形态学、解剖学和生理学的研究较多, 但缺乏遗传基础研究[3]。在基因水平解析小麦茎秆断裂强度的遗传机制, 对利用分子标记辅助选择和分子设计育种具有重要的意义。
国内外学者利用分子标记技术对小麦茎秆基部节间长度、茎粗、秆壁厚度及茎秆强度等进行了QTL定位, 共检测到30多个QTL[4, 5, 6, 7, 8, 9, 10, 11, 12]。主要分布在3B[4, 5, 6, 7]、4B[8, 9, 10]、5A[11, 12]染色体上。其中, Hai等[4]利用CA9613× H1488构建的113个DH家系, 定位到6个控制茎秆强度、茎壁厚、髓腔直径和茎粗的QTL, 其中在3A和3B上检测到2个控制茎秆强度的QTL, 其表型贡献率分别为10.6%和16.6%。Marza等[6]利用132个F12重组自交系, 检测到3个与倒伏程度相关的QTL, 位于1B、4AL和5A染色体上, 贡献率分别为16.7%、14.1%和23.0%。Verma等[8]利用Milan× Catbird创建的含有96个株系的DH 群体, 在4B和2D染色体上分别检测到与基部第1节间长和抗折断力相关的QTL, 贡献率分别为11%和13%。
随着基因芯片技术和测序技术的发展, 自动化程度更高的第3代单核苷酸多态性(SNP)标记迅速应用[13], 该标记技术应用在基因组中具有遗传稳定、数量多、分布广且易于检测等特点, 适合于数量庞大的检测分析[14], 在模式动植物和重要农作物中发挥着越来越重要的作用, 已成功应用于人类[15]、果蝇[16]、水稻[17]、玉米[18]等多种生物的遗传研究。而小麦是异源六倍体, 基因组庞大, 与水稻、玉米等作物相比, 小麦SNP标记的开发与利用较晚。迄今, 未见基于SNP标记对小麦茎秆断裂强度研究的报道。
近年来, 基于遗传连锁图谱的QTL作图及连锁不平衡(LD)的关联作图(genome-wide association study, GWAS)技术, 分别先后被应用于控制小麦重要数量性状的功能性等位变异检测及基因研究中[19, 20], 连锁不平衡关联作图无需构建专门的作图群体, 并能同时检测同一座位的多个等位基因, 但该方法易受群体结构的影响而产生标记与性状间的伪关联[21], 在作图的效率上很大程度受群体LD水平、标记密度等的影响; 而遗传连锁作图适用于全基因组QTL扫描, 可以对某一个QTL进行精细的遗传定位, 但该技术需要根据所研究的性状构建专门的遗传群体, 而且获得的分子标记受到群体亲本遗传背景影响, 实效性及应用范围常常受到一定限制。因此, 两种作图策略在QTL定位的精度和广度、提供的信息量、统计分析方法等方面具有明显的互补性。联合两种作图策略可以极大地提高对复杂数量性状的遗传解析力度[22, 23]。目前, 在玉米[23]、拟南芥[21]、毛白杨[24]中均有联合利用两种作图策略进行数量性状遗传解析的报道, 但在小麦中相关报道较少。
茎秆断裂强度是影响小麦倒伏的重要因素, 迄今缺乏遗传基础研究[3]。虽有茎秆强度基因定位的报道, 但因研究使用的群体遗传背景、群体大小以及分子标记技术不同等原因, 检测到的一致性基因位点不多; 另外, 前期研究使用的标记大多是传统的SSR、RFLP等标记, 遗传图谱包含多态性标记少、标记间距离大, 定位到的茎秆强度相关性状连锁不紧密, 难以在育种实践或深入研究中得以应用。本研究利用2个高密度SNP图谱, 利用关联分析和连锁分析两种策略检测小麦茎秆断裂强度, 以期找到与小麦茎秆断裂强度相关性状紧密连锁的SNP 标记, 为分子标记开发、精细定位及其在小麦育种中聚合有利基因/QTL提供参考。
以山农01-35× 藁城9411重组自交系(RIL)群体进行连锁分析, 母本山农01-35为大粒、成穗率低的高产品系, 其茎秆直径为0.61 cm; 父本藁城9411为小粒、分蘖成穗率高品种, 茎秆较细, 直径0.44 cm。F1代经一粒传法自交9代, 得到含173个家系(F8:9)。
全基因组关联分析采用205个品种(系)构成的自然群体, 包括我国冬麦区20世纪80年代以来的推广品种或骨干亲本132个、高代品系73份, 其中73份高代品系全部来自山东省。经小麦全基因组均匀分布的3297个SNP标记检测, 用Structure 2.0软件分析结果表明, 该自然群体分为4个亚群[25]。
将2个群体在2013— 2014年和2014— 2015年2个生长季种植于山东农业大学试验站(山东泰安), 随机区组设计, 2次重复, 每小区4行, 行长2 m, 行距0.21 m。播种期分别为2013年10月6日和2014年10月9日, 按当地常规方式进行田间管理, 生长期内没有发生严重病虫害和倒伏, 成熟时按小区机械收获各株系, 收获期分别为2014年6月8日和2015年6月9日。
参考魏凤珍等[26]和姚金保等[27]描述的表型调查方法, 略加改动。花后27 d调查株高; 花后18 d和27 d, 随机选每株系5个主茎, 在离地面20 cm处用YYD-1型茎秆强度测定仪(浙江托普仪器有限公司)测定田间茎秆断裂强度。花后18 d和27 d, 取每株系5个主茎, 烘干后截取茎秆第2和第3节中间位置靠下4 cm称重, 用YYD-1 型茎秆强度测定仪(浙江托普仪器有限公司)测定2个节间的茎秆断裂强度; 同时, 用游标卡尺测茎秆外径, 并取压断的4 cm茎秆上部, 用游标卡尺测其壁厚。
利用SPSS 19.0对上述表型数据进行统计分析。用如下公式, 分别计算各性状的广义遗传率。

式中, σ 2g为遗传方差, σ 2ge为遗传与环境互作方差, σ 2e为环境方差, n为环境数, r为重复数。
DArT标记的检测及序列信息由澳大利亚Triticarte Pty. Ltd. (http://www.triticarte.com.au/)完成。SNP分析由美国加利福尼亚大学戴维斯分校植物科学系生物技术检测中心利用Illumina SNP Genotyping 技术测试平台, 使用微珠芯片技术(BeadArray)完成, 用Genomestudio v1.0软件分析其多态性。通过GrainGene2.0 (http://wheat.pw.usda. gov/GG2/index.shtml)获得SSR引物序列等信息, 引物由上海桑尼生物科技有限公司合成。首先利用两亲本进行多态性筛选, 再用筛选出的多态性引物检测RIL群体所有株系。
1.4.1 RIL群体连锁图谱 利用陈建省等[28]构建的高密度遗传图谱, 该图谱覆盖小麦21条染色体, 包含6244个标记(SNP标记6001个, DArT标记216个, SSR标记27个), 总长度为4895.29 cM, 两标记间平均遗传距离为0.78 cM。
1.4.2 自然群体复合遗传图谱 利用陈广凤等[25]构建的高密度复合遗传图谱, 该图谱包含24 355个SNP标记位点, 覆盖小麦全基因组3674.16 cM, 单个染色体长度为118.91~241.38 cM, 标记间平均遗传距离为0.15 cM; 以B基因组中包含的SNP标记最多, 占标记位点总数的50.60%, 其次是A基因组(39.10%), D 基因组含标记最少, 仅占标记位点总数的10.31%。
1.5.1 连锁分析 用MAPMAKER/EXP3.0b软件和Mapchart 2.1构建分子标记连锁图。利用基于混合线性模型的QTLNetwork 2.0 软件对RIL群体茎秆断裂强度进行QTL分析。以单个环境中重复的平均值及2个环境平均值分别单独进行QTL定位。以P = 0.05为统计检测阈值, 即当标记的P值小于统计检测阈值时, 认为该标记处存在1个与性状有关的QTL。以茎秆强度相关性状英文简写和所对应的染色体序号及在染色体上的位置命名检测到的QTL, 如QSBSF4B.4-13表示位于4B染色体上第13个标记区间的QTL。
1.5.2 全基因组关联分析 应用TASSEL 3.0软件(http://www.maizegenetics.net/)中的MLM (mixed linear model)进行性状和标记之间的关联分析, 选择在小麦21条染色体长臂和短臂上均匀分布的3297个SNP标记, 用Structure 2.0软件进行群体结构分析, 采用admixed model, 设K值为2~15, 参数iterations为100 000, burn-in period为100 000。每个K值重复运行5次。以设定的K值所对应的最大似然值为目标, 选择合适的K值作为亚群数目。用TASSEL3.0软件计算Kinship值, 对群体结构和基因型过滤后, 运行MLM_Q+K模型。当标记的P ≤ 0.0001 (最小频率为0.05)时认为标记与性状存在关联。
母本山农01-35茎秆强度相关性状均明显大于父本藁城9411, 表明两亲本的抗倒伏特性差异很大; 由其衍生的RIL群体在各性状上均出现超亲分离, 且两年度表现一致(表1)。RIL群体的茎秆断裂强度、株高、花后27 d第2节壁厚、充实度的偏度及峰度绝对值都小于1, 均符合正态分布, 其他4个性状也基本符合正态分布。株高和茎秆断裂强度变异系数分别为9.52%~30.66%和9.32%~40.83%之间, 说明这2个性状具有较大的改良潜力。
| 表1 两年度RIL群体和自然群体小麦茎秆断裂强度的表型值 Table 1 Phenotypic values of stem-breaking-strength-related traits in RIL and natural populations in two growing seasons |
自然群体茎秆断裂强度变幅较大(0.05~0.75 N), 变异系数为7.13%~43.67% (表1), 其中花后27 d第2节变异系数最大为43.67%; 偏度和峰度绝对值小于1, 符合正态分布。株高变幅为57.25~130.00 cm, 平均值为79.96 cm, 变异系数均超过10%, 偏度和峰度的绝对值小于1, 符合正态分布; 花后27 d第2节间充实度、壁厚变异系数为7.65%~49.34%, 平均值分别为1.73 × 10-2 g cm-1和0.28 cm, 偏度和峰度绝对值小于1, 符合正态分布。
RIL群体在2个环境中, 花后18 d第2节与茎秆第3节断裂强度差异显著(P < 0.05); 花后27 d的第2节与第3节茎秆断裂强度差异也达到显著水平; 第2节茎秆断裂强度在花后18 d与花后27 d差异显著, 而第3节茎秆断裂强度在2个取材时期差异不显著(图1)。说明在RIL群体中, 基部节间茎秆强度存在显著差异, 取样时间对第2节茎秆强度有显著影响, 而第3节茎秆强度则不受取样时间影响。
自然群体在2个环境中, 花后27 d的第2节与第3节茎秆断裂强度(图1)差异均达到显著水平(P< 0.05); 而第2节茎秆断裂强度在花后18 d与花后27 d差异显著, 第3节茎秆断裂强度在2个取材时期差异也达到显著水平; 说明自然群体中取材时期和取材部位间的茎秆强度都存在显著差异。
方差分析表明, RIL群体的茎秆断裂强度、株高、花后27 d第2节间充实度和壁厚的基因型效应差异显著, 茎秆断裂强度相关性状的广义遗传力为56.92%~78.08%; 自然群体的茎秆断裂强度、株高、花后27 d第2节间充实度和壁厚的基因型差异也达显著水平, 广义遗传力为53.18%~78.64% (表2)。表明茎秆断裂强度遗传力较高, 适合进行遗传分析。
| 表2 小麦茎秆断裂强度的方差分析及广义遗传率(h2B)计算 Table 2 Analysis of variance and broad-sense heritability (h2B) for stem-breaking-strength-related traits of wheat in two environments |
相关性分析表明, 株高与田间茎秆断裂强度呈显著负相关(表3)。花后27 d, 节间茎秆断裂强度与第2节间充实度、壁厚均呈显著正相关, 说明该时期第2节间充实度和厚度增大, 有利于增加茎秆断裂强度、提高抗倒伏能力。
| 表3 茎秆强度相关性状之间的相关性 Table 3 Pairwise correlation coefficients among stem-breaking-strength-related traits |
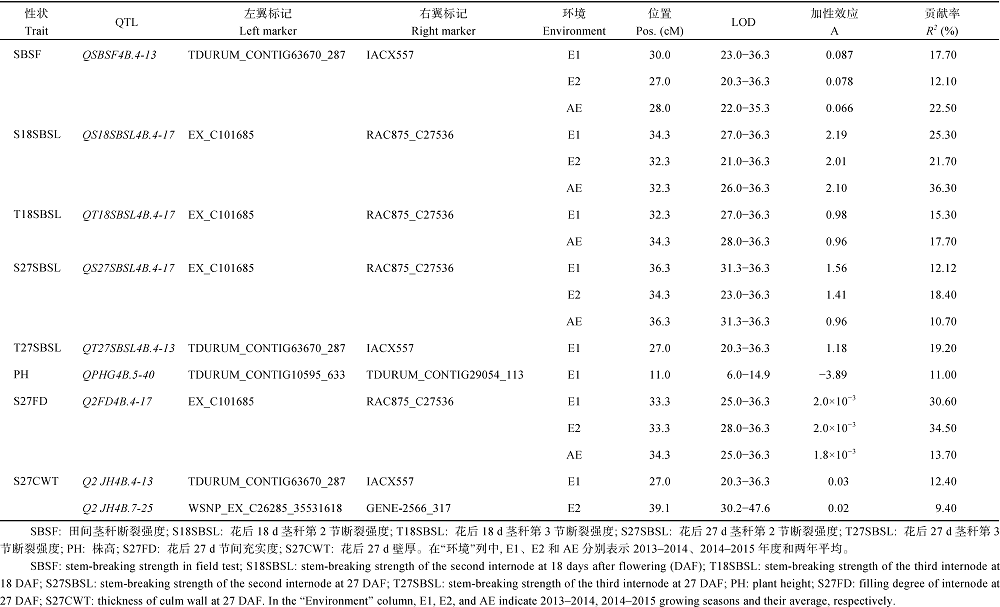
2.3.1 连锁分析 利用单个环境(E1和E2)中表型数据及环境平均值(AE)分别进行QTL分析, 共检测到控制茎秆断裂强度9个主效QTL, 均位于4B染色体上, 表型贡献率为9.40%~36.30% (图2和表4), 其中QSBSF4B.4-13的增效基因来自山农01-35, 可增加田间茎秆断裂强度0.083 N, 在E1、E2和AE中均被检测到, 是一个稳定位点。
 | 图2 茎秆断裂强度QTL在遗传图谱上的分布 各性状详细名称见表1。Fig. 2 Distributions of identified QTL for stem-breaking strength on genetic linkage maps The trait names correspond with those given in Table 1. |
| 表4 茎秆断裂强度相关性状的加性QTL Table 4 Additive QTLs detected for stem-breaking-strength-related traits in RIL population |
花后18 d, 检测到控制第2节和第3节茎秆断裂强度的QTL各一个, 增效基因位点全部来源于山农01-35, 可分别增加茎秆断裂强度2.10 N和0.98 N。QS18SBSL4B.4-17位点在E1、E2、AE中均被检测到。
花后27 d, 检测到第2节和第3节茎秆断裂强度、株高和第2节间充实度的QTL各一个, 以及2个控制秆壁厚的QTL。QS27SBSL4B.4-17和Q2FD4B.4-17在E1、E2和AE环境下均被检测到, 是稳定的QTL。除株高外, 增效基因位点全部来源于山农01-35, 检测到的QS27SBSL4B.4-17位点与QSBSF4B.4-13位点在连锁图谱的遗传距离为5.87 cM, 说明4B.4-17位点附近存在控制茎秆断裂强度相关性状的基因。
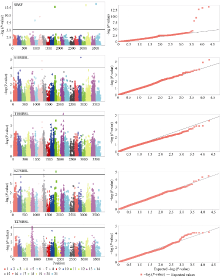
2.3.2 全基因组关联分析 共检测到37个与小麦茎秆断裂强度相关联的显著关联位点(P< 0.0001), 分布在1B、2B、2D、3A、3B、4B、5A、5D、6B、7A、7B和7D染色体上(表5)。单个关联位点表型变异贡献率为7.76%~36.36% (表5)。通过TASSEL V3.0软件分析, 获得2个环境下茎秆断裂强度全基因组关联分析的QQ图(图3), 关联群体的群体结构得到了较好控制。
| 表5 茎秆断裂强度的关联位点及其对表型变异的贡献率(R2) Table 5 Loci associated with stem-breaking-strength-related traits and contribution percentage of phenotypic variation (R2) |
检测到12个与小麦田间茎秆强度显著关联位点, 分布在2B、2D、3A、4B、7A 和7D染色体上(表5和图3)。其中, 在7A上检测到7个位点(图4), E2环境中检测到贡献率超过20%的5个显著关联标记(Kukri_c13329_800、BS00072153_51、Tdurum_contig 4974_355、wsnp_Ex_c14654_22713386和D_contig 06359_118)。这些SNP可能与控制田间茎秆断裂强度基因显著关联。值得一提的是, 在复合遗传图谱中, 4B染色体上的Tdurum_contig4974_355与利用RIL群体检测到的田间茎秆断裂强度标记IACX557, 二者的遗传距离为6.7 cM, 说明4B染色体上的这个区段由存在控制小麦茎秆断裂强度的重要基因。
 | 图4 茎秆断裂强度显著关联标记在复合遗传图谱上的分布Fig. 4 Distributions of identified QTL for stem-breaking strength on composite map |
花后18 d, 检测到3个与第2节茎秆断裂强度显著关联的位点, 分布在1B、3B、4B 染色体上, 贡献率为8.65%~9.21%。检测到6个与第3节茎秆断裂强度显著关联的位点, 均位于2B上3 cM的染色体区段内(图4), 贡献率为8.10%~8.41%。
花后27 d, 检测到3个与第2节茎秆断裂强度显著关联的位点, 分布在3A、6B、7B染色体上, 贡献率为9.09%~11.42%。检测到3个与第3节茎秆断裂强度显著关联的位点, 分布在5A、5D、7B染色体上, 贡献率为7.99%~9.29%。与株高显著关联的标记有3个, 分布在2B、4A、5B染色体上, 与第2节间充实度、壁厚显著关联的标记共7个, 分布在1A、1B和3A染色体上, 贡献率为7.88%~13.77%。
小麦茎秆断裂强度是衡量抗倒性的重要指标之一, 与小麦茎秆的茎粗、壁厚、茎秆结构、茎秆密度等形态学特征关系密切[4]。前人报道茎粗、壁厚和节间充实度与抗倒性呈显著正相关[26, 29]; 本研究结果阐明第2茎秆断裂强度与茎秆第2节间充实度、壁厚呈显著正相关, 而与株高呈显著负相关(表2), 结果进一步证实适当降低株高、增加茎粗、壁厚和节间充实度, 尤其是提高第2、第3节间茎秆断裂强度, 对增强小麦抗倒伏能力具有重要实践意义, 闵东红等[30]也有类似的报道。小麦株高是影响倒伏的重要农艺性状, 也是育种家田间选择淘汰后代材料的重要依据之一。20世纪60年代以来, 矮化育种[31]极大提高了小麦抗倒伏能力。但小麦植株过矮会导致叶层密集、病虫害加重、植株早衰和生物产量降低等负面效应。一般认为株高在75~85 cm的品种不易倒伏[1]。目前, 在小麦矮化育种的基础上, 通过分子标记辅助选择(marker-assisted selection, MAS)来提高小麦茎秆断裂强度, 增强小麦抗倒伏能力, 是实现小麦再高产的一条有效途径。本研究在4B染色体上检测到的QSBSF4B.4-13和QS18SBSL4B.4-17与茎秆断裂强度相关性状紧密连锁, 可为小麦育种中聚合和利用控制茎秆断裂强度相关性状的有利基因/QTL提供参考。
前人对小麦重要农艺性状研究方法多为单独利用遗传群体连锁作图的传统QTL定位或利用自然群体进行全基因组关联分析, 但这两种作图策略各有优缺点, 关联作图分析具有高作图分辨率, 但连锁不平衡作图效率低, 易造成假阳性关联结果[32]; 而遗传连锁作图却能利用稀有等位基因和较低的标记覆盖度将所有QTL 定位于染色体不同位置, 但作图分辨率低且遗传分离只涉及到父母本[33]。Qin等[34]通过联合连锁与关联分析策略得到一个控制玉米粒长的候选基因(ZmCKX10), Lu等[23]通过联合关联与连锁分析策略同时定位到与玉米散粉至吐丝的间隔期紧密连锁的分子标记(PZA03109.1)。本研究通过联合连锁与关联分析策略利用2个高密度SNP 图谱分别在4BS染色体上定位到控制小麦茎秆断裂强度的标记(RAC875_C27536和Tdurum_contig4974_355), 结果相互验证, 置信区间较小, 随着小麦基因组测序的逐渐完成, 研究结果能为小麦茎秆断裂强度的精细定位或功能基因克隆奠定基础。
连锁和关联分析都可以检测数量性状位点, 两种方法定位到的QTL在位置上大部分具有一致性[35, 36]。本研究利用目前最具发展潜力的SNP标记, 具有遗传稳定、密度高、易于检测等优点, 但其分型错误率和成本相对较高, 且需转化为CAPS标记或其他标记才能便于在标记辅助选择中利用, 操作繁琐。因此, 在新一轮高密度遗传图谱构建中, 可以根据QTL所在染色体上的相同或邻近标记遗传距离及物理距离, 并结合小麦测序信息和比较基因组学策略, 整合前人定位的小麦茎秆强度相关QTL, 进一步验证和发掘小麦茎秆强度稳定的主效基因位点。本研究通过连锁与关联分析都在4BS染色体上定位到控制茎秆断裂强度相关性状的标记, 但两种定位方法定位到的基因位点在染色体上仍有一定距离, 可能主要是由于两种策略的原理及利用的群体不同; 且小麦茎秆断裂强度相关性状多数为多基因控制的数量性状(单基因遗传率低, 环境影响大), 在环境检测中出现不稳定性等因素, 这有待进一步多环境确认。
目前, 国内外在小麦茎秆断裂强度QTL定位方面的报道较少。本研究在4B 染色体上通过连锁分析检测到茎秆断裂强度相关的9个QTL; 关联分析检测到2个与茎秆断裂强度显著关联标记。Verma等[8]和陈广凤等[25]也在4B染色体上检测到控制倒伏指数、株高、节间长度的QTL, 说明4B上存在控制茎秆强度相关性状的重要基因。本研究通过关联分析, 发现在3A染色体上存在田间和花后27 d第2节茎秆断裂强度的显著关联标记各1个, 在3B染色体上存在花后18 d第2节茎秆断裂强度的显著关联标记, Li等[4]在3A和3B上也检测到控制茎秆强度的2个QTL; Berry等[37]在3A上检测到2个控制茎秆强度的QTL。根据本研究结果和前人报道, 初步推断3A染色体上可能存在控制茎秆强度的重要基因。小麦4B染色体上存在矮秆基因Rht1, 本研究通过连锁分析在也4B染色体上检测到1个株高QTL (QPHG4B.5-40), 但该基因位点与Rht1不在同一连锁群上, 可能为一个控制株高的新位点。小麦4D染色体上除矮秆基因Rht2[8]外, 前人定位到多个控制株高的QTL[25, 38, 39], 而本研究未检测到4D染色体上与株高相关的QTL/显著关联标记。前人已在2D [4]和5AL [11, 12]上发现控制茎秆壁厚的QTL, 本研究在1A、3A和4B上单环境中检测到茎秆壁厚相关QTL/显著关联标记。
已报道与Rht1紧密连锁或直接相关的农艺性状有多个, Cadalen等[38]利用DH群体在4B染色体上检测到株高特异标记Xfba1-4B, 并与矮秆基因Rht1紧密连锁; Quarrie等[40]在Rht1附近检测到控制穗粒数的QTL簇; Huang等[41]检测到的Xgwm149标记和Liu等[42]检测到的Xgwm113标记也在Rht1附近; 而Shimin等[43]在Xwmc657-Xgwm495 (Rht1在本区间内)检测到排除Rht1影响的控制穗数、穗长和穗粒数的QTL。本研究定位到的茎杆强度分子标记RAC875_C27536和Tdurum_contig4974_355也都在Rht1附近, 且在复合遗传图谱上的遗传距离为6.7 cM, 但尚不能确定以上位点与Rht1的关系。
在4B染色体TDURUM_CONTIG63670_287- IACX557和EX_C101685-RAC875_C27536等区段上, 检测到9个控制小麦茎秆断裂强度、株高、茎秆第2节间充实度、茎秆第2节壁厚相关性状的加性QTL, 可解释表型变异9.40%~36.30%。主效、稳定基因位点4B.4-13、4B.4-17和显著关联标记Tdurum_contig4974_355, 可用于茎秆断裂强度相关性状的分子标记开发和基因精细定位。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|