第一作者联系方式: E-mail:2014101085@njau.edu.cn
为发掘水稻穗部性状有利等位变异, 构建了以籼稻保持系II-32B为遗传背景的A7444染色体片段置换系群体; 利用QTL IciMapping 4.1软件对该群体7个穗部性状进行了QTL定位。结果2年共检测到26个QTL。2年均检测到的13个QTL中, 控制一次枝梗数的4个QTL位于第1、第6、第8和第9染色体, 平均贡献率分别为15.16%、13.10%、29.74%和11.21%, 平均加性效应分别为-1.40、1.01、1.11和0.77。控制二次枝梗数的2个QTL位于第6和第8染色体, 平均贡献率分别为10.97%和21.39%, 平均加性效应分别为5.45和6.36。控制每穗总粒数的3个QTL位于第2、第6和第8染色体, 平均贡献率分别为8.65%、12.52%和31.22%, 平均加性效应分别为-18.61、22.23和31.87。控制每穗实粒数的1个QTL位于第8染色体, 平均贡献率为28.06%, 平均加性效应30.85。控制千粒重的2个QTL位于第2染色体, 平均贡献率分别为44.65%和17.51%, 平均加性效应分别为2.88和-2.51。控制粒宽的1个QTL位于第10染色体, 平均贡献率为21.96%, 平均加性效应为0.11。第2、第6和第8染色体分别存在同时控制二次枝梗数、每穗总粒数和每穗实粒数QTL的区段。 qSBN6和 qSBN8所在区间与 Hd1和 DTH8的相同, 但分别存在16处和1处碱基差异, 推测为 Hd1和 DTH8的不同等位基因。 qSBN2为新检测到的控制二次枝梗数位点。研究结果为实施分子标记聚合育种提供了有用信息。
For mining elite alleles controlling panicle traits in rice, a mapping population was constructed with chromosome segment substitution lines (CSSLs) by crossing and back crossing the indica variety II-32B (receptor parent) and the japonica variety A7444 (donor parent). A total of 26 QTLs associated with seven panicle traits were detected across two years using the QTL IciMapping 4.1 software, and 13 QTLs were identified repeatedly in both years. Among them, the four QTLs for primary branch number (PBN) were located on chromosomes 1, 6, 8, and 9 with average phenotypic variation explained (PVE) of 15.16%, 13.10%, 29.74%, and 11.21%, and average additive effect (AE) of -1.40, 1.01, 1.11, and 0.77, respectively; the two QTLs for secondary branch number (SBN) were located on chromosomes 6 and 8 with average PVE of 10.97% and 21.39%, and average AE of 5.45 and 6.36, respectively; the three QTLs for spikelet number per panicle (SPP) were located on chromosomes 2, 6, and 8 with average PVE of 8.65%, 12.52%, and 31.22%, and average AE of -18.61, 22.23, and 31.87, respectively; the unique locus for grains per panicle (GPP) was located on chromosome 8 with average PVE of 28.06% and average AE of 30.85; the two QTLs for 1000-grain weight (TGW) were located on chromosome 2 with average PVE of 44.65% and 17.51%, and average AE of 2.88 and -2.51, respectively; and only one QTL for grain width (GW) was located on chromosome 10 with average PVE of 21.96% and average AE of 0.11. Segments simultaneously controlling SBN, SPP and GPP were found on chromosomes 2, 6, and 8. According to bi-parent DNA sequence comparison, QTLs qSBN6and qSBN8were speculated to be different from Hd1and DTH8 because 16 and 1 base difference were found, respectively. QTL qSBN2seems to be a novel locus that controlling secondary branch number. These results are valuable for pyramiding breeding through marker-assisted selection.
水稻的产量最终表现在穗部, 穗部性状的改变直接影响水稻产量[1]。穗部相关性状包括穗长、一次枝梗数、二次枝梗数、粒数、千粒重等。穗部相关性状QTL定位对于聚合有利等位基因, 提高水稻产量具有重要意义。
根据Gramene网站(http://www.gramene.org/)统计, 迄今共定位了260个控制水稻穗长的QTL, 54个控制一次枝梗数的QTL, 33个控制二次枝梗数的QTL, 353个控制每穗总粒数的QTL, 131个控制每穗实粒数的QTL, 315个控制千粒重的QTL, 93个控制粒宽的QTL。关于穗部性状基因的克隆和生物学功能研究前人已有报道, 如第11染色体上穗长基因SP1编码PTR家族转运蛋白, 决定水稻穗形大小[2]。第6染色体上基因OsAPO1通过延迟枝梗分生组织的退化时间, 正向调节一次枝梗数和小穗数[3]。第10染色体上的TAW1通过延长枝梗分生组织发育时间和延迟小穗分生组织形成来调控水稻花序发育, 显性功能获得性突变体tawawa1-D, 表现为花序分生组织活性增大、小穗特化延迟, 导致枝梗形成时间延长、二次枝梗数比例增加[4]。第1染色体上影响每穗粒数基因Gnla编码一种降解细胞分裂素的酶, 该基因表达的减弱, 使得细胞分裂素在花序分生组织中累积, 增加每穗粒数[5]。第6染色体上千粒重基因TGW6, 通过直接控制胚乳的长度和间接参与从源到库的碳水化合物运输影响籽粒重量[6]。控制粒宽基因GW2编码环型E3泛素连接酶, 抑制颖壳的细胞分裂控制粒宽[7], GW5可能通过泛素蛋白酶体途径调节粒宽和粒重[8], GW8的高表达能促进细胞分裂和灌浆从而促进水稻粒宽增加[9]。
本课题组在筛选杂交水稻制种性状的研究中发现了剑叶角度150º 左右的特异品种A7444。A7444不仅剑叶角度大, 还具有二次枝梗数少、粒大的特点[10]。本研究以A7444为供体亲本, 以二次枝梗数多的籼稻保持系II-32B为受体亲本, 通过杂交、回交、自交和分子标记辅助选择, 构建了一套以籼稻保持系II-32B为遗传背景的A7444染色体片段置换系群体, 利用该群体检测了7个穗部性状的QTL, 以期为聚合穗部性状有利等位基因提供分子标记信息。
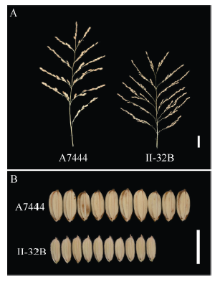
选用父本粳糯稻A7444 (图1-A左)和母本籼稻II-32B (图1-A右)及它们杂交衍生的66个株系组成的染色体片段置换系群体。A7444是在太湖水稻地方品种资源中发现的粳糯稻品种, 平均每穗二次枝梗数为25个, 千粒重为43.5 g。II-32B是生产上大面积应用的籼稻保持系, 平均每穗二次枝梗数为47个, 千粒重为25 g。双亲在穗部性状上差异极显著(图1和表1)。
| 表1 双亲及IIA-CSSL群体7个穗部性状2年的表型值 Table 1 Phenotypic values of seven panicle traits of parents and IIA-CSSLs across two years |
以粳糯稻A7444为父本, 籼稻II-32B为母本, 2007年夏季于南京农业大学江浦农学试验站杂交获得F1种子。同年冬季在海南省三亚市吉阳镇育种基地种植F1植株群体。在SSR分子标记鉴定为真实F1植株的群体中随机选3个单株去雄, 以II-32B为父本回交获得BC1F1种子。2008年夏季于南京农业大学江浦农学试验站种植BC1F1植株群体。抽穗期选取剑叶角度大于90° 的单株去雄, 用II-32B做父本回交。成熟期分单株收获BC2F1种子, 同时收取回交单株其他分蘖穗的自交种子BC1F2。2009年夏季于南京农业大学江浦农学试验站, 分别种植BC2F1株系和BC1F2株系群体。自2010年(BC3F1世代)至2014年(BC6F2世代), 每年均按株系种植, 利用双亲间呈现多态的141对SSR标记对各世代单株进行标记基因型分析, 根据单株标记基因型特征对候选单株进一步回交或自交, 最终衍生出66个株系组成的染色体片段置换系群体。
将双亲及66个CSSL株系种植于南京农业大学江浦农学试验站的试验田。2014年5月12日播种, 6月16日移栽; 单本栽插。株行距为17 cm× 25 cm。所有材料均种植3个重复, 每重复每个家系种植3行, 每行8株。常规田间管理。2015年5月12日播种, 6月15日移栽。栽插规格和田间管理同2014年。
成熟后每个小区取中间行整齐一致的3株主茎穗, 自然晾干后调查穗长、每穗一次枝梗数、每穗二次枝梗数、每穗总粒数、每穗实粒数、千粒重、粒宽7个穗部性状。
采用基于逐步回归线性分析结合极大似然估计(likelihood ratio test with stepwise regression, RSTEP-LRT)的QTL IciMapping 4.1软件中的CSL程序(ICIM-CSL)[11, 12]分别检测控制各性状的QTL。LOD阈值设为3.00, 当实际LOD值大于LOD阈值时, 就认为该区段存在1个QTL, 同时估算每个QTL的加性效应值和贡献率大小。遵循McCouch等[13]的原则命名QTL。
在基因的两端设计引物进行扩增, 扩增产物由金斯瑞公司测序。利用BioXM 2.6软件进行基因和氨基酸序列的比对。
II-32B/A7444组合染色体片段置换系的构建过程及候选单株的系谱图如图2所示。经过2010— 2014年对各世代单株的标记基因型鉴定, 以及入选单株的回交和自交, 共从BC2F4、BC3F3、BC4F2、BC3F5、BC4F4、BC5F3、BC6F2等世代的单株中获得候选置换系111个, 并从中获得一套由66个株系组成的以籼稻II-32B为背景、粳稻A7444为前景的染色体片段置换系群体, 取名IIA-CSSL群体(图3)。该群体所有置换系携带的置换片段总长度约为989.93 Mb, 相当于粳稻基因组总长的2.60倍, 覆盖了A7444全基因组的94.98%。
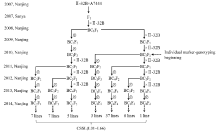 | 图2 IIA-CSSL群体构建过程× ,  分别表示杂交和自交。Fig. 2 Breeding procedure of the IIA-CSSL population× , 分别表示杂交和自交。Fig. 2 Breeding procedure of the IIA-CSSL population× ,  indicate hybridization and self-fertilization, respectively indicate hybridization and self-fertilization, respectively |
表1显示, 2014年母本II-32B二次枝梗数为47个, 父本A7444为25个, 相差近一倍; A7444的千粒重(44.4 g)接近II-32B千粒重(23.8 g)的2倍; 其余5个性状的双亲差异也都达到极显著水平。IIA-CSSL群体中, 除粒宽和千粒重外, 其余5个性状均表现出双向超亲分离; 一次枝梗数、每穗总粒数、每穗实粒数群体均值均显著高于高值亲本II-32B, 千粒重和粒宽介于亲本II-32B和A7444之间, 二次枝梗数和穗长与亲本II-32B差异不显著。2015年表现趋势与2014年一致。方差分析结果显示, 各穗部性状在IIA-CSSL群体的株系间存在极显著差异, 适合QTL的检测分析。
表2显示, 同一性状2年间相关系数都大于0.65, 均达到极显著水平, 表明各性状在不同年份中表现稳定。二次枝梗数与穗长、一次枝梗数、每穗总粒数和每穗实粒数表现出极显著正相关; 一次枝梗数与每穗总粒数和每穗实粒数呈极显著正相关; 同时, 穗长与每穗总粒数、每穗总粒数与每穗实粒数、粒宽与千粒重均呈极显著正相关。而穗长与粒宽、一次枝梗数、每穗实粒数和千粒重间的相关性不显著; 千粒重与二次枝梗数、每穗总粒数和每穗实粒数间的相关性也不显著。
| 表2 7个穗部性状间的相关系数 Table 2 Correlation coefficients among seven panicle traits |
2年共检测到控制7个性状的26个QTL (表3), 分布在第1、第2、第3、第5、第6、第7、第8、第9和第10染色体上, 2年均被检测到的QTL有13个。
| 表3 7个穗部性状2年定位到的QTL及其遗传参数 Table 3 QTLs detected for seven panicle traits and their genetic parameters in IIA-CSSL population across two years |
在2015年仅检测到一个穗长性状QTL qPL3, 贡献率为13.94%, 增效等位基因来自亲本A7444, 加性效应为1.41 cm。
2年共检测到5个一次枝梗数QTL, 分布在第1、第6、第8、第9和第10染色体上, 贡献率在7.60%~39.36%之间, 加性效应变幅为-1.56~ +1.45。qPBN1增效等位基因来自亲本II-32B, 其余4个位点增效等位基因均来自亲本A7444。2年均被检测到的QTL有4个, 分别是qPBN1、qPBN6、qPBN8和qPBN9。qPBN8的贡献率最大, 2年分别为20%和39%, 加性效应分别为0.76个和1.45个; 2年平均为1.105个。表明在qPBN8位点以A7444的2个等位基因替代II-32B的2个等位基因可使一次枝梗数增加2.21个。
2年共检测到4个二次枝梗数QTL, 分布在第2、第6、第7和第8染色体上, 贡献率在8.36%~23.20%之间, 加性效应变幅为-5.72~6.38。qSBN2增效等位基因来自II-32B, 其余3个位点的增效等位基因均来自A7444。qSBN6和qSBN82年均被检测到, qSBN8的贡献率最大, 2年分别为23%和20%, 加性效应分别为6.33个和6.38个; 2年平均为6.36个。表明在qSBN8位点以A7444的2个等位基因替代II-32B的2个等位基因可使二次枝梗数增加12.71个。
2年共检测到4个每穗总粒数QTL, 分布在第1、第2、第6和第8染色体上, 贡献率在8.21%~ 31.54%之间, 加性效应变幅为-19.85~ +33.36。qSPP2增效等位基因来自亲本II-32B, 其余3个位点增效等位基因均来自亲本A7444。2年均检测到的QTL有3个, 分别是qSPP2、qSPP6和qSPP8。qSPP8的贡献率最大, 2年分别为30%和32%, 加性效应值分别为30粒和33粒; 2年平均为31.5。表明在qSPP8位点以A7444的2个等位基因替代II-32B的2个等位基因可使每穗总粒数增加63粒。
2年共检测到3个每穗实粒数QTL, 分布在第2、第6和第8染色体上, 贡献率在11.08%~28.78%之间, 加性效应变幅为-23.58~ +33.39。2年均被检测到的qGPP8位点的增效等位基因来自A7444, 贡献率分别为29%和27%, 加性效应值分别为28粒和33粒。表明在qGPP8位点以A7444的2个等位基因替代II-32B的2个等位基因可使每穗实粒数增加61粒。
2年共检测到4个千粒重QTL, 分布在第2、第8和第10染色体上, 贡献率在11.00%~49.03%之间, 加性效应变幅为-2.62~ +3.06。qTGW2.1增效等位基因来自A7444, 其余3个位点增效等位基因均来自亲本II-32B。2年均被检测到的QTL有2个, 分别是qTGW2.1和qTGW2.2。qTGW2.1贡献率最大, 2年分别为49%和40%, 加性效应分别为3.06 g和2.69 g。表明在qTGW2.1位点以A7444的2个等位基因替代II-32B的2个等位基因可使千粒重增加5.75 g。
2年共检测到5个粒宽QTL, 分布在第1、第2、第5、第9和第10染色体上, 贡献率在12.34%~ 27.70%之间, 加性效应变幅为-0.13~ +0.14。qGW1增效等位基因来自亲本II-32B, 其余4个位点增效等位基因均来自亲本A7444。2年均被检测到的qGW10位点的贡献率分别为28%和16%, 加性效应值分别为0.12 mm和0.10 mm。表明在qGW10位点以A7444的2个等位基因替代II-32B的2个等位基因可使粒宽增加0.22 mm。
检测到穗部性状QTL成簇分布区间3个(图4)。第2染色体的RM3688-RM6617区间中, 来自A7444的等位基因可减少每穗总粒数, 同时增加千粒重。第6染色体的RM136-AP22区间和第8染色体的RM3572-RM5556区间, 均来自A7444的等位基因, 可同时增加一次枝梗数、二次枝梗数、每穗总粒数以及每穗实粒数。QTL成簇分布可能是一因多效或基因连锁引起。QTL成簇分布解释了性状间相关性的遗传基础。
 | 图4 IIA-CSSL群体中检测到的7个穗部性状QTL以及在附近位置已报道的穗部性状基因在染色体上的位置黑色图案为2014年检测到的QTL, 白色图案为2015年检测到的QTL。缩写同表1。Fig. 4 Chromosome locations of the QTLs for seven panicle traits detected in IIA-CSSL population and some other reported genes of panicle traitsThe black symbols stand for QTLs detected in 2014, the white symbols stand for QTLs detected in 2015. Abbreviations are the same as those given in Table 1. |
通过检索文献发现, 主效QTL qSBN6所在的第6染色体RM136 (8.75 Mb)-AP22 (10.94 Mb)区间内已有水稻抽穗期基因Hd1(9.34 Mb)被克隆, 该基因序列全长2285 bp, 共有2个外显子, 编码产物包含395个氨基酸, 能够降低穗部一次枝梗的数目, 导致穗粒数的减少[14]。II-32B和A7444的Hd1基因测序分析的结果显示, 相比于II-32B的Hd1, A7444中共检测到Hd1的16个变异。它们是第1外显子426~427 bp处的1个2 bp替换、695~728 bp处的1个33 bp缺失; 第1内含子93~94 bp处的1个2 bp插入; 第3内含子2233 bp处和2296 bp处各1 bp缺失; 以及其余11个位置各1 bp替换(图5-A)。第1外显子中的33 bp插入使A7444的预测氨基酸序列比II-32B少了11个氨基, 而第2外显子上1个由C到T的替换则导致A7444中Hd1翻译的提前终止, 使A7444的多肽链长度又少了38个氨基酸残基(图5-B)。
 | 图5 双亲及日本晴Hd1和DTH8/Ghd8的基因序列(A, C)及其预测氨基酸序列(B, D)比对Fig. 5 Gene sequence (A, C) and deduced peptide sequence (B, D) alignment of Hd1and DTH8/Ghd8 between two parents and Nipponbare |
在第8染色体RM3572 (3.93 Mb)-RM5556 (4.59 Mb)区间存在的抽穗基因DTH8/Ghd8(4.33 Mb)已被克隆, 该基因只含1个外显子, 编码产物包含297个氨基酸, 其迟熟等位基因延迟抽穗, 提高株高、每穗总粒数和枝梗数[15, 16]。我们分析II-32B和A7444的DTH8/Ghd8基因测序结果, 并比对Nipponbare中相应的基因序列显示, 在A7444与Nipponbare品种中, DTH8/Ghd8基因序列完全相同, 但在II-32B的外显子1312 bp处检测到1个1 bp插入(图5-C)。这个1 bp插入直接导致后续阅读框的移位, 使预测氨基酸序列少了7个氨基(图5-D)。测序结果表明这2个基因在双亲之间存在差异, 也证实了本实验检测到的QTL的真实性。
本研究利用IIA-CSSL群体定位到影响二次枝梗数的3个多效性区间。qSBN2所在第2染色体的RM5427-3688区间同时控制二次枝梗数、每穗总粒数和每穗实粒数QTL, 前人文献及相应数据库中未发现该区间关于二次枝梗数QTL的报道。在2年中均重复检测到qSBN6和qSBN8, 其所在区间即第6染色体的RM136-AP22和第8染色体的RM3572- RM5556, 同时还影响一次枝梗数、每穗总粒数以及每穗实粒数, 且增效等位基因均来自低值的供体亲本A7444, 表明该CSSL群体中控制这些性状的位点极可能存在紧密连锁或一因多效, 同时也从分子水平上解释了二次枝梗数与一次枝梗数、每穗总粒数和每穗实粒数间的极显著正相关。水稻基因组注释数据库(rice genome annotation project, http://rice. plantbiology.msu.edu/)信息和前人的报道显示, 在RM136-AP22区间中精细定位了Hd1[17], 该基因导致穗发育时顶端分生组织中类Terminal Flower 1基因上调、穗形成相关基因的表达提前, 可能通过影响叶片中成花素基因的表达进而影响作物的大田产量[18]; RM3572-RM5556区间中有DTH8/Ghd8基因被克隆, DTH8/Ghd8通过调节Ehd1、RFT1和Hd3a的表达, 在长日照条件下能够显著增加播种至抽穗天数、株高和每穗粒数[15], 且该基因能够上调控制基因MOC1的表达, 从而增加水稻的分蘖数、一次枝梗和二次枝梗数[16]。序列对比分析表明研究中检测到的qSBN6和qSBN8即Hd1和DTH8/Ghd8, 验证了抽穗期基因Hd1和DTH8/Ghd8对水稻穗部性状的影响。值得注意的是, qSBN8位点表现突出, 2015年对一次枝梗数贡献率达39%, 2014年和2015年对每穗总粒数贡献率均达30%, 说明该位点极富研究与利用价值。
供体亲本A7444比受体亲本II-32B抽穗期晚20 d左右, 每穗总粒数和二次枝梗数少; IIA-CSSL群体中, 携带A7444 Hd1和DTH8/Ghd8等位基因所在区间的株系, 抽穗期均延迟, 但一次枝梗数、二次枝梗数和每穗总粒数显著增加。二次枝梗的有无和多少是稻属植物种的特征。在稻属植物的22个种中, 短药野生稻(Oryza brachyantha A. Chev. et Roehr)、颗粒野生稻(Oryza granulata Nees et Arn ex Hook)和疣粒野生稻(Oryza meyeriana Baill)没有一次枝梗和二次枝梗; 药用野生稻(Oryza officinalisWall. ex Watt)、马来野生稻(Oryza ridleyiHook)和多年野生稻(Oryza rufipogonW. Griffith)有一次枝梗而无二次枝梗[19]。亚洲栽培稻种(Oryza sativa L.)经过长期的进化和人为驯化, 生产上栽培的品种均有一次枝梗和二次枝梗, 相对于野生稻祖先, 籽粒产量有了显著提高。研究表明, TAW1通过提高花序分生组织活性和抑制小穗分生组织特征相的转变来调控花序发育[4]。周峰等[20]研究发现, 独脚金内酯作为一种植物激素或其前体能够抑制植物的分枝和侧芽的生长, 而D53蛋白作为独脚金内酯信号转导途径的抑制子参与调控植物分枝的生长发育。据此推测A7444中可能存在类似TAW1或D53蛋白调控机制参与调控抑制枝梗的生长发育。
其余检测到15个QTL中, 部分QTL与前人报道的相关QTL的位置相近。如控制一次枝梗数的QTL中, 第9染色体RM105标记区域检测到的qPBN9与穗形基因DEP1位置接近, 该基因编码的G蛋白γ 亚基受到刺激后, 促进GDP转换为GTP, 活化异三聚体G蛋白, 导致α 亚基与β 、γ 亚基解离, 将信号传递给下游感受器, 使得穗颈节长度降低, 稻穗变密、并增加一次枝梗数[21]; 第10染色体RM5620标记附近检测到的qPBN10与Yoshida等克隆的TAW1基因位置相近, 该基因编码一个功能未知的核蛋白, 在茎端分生组织、花序分生组织以及枝梗分生组织高水平表达, 可增加一次枝梗数[4]。控制每穗总粒数的QTL中, 第1染色体RM6696标记区域检测到的QTL qSPP1与Koumoto等克隆的基因MOC2位置相近, moc2突变体缺乏胞质果糖-1, 6-二磷酸酶, 导致蔗糖供应不足, 表现出穗小、每穗粒数少[22]。控制千粒重的QTL中, 在第2染色体RM6617标记附近检测到的qTGW2.1贡献率最大, 2年分别为49.03%和40.27%, 徐建龙等[23]在该标记附近也定位到控制粒长、粒宽和千粒重的QTL; RM525标记附近检测到的qTGW2.2与粒形基因GS2比较接近, 该基因与转录共激活子OsGRF互作调控细胞伸长和细胞分裂, 影响水稻粒型和粒重[24]。控制粒宽的QTL中, 第5染色体RM7293标记附近检测到的qGW5与已克隆的GW5[8]位置接近。
对于本研究新发现的qSBN2位点, 在以后的工作中可精细定位和图位克隆, 深入研究控制水稻二次枝梗数基因的遗传作用机理。在育种工作中可聚合贡献率突出的有利等位基因qSBN8和qTGW2.1培育高产品种。
在IIA-CSSL群体中检测到同时控制二次枝梗数、每穗总粒数和每穗实粒数QTL的3个染色体区段。明确了qSBN6和qSBN8是Hd1和DTH8的新等位基因。发现了qSBN2是控制稻穗二次枝梗数的新位点。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|