第一作者联系方式: 王娟, E-mail: cottonwj@126.com; 董承光, E-mail: dcg318@163.com
棉花机械采收对品种的生育期、株型及对脱叶剂敏感度有较高的要求。本研究利用覆盖全基因组有多态性的214对SSR标记对118份含有一个或多个机采性状的种质资源的株高、始节高、始节位、第一果枝平均长度、生育期及脱叶率6个机采相关性状进行关联分析。利用Structure 2.3.1软件进行群体结构分析, 并结合2年2点12个重复的田间表型数据, 采用Tassel 5.0软件的混合线性模型MLM关联定位。结果检测到460个等位基因, 涉及905个基因型, 基因多样性指数平均为0.5151, PIC值平均为0.4587, 基因多样性指数和PIC值都大于平均数的标记有99个, 占总标记数的46.3%, 说明该批SSR标记具有较多的等位变异数和较高的遗传多样性。群体结构分析将118份供试材料划分为4个亚群, 结果显示各类群中材料与地理来源无对应关系。关联分析结果显示4种环境中, 在显著条件下( P<0.05), 共检测到124个与6个机采相关性状相关的位点, 对表型变异解释率范围为2.23%~14.15%; 其中在极显著条件下( P<0.01), 共检测到20个与机采相关性状相关的位点, 对表型变异解释率范围为4.84%~14.15%。基于本研究的结果, 鉴定出典型的载体材料11份, 分别为系7、金垦9号、Y11、豫棉18、AY-4、K2、朝阳棉2号、DZ22、中棉所43、C2和关农长早B14。以上发掘出的控制棉花适宜机采性状的优异等位基因及优异亲本资源, 可为机采棉的分子辅助选择育种提供理论依据。
Cotton suitable for mechanical harvest should have higher requirement in traits, for example, shorter growth period, ideal plant type and high sensitivity to defoliant. A total of 214 pairs of SSR with high polymorphism and uniform distribution on whole genome were used to scan polymorphism in 118 cotton varieties with one or more mechanical harvest-related traits. Molecular marker data and six phenotypic traits were analyzed by the method of MLM (mixed linear model) in Tassel 5.0 on the basis of population structure, analysis loci with elite allelic variation and typical materials carrying elite alleles were identified based on phenotypic effect values. We detected 460 alleles and 905 genotypes. The average genetic diversity index was 0.5151, and the average polymorphic information content (PIC) per marker was 0.4587. Ninety-nine markers achieved the aforementioned average values accounted for 46.3% of the total markers, shows that the SSR markers have more allelic variance and higher genetic diversity. All the 118 cotton varieties were divided into four subgroups by analysis of population genetic structure. There was no corresponding relation between each kind of group of materials and the geographical source. A total of 124 loci ( P<0.05) and 20 loci ( P<0.01) associated with mechanical harvest-related traits were detected by association analysis, with explained variance ranging from 2.23% to 14.15% and from 4.84% to 14.15% respectively. Based on the results of this study, we identified 11 typical materials, including Xi 7, Jinken 9, Y11, Yumian 18, AY-4, K2, Chaoyang 2, DZ22, Zhongmiansuo 43, C2, and Guanrongchangzao B14. The elite alleles and resources can be useful for marker-assisted selection breeding.
棉花是一种重要的经济作物之一, 集纤维、蛋白、油用于一体[1]。新疆具有棉花生产的优越自然生态条件, 是我国重要的植棉基地; 尤其在新疆生产建设兵团, 棉花种植的高度集约化和规模化, 为棉花机械化采收提供了有利的条件。目前, 新疆生产建设兵团在采棉机械研制、机采棉配套种植技术及机采棉清理加工等技术方面都有长足发展, 但机采棉育种研究相对较滞后, 大田推广种植的大多数品种只能兼顾机采的某个性状, 由于不适应机械采收而影响了棉花产量及品质, 降低了机采棉经济效益, 因此急需加大机采棉育种的研究力度。适宜机采的棉花品种要求具有综合性状, 尤其对生育期、株型及对脱叶剂敏感度要求高, 而这些性状是由较为复杂的数量性状基因控制。
近年来, 随着分子标记技术的不断发展和棉花遗传图谱的不断加密[2, 3], 使得控制棉花重要数量性状基因的发掘研究更为便利, 已有多个实验室报道了利用陆地棉种内不同类型群体构建的遗传图谱进行棉花产量和纤维品质等性状的QTL定位结果[4, 5, 6, 7, 8]。努斯热提· 吾斯曼等[9]通过对机采棉品种新陆早33和陆地棉遗传标准系TM-1构建的F2群体进行QTL定位, 共获得控制3个机采棉性状的5个QTL, 最大解释的表型变异率为12.64%。以上研究只是对机采棉的部分农艺性状对产量的贡献和SSR标记的初步定位, 由于陆陆群体标记的数量有限, 育种研究者对机采性状遗传及QTL所掌握信息量较少, 从而限制了机采棉育种的发展。关联分析方法作为一种剖析复杂数量性状的有效方法, 是目前研究的热点, 此法无需构建作图群体, 应用品种资源群体可以使检测的等位基因数目更多, 同时可以考察多个性状的大多数QTL关联位点及其等位变异, 并且已在玉米[10, 11]、大豆[12, 13]、小麦[14, 15]、大麦[16]等作物中得到应用。在棉花上, 不同国家研究学者应用此法通过不同生态区的形态鉴定, 结合SSR标记的基因组扫描, 发掘出主要农艺性状[17, 18, 19]、产量[20, 21, 22]、纤维品质[23, 24]、抗性[25]等的优异等位变异。但目前还未见机采相关性状优异基因发掘方面的报道。
本研究以本单位多年收集的具有一个或多个机采优异性状的陆地棉早熟品种、品系等种质资源为研究群体, 利用覆盖棉花遗传图谱的SSR标记进行全基因组扫描和关联分析, 发掘控制棉花适宜机采性状的优异基因、优异等位变异及优异亲本资源, 以期为机采棉育种中高效发掘和利用这些重要性状提供信息, 并为机采棉分子辅助选择育种提供理论依据。
选取来自新疆农垦科学院棉花研究所种质资源库, 具有一个或多个适宜机采性状的118份优异种质资源, 来源于我国西北内陆棉区、黄河流域棉区、长江流域棉区、北部特早熟棉区以及国外等地(表1)。于2013— 2014年分别在新疆石河子新疆农垦科学院棉花研究所试验基地(北疆)与新疆库尔勒兵团第二师农科所试验基地(南疆)种植试验材料, 采用完全随机区组设计, 双行区, 行长5 m, 株距10 cm, 平均行距35 cm, 3次重复, 人工点播, 膜下滴灌栽培, 以当地常规方法进行田间管理。
| 表1 118份陆地棉种质材料的名称及来源 Table 1 Name and origin of 118 upland cotton varieties |
在棉花成铃后期分别在各重复中每小区选中间连续10株调查株高、第一果枝始节高度、第一果枝始节位、每果枝第一果节平均长度; 于2013年9月10日和2014年9月10日, 喷施德国拜耳作物科学公司的脱叶剂脱吐隆悬浮剂和乙烯利混合液, 配方为脱吐隆225 g+乙烯利1800 mL hm-2, 分别在当天和施药后15 d调查选定10株的总叶片数, 计算脱叶率; 并记录出苗期、吐絮期和生育期。各性状调查方法参考棉花种质资源描述规范和数据标准[26]。
参考Zhao等[3]发表的四倍体棉花遗传图谱, 平均每8~10 cM选取一个SSR标记, 并进一步结合钱能等[27]、Song等[28]、薛艳等[29]、Sun等[30]、艾先涛等[31]的研究结果, 选取多态性高的引物557对进行扩增, 最终获得多态性的引物214对。SSR引物序列来自CMD (Cottonmarker database) (http://www. cottonmarker.org/), 由生工生物工程(上海)股份有限公司合成。分子标记名称中的大写字母, 如NAU、BNL等, 代表引物的来源, “ a” 、“ b” 、“ c” 、“ d” 表示一个标记在材料中有多个多态性位点, 位点顺序依次按分子量由大到小排列。DNA提取参照Paterson等[32]发表的CTAB法, SSR实验操作程序和PCR扩增参照Zhang等[33]方法, 并使用毛细管电泳分析仪Fragment Analyzer-XL960 SSR/Tilling对PCR产物参照说明书进行分析, 电泳后数据结果自动存于ProSize 2.0系统软件, 以行、列或单独的样品组合显示在数字胶视图中, 根据胶视图中DNA Ladder以及单个样品不同位点片段大小, 输入预设范围, 系统即可自动读取0、1数据并以Excel形式输出。
利用R语言将2年2点4种环境的表型数据进行BLUP分析, 绘制4种环境的表型性状Boxplot图; 利用PowerMarker V 3.25软件[34]进行基因型遗传多样性分析, 获得等位基因频率、等位基因数、基因多样性指数及基因型多态信息含量。利用Structure 2.3.1软件[35]进行群体结构估计, 参数设置为: K值选取1~10, 重复次数为5; 将MCMC (markov chain monte carlo)开始时的不作数迭代设为50 000次, 再将不作数迭代后的MCMC设为50 000次, 其余参数采用软件默认的设置。根据ln P(D)计算Δ K, 依据Δ K值选择一个合适的K值[36], 并得到该K值对应的Q矩阵(第i材料的基因组变异来源于第k群体的概率)。
利用软件Tassel 5.0[37]将基因型数据生成亲缘关系矩阵(K矩阵), 结合等位变异数据、基因型数据、各个环境的表型值、Q矩阵, 利用MLM (mixed linear model)[38]进行性状和标记之间的关联分析, 并计算标记位点在P< 0.05和P< 0.01时对表型变异的贡献率(R2)。在已获得关联位点的基础上, 再进行优异等位变异的发掘, SSR位点等位变异表型效应计算方法为ai = (Σ xij/ni) - (Σ Nk/nk)。其中ai代表第i个等位变异的表型效应值, xij为携带第i个等位变异的第j材料性状表型测定值, ni为具有第i等位变异的材料数, Nk为所有材料的表型测定值, nk为材料数。若ai为正, 则认为该等位变异为增效等位变异, 反之为减效等位变异[39]。最终获得与表型性状显著关联的位点等位变异、表型效应及典型品种。
118份种质资源材料的6个主要机采性状2年2点4种环境的表型结果如表2所示。各性状平均变异系数的变幅在1.71%~11.12%之间, 其中每果枝第一果节平均长度变异系数最大为11.12%, 说明该性状受生态环境影响较大; 生育期变异系数最小为1.71%, 说明该性状较稳定。6个性状的遗传力变化范围介于0.43~0.72之间, 其中最高的为株高, 值为0.72, 最低的为脱叶率, 值为0.43。
| 表2 118份材料4个环境下表型性状BLUP结果 Table 2 BLUP results of traits for 118 upland cotton varieties in four environments |
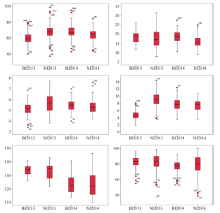
由图1可见, 4个环境中同一性状在同一地点的不同年份变化趋势和幅度较为一致的性状为始节高和脱叶率; 4个环境中同一性状在同一年份的不同地点变化趋势和幅度较为一致的性状为生育期和始节位。总的来看, 6个性状在2个地点不同年份的性状变化趋势相对稳定。
表3表明, 214个SSR标记位点在118份材料中检测到460个等位基因, 涉及905个基因型, 不同的SSR标记检测到的基因型数目不同, 变幅为2~14个, 平均为4.23个, 其中大于平均数的标记有56个, 占所有多态性标记数的26.2%。214个标记所揭示的基因多样性指数平均为0.5151, 变幅为0.0333~0.8725; PIC值平均为0.4587, 变幅为0.0328~0.8594, 基因多样性指数和PIC值都大于平均数的标记有99个, 占总标记数的46.3%; 如标记NAU3995, 等位基因数和基因型数分别为5和14, 基因多样性和PIC值分别为0.8492和0.8376。总体而言, 本试验选用标记具有较多的等位变异数和较高的基因多样性。
| 表3 214对多态性SSR标记在118份材料中的引物多态性信息 Table 3 Polymorphism information of 214 SSR loci in 118 upland cotton varieties |
Structure 2.3.1软件群体结构分析结果显示, 随着假定亚群数K值的逐渐增大, ln P(D)值也呈持续增大的趋势, 没有找到一个拐点, 无法判断k的取值。因此, 决定采用通过Δ K来确定群体结构的K值[40]。在k=4时, Δ K出现峰值拐点(图2-B), 由此推测该群体材料被分为4个亚群。依据亚群中个体的概率大于等于0.6时, 个体被分配到相应聚类的亚群; 亚群中个体的概率小于等于0.6时, 个体被分配到一个混合的类群的分配原则[41], 分析各供试材料对应的Q值(各供试材料被分配到相应群中的概率)。结果表明118份材料归属为4个亚群(图3)。分别包含34、19、23和12份材料, 4个亚群各占比例为28.8%、16.1%、19.5%和10.2%; 另有30份材料在任意一个亚群内Q值均小于0.6而无法明确其归类, 单独划为一个混合群, 占比例为25.4%。分群结果显示各类群中材料与地理来源无对应关系。
采用Tassel软件中的混合线性模型(MLM)方法, 以2年2点4种环境的表型数据BLUP分析后的育种值为数据依据, 对118份材料的6个机采相关性状进行关联分析(表4)。结果显示, 4个环境中, 在P< 0.05显著条件下, 检测到124个与6个机采相关性状相关的位点, 对表型变异解释率范围为2.23%~ 14.15%, 平均值为4.73%。而在P< 0.01极显著条件下, 共检测到20个与机采相关性状相关的位点, 对表型变异解释率范围为4.84%~14.15%, 平均值为7.92%。其中与株高相关的位点有3个, 表型变异解释率范围为6.13%~10.27%, 平均值为7.52%, 贡献率最高的位点为NAU3095b; 与果枝始节高相关的位点有2个, 表型变异解释率范围为6.21%~6.44%, 平均值为6.33%, 贡献率最高的位点为CIR0170b; 与始节位相关的位点有4个, 表型变异解释率范围为5.84%~9.67%, 平均值为7.33%, 贡献率最高的位点为CIR0170b; 与每果枝第一果节平均长度相关的位点有6个, 表型变异解释率范围为6.49%~10.68%, 平均值为7.97%, 贡献率最高的位点为DPL0209; 与生育期相关的位点只有1个, 为NAU1362c, 表型变异解释率为4.84%; 与脱叶率相关的位点有4个, 表型变异解释率范围为6.01%~14.15%, 平均值为10.29%, 贡献率最高的位点为NAU2560b。
| 表4 表型性状与标记的关联分析结果 Table 4 Association analysis results of phenotypic traits |
表5表明, 有17个标记位点同时与2个以上性状相关联。其中NAU4926c同时与株高、始节位、生育期、脱叶率4个性状相关联; 5个标记位点NAU0911b、NAU2954b、NAU3277a、NAU3277b、NAU6966同时与3个性状相关联; 11个标记位点BNL3089c、BNL3257a、BNL3997c、CIR0170b、NAU2317c、NAU3031a、NAU3052a、NAU3053a、NAU3095b、NAU4926a、NAU5189b同时与2个性状相关联。
| 表5 4个环境下与2个以上性状关联的位点 Table 5 Loci associated with more than two phenotypic traits in four environments (P< 0.05) |
依据关联分析结果, 与6个主要机采性状显著关联(P< 0.01)的SSR位点有20个, 鉴定典型的载体材料11份(表6), 分别为系7、金垦9号、Y11、豫棉18、AY-4、K2、朝阳棉2号、DZ22、中棉所43、C2和关农长早B14。
| 表6 与机采性状显著关联的位点等位变异对应的表型效应 Table 6 Phenotypic effect of marker alleles at loci significantly associated with mechanical harvest-related traits |
株高关联位点等位变异中, NAU3095b是增效表型效应(+0.84 cm)最大的等位变异, 典型材料是C2; BNL3089c是减效表型效应(-1.65 cm)最大的等位变异, 典型材料是关农长早B14。始节高关联位点等位变异中, CIR0170b是增效表型效应(+0.92 cm)最大的等位变异, 典型材料是AY-4; NAU5499b是减效表型效应(-0.52 cm)最大的等位变异, 典型材料是朝阳棉2号。始节位关联位点等位变异中, CIR0170b是增效表型效应(+0.20 cm)最大的等位变异, 典型材料是AY-4; NAU4926c是减效表型效应(-0.12 cm)最大的等位变异, 典型材料是K2。第一果枝平均长度关联位点等位变异中, DPL0209是增效表型效应(+0.17 cm)最大的等位变异, 典型材料是Y11; HAU1952a是减效表型效应(-0.58 cm)最大的等位变异, 典型材料是系7。生育期关联位点等位变异只有1个NAU1362c, 表现为增效表型效应(+0.60 cm), 典型材料是豫棉18。脱叶率关联位点等位变异中, NAU2560b是增效表型效应(+7.77 cm)最大的等位变异, 典型材料是中棉所43; NAU0874a是减效表型效应(-5.94 cm)最大的等位变异, 典型材料是DZ22。
中国新疆地广人稀, 植棉劳动力紧张, 人工采棉已经不能适应棉花规模化发展的需要, 因此, 大力发展机采棉技术是提高棉花生产经济效益, 推进新疆农业现代化的重大举措。从棉花品种角度出发, 机采棉品种要适应机采, 除达到一般品种的产量、品质特性外, 还需要品种具有较好的早熟性, 株型紧凑, 果枝始节位高, 对脱叶剂敏感等关键机采性状, 才能达到农机农艺融合, 提高机采棉经济效益。本研究中考查的6个性状中, 株高、始节高、始节位、每果枝第一果节平均长度等性状反映合理株型性状, 生育期性状反映早熟性性状, 脱叶率反映对脱叶剂敏感性程度。对这6个性状进行关联分析, 挖掘优异等位变异, 对机采棉育种意义较大。
群体结构是影响关联分析的主要因素之一。群体结构的存在会通过影响位点LD水平而影响关联分析的准确性, 造成关联分析出现假阳性[42], 即检测到的多态性位点和表型性状的关联不是由功能位点造成的, 而是由亚群的存在所导致。已有研究表明, 同时使用群体结构信息(Q值)和品种间亲缘关系(Kinship值)的MLM (Q+K)模型要优于单独使用Q值或Kinship值的GLM(Q)和MLM(K)模型[22, 43]。本研究对多位点基因型数据采用了Structure软件中的混合线性模型(MLM)(Q+K)进行群体结构的分析和校正, 计算每个材料归入各亚群的概率(Q值), 并将其作为协变量纳入回归分析, 有效的矫正了亚群存在导致的伪关联。本研究所选118份材料的等位变异频率特征类型数k = 4时, Δ k达到峰值拐点, 即可将所选材料分为4个亚群。但其所分亚群与所选材料的地理来源无直接关系。其原因可能为各生态棉区间棉花品种交流较为广泛, 造成遗传背景较为复杂, 不能完全与生态区来源或系谱来源对应。
本研究共检测到124个与6个机采相关性状相关的位点, 其中在P< 0.01极显著条件下, 共检测到20个与机采相关性状相关的位点, 对表型变异解释率平均值为7.92%, 在机采棉育种实践中可利用这些等位基因进行材料的等位条带分子标记辅助选择研究; 另外, 本研究在所有检测到的关联位点中, 有17个标记位点同时与2个以上性状相关联, 其中NAU4926c位点同时与4个性状相关联。表明这些标记可以用于同时改良多个育种目标性状, 以实现多个育种目标性状的聚合育种研究。
挖掘与优异机采性状相关的优异种质是机采棉育种研究的重要内容之一。本研究在关联分析基础上, 挖掘得到11份含有优异机采性状的典型材料。其中C2材料含有与株高性状相关的优异等位基因, AY-4材料聚合与始节高和始节位性状相关的优异等位基因, 系7材料含有与第一果枝平均长度性状相关的优异等位基因, 中棉所43品种含有与脱叶率性状相关的优异等位基因, 这些可作为机采棉育种材料中株型紧凑、果枝始节位高及对脱叶剂敏感的骨干亲本。当然, 上述所挖掘的优异等位基因及载体材料还需进一步在大群体和更多环境中验证。
4种环境中, 在显著条件下(P< 0.05), 共检测到124个与6个机采相关性状相关的位点, 对表型变异解释率范围为2.23%~14.15%; 其中在极显著条件下(P< 0.01), 共检测到20个与机采相关性状相关的位点, 对表型变异解释率范围为4.84%~14.15%; 鉴定出典型的载体材料11份, 分别为系7、金垦9号、Y11、豫棉18、AY-4、K2、朝阳棉2号、DZ22、中棉所43、C2和关农长早B14。
致谢: 感谢华中农业大学林忠旭教授和聂新辉博士, 中国农业大学杨晓红教授和徐根博士在数据分析方面给予的指导和帮助。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|