第一作者联系方式: 李国君, E-mail: liguojun911@163.com, 马艺文, E-mail: mayiwen3070@163.com
以我国玉米育种中常用的16份自交系为材料, 通过对SNAC ( Stress-responsive NAM, ATAF1/2, CUC2)基因编码区及上游启动子区800 bp核苷酸序列进行测序, 检测SNAC基因在不同杂种优势类群材料中的遗传变异。在12个SNAC基因中, 其中有4个基因在上游800 bp区检测到遗传变异, 有4个基因变异位点超过30个, 多态性较高。虽然大多数SNAC基因变异以SNP (Single nucleotide polymorphism)为主, 但在 ZmNAC031467基因中检测到较多的插入缺失变异(InDel), 达到基因总遗传变异的63.3%。通过PLACE软件对上游启动子有变异的4个基因进行3种耐逆结合元件的预测, 结果显示4个基因均含有3种耐逆结合元件, 但是基因突变对启动子结合元件的影响较小。再对检测到的遗传变异进行核苷酸多态性分析和中性检验, 有7个SNAC基因核苷酸多态性较高, 其中 ZmNAC080308基因的多态性达到0.00962, 推测这些基因在遗传漂移过程中受自然选择影响较大。利用 t检验初步发现 ZmNAC070395和 ZmNAC080398基因的2个变异位点与耐旱相关性状关联, 为进一步分析SNAC基因核苷酸变异与耐旱性状的关系提供一定的借鉴。
The coding regions and their upstream 800 bp promoter regions of SNAC genes ( Sress-responsive NAM, ATAF1/2, CUC2) were sequenced in 16 maize inbred lines commonly used in China. Among 12 SNAC genes, genetic variations in promoter region were only identified in four SNAC genes, and more than 30 variations were identified in four SNAC genes, showing higher polymorphism in the four genes than other in SNAC genes. Although most of the SNAC genes were mainly SNP (Single nucleotide polymorphism) mutations, more insertion/deletion mutations were detected in ZmNAC031467 gene, reaching 63.3% of the total genetic variations. The PLACE software was used to predict three kinds of stress-tolerant binding elements in SNAC gene, but little effect was found to be related with the variations. Additionally, high nucleotide polymorphisms were identified in seven SNAC genes, especially with the highest π value of 0.00962 in ZmNAC030308, which suggested that they were greatly influenced by natural selection in the genetic drift. With the t-test, two mutations of ZmNAC070395 and ZmNAC080398 genes were associated with drought-tolerant traits, which provides references for further analysing the relationship between nucleotide variation in SNAC and drought tolerance traits.
玉米(Zea maysL.)是我国第一大作物, 在国民经济发展和国家粮食安全中占有举足轻重的地位。干旱、高盐、异常温度等非生物胁迫是植物生长发育过程中面临的主要非生物逆境胁迫因子, 对作物造成伤害, 据调查, 全球作物产量减少约70%是由非生物胁迫直接影响的[1]。因此, 提高抵抗非生物胁迫能力是目前玉米育种的主要目标之一。在基因的非生物耐性应激防御中, 转录调节在作物生长发育和环境互作中起到非常重要的作用, 作物中一些与逆境相关应答基因的表达以及作物抗逆性的提高与这些转录因子的调控是息息相关的[2]。NAC (NAM, ATAF1/2, CUC2)转录因子是植物特有的一类转录因子家族, 研究显示NAC转录因子在植物生长发育中有重要的调控作用, 同时也参与对非生物胁迫和病原菌侵染等生物胁迫的抗逆反应[3]。本研究通过检索转录因子数据库得到玉米NAC转录因子蛋白, 并对聚类获得的SNAC亚族基因进行遗传变异分析, 旨在为挖掘耐旱相关基因及其遗传变异提供参考依据。
目前研究发现, NAC家族和植物顶端分生组织的形成、器官边界的建立有关[4]。转录因子NAC最初发现于矮牵牛的NAM, 以含有一致蛋白质保守序列的NAM、ATAF1/2和CUC2首字母命名[5]。NAC转录因子家族成员N末端都具有大约150个氨基酸残基组成的高度保守的DNA结合结构域, 可以形成螺旋-转角-螺旋的三维结构, 并能特异结合目标DNA和蛋白。它能识别并结合核心序列CATGTG和CACG[6]。其C端为不稳定的转录激活域, 具有高度多态性, 含有较高频率的简单氨基酸, 而且富含丝氨酸、苏氨酸、脯氨酸、谷氨酸等重要氨基酸, 这些可能与NAC的功能多样化有关[7]。目前, 在多个物种中都报道存在大量的NAC转录因子, Fang等[8]根据NAM保守的氨基酸序列把水稻基因组中140个NAC转录因子经聚类分析分成五5个亚族。Shen等[9]将11个物种1232个NAC转录因子聚类为8个亚族, 每个亚族又分成不同亚群。Nuruzzaman等[10]把在水稻和拟南芥中搜索到的151个和117个NAC转录因子共聚类为16个亚族。Nakashima等[11]将水稻、拟南芥等4个物种的NAC转录因子家族分成6个亚族。虽然不同学者对物种NAC转录因子的聚类分析结果不尽相同, 但研究发现大多数耐逆相关的NAC转录因子都被分在同一个亚族。因此, 把这一类NAC转录因子亚族统称为SNAC (Stress-respon- sive NAC)转录因子。
与拟南芥和水稻相比, 对玉米SNAC家族的研究相对薄弱[12]。玉米对非生物逆境非常敏感。水分不足或高温伤害可以造成玉米植株矮小、果穗小、授粉不良、籽粒败育、有效穗数少、穗粒重低、产量降低。2009年有专利表明ZmSNAC1~ZmSNAC5是与非生物逆境胁迫相关的转录因子(WO 2009/ 052476 A2)。此外, Lu等[13]在拟南芥中验证了玉米ZmSNAC1基因的抗脱水功能。Mao等[14]在玉米第10染色体上的SNAC基因ZmNAC111 (GRMZM2G 127379)上游572 bp处启动子区82 bp发掘一个微型回文转座因子(MITE)插入位点, 该位点普遍存在于温带玉米种质中, 而热带玉米种质中缺失, 证明与玉米苗期耐旱性有关。因此, 考虑到SNAC家族在玉米抵抗非生物逆境上的功能, 本研究以我国常用的16份不同杂种优势类群的玉米自交系为材料, 以SNAC基因为研究对象, 通过测序对基因遗传变异进行研究, 初步研究玉米中SNAC基因的核苷酸多态性和基因进化过程中受到的选择压力, 并对SNAC亚族基因启动子区有变异的进行耐旱相关结合元件的分析以及初步的耐旱性关联分析。
为了分析玉米SNAC基因中的遗传变异, 选用玉米育种及生产中常用的16个自交系(表1), 其中含杂种优势亚群PB、LAN (Lancaster)、SPT (唐四平头)、LRC (旅大红骨)血缘自交系各3份, Reid血缘自交系4份。研究材料涵盖了我国玉米种质的五大杂种优势类群, 大多数选自我国广泛种植的杂交种的亲本, 多样性丰富。
| 表1 研究用玉米自交系的基本情况 Table 1 Basic information of tested maize inbred lines |
分别挑选各玉米自交系大小一致且饱满的玉米种子20粒, 均匀地摆放于装有栽培土的塑料花盆中, 将土浇灌至湿润但不漏水的状态, 2~3 d浇灌一次, 待幼苗长至三叶期时将幼苗剪到自封袋中放入超低温冰箱保存以备提取DNA使用。采用CTAB小量法[18]提取16份玉米自交系总DNA。
根据聚类获得的16个玉米SNAC基因编码区及启动子区800 bp核苷酸序列, 使用Primer 5.0软件设计引物, 采用巢式引物扩增序列较长的基因。16个SNAC基因中扩增出12个基因的全长, 引物信息详见表2, 由美吉生物公司完成12个SNAC基因的PCR扩增、产物回收与纯化及样品测序。
| 表2 扩增12个SNAC基因序列所用引物信息 Table 2 Basic information of primers for 12 SNAC genes |
通过ClustalW软件比对玉米SNAC基因序列, 应用PLACE软件分析启动子区域抗逆结合元件, 从MaizeGDB中查找相应基因的启动子区序列导入PLACE中根据需要的元件进行筛选, 又将测序后启动子区有变异的进行结合元件分析。采用DNAsp 5.0软件分析基因序列, 应用π 评估SNAC基因编码区及800 bp启动子区段的核苷酸多态性; 利用Tajima’ s D进行中性检验; 应用SAS软件对耐旱选择指数和变异位点进行t检验分析。
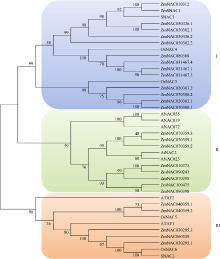
前期研究中的聚类分析表明, 其中的26个NAC蛋白分布于SNAC亚族中, 被称为玉米SNAC转录因子, 编码它们的16个基因被称为玉米SNAC基因[19]。进一步根据序列特征对这26个未知的玉米SNAC转录因子进行族内聚类分析(图1), 研究发现可以将它们分成3组, 第1组含有4个已知NAC转录因子ZmSNAC1、SNAC1、OsNAC4、OsNAC3及13个玉米SNAC转录因子; 第2组含有ANAC055、ANAC019、ANAC072、AtNAC2、ANAC025及8个玉米SNAC转录因子; 第3组包括5个已知的NAC转录因子ATAF2、OsNAC5、ATAF1、OsNAC6、SNAC2及5个玉米SNAC转录因子。其中拟南芥和水稻中的SNAC1、OsNAC4、ANAC055、ANAC019、ANAC072等基因均已报道与耐逆性相关, 可以提高植物的耐旱性[19]。根据比对结果, 对序列相近的组内玉米SNAC转录因子的研究可以借鉴拟南芥和水稻中已知的NAC转录因子的研究内容。
植物启动子中包含着许多响应非生物胁迫的保守元件, 调控下游功能基因的时空表达。为了阐明SNAC亚族基因启动子区的保守元件可能的调控机制, 对启动子区有变异的4个基因800 bp启动子区进行分析, 鉴定了与逆境相关的顺式作用元件。应用PLACE软件, 鉴定启动子区3种耐逆响应元件, 即干旱/脱水响应元件(DRE)、低温响应元件(LTRE)和脱落酸响应元件(ABRE)[20], 基于B73序列4个基因启动子的研究结果, 4个SNAC亚族基因中均检测到3种元件。ZmNAC030295基因中含有4个DRE元件、2个LTRE元件和5个ABRE元件, DRE元件主要在300~700 bp间, LTRE元件在400~500 bp间, ABRE元件在400~700 bp间; ZmNAC040359基因含有2个DRE元件、2个LTRE元件和4个ABRE元件, DRE元件主要在600~700 bp间, LTRE元件在50~700 bp间, ABRE元件在500~700 bp间; ZmNAC050302基因含有5个DRE元件、3个LTRE元件和7个ABRE元件, DRE元件主要在200~800 bp间, LTRE元件在200~600 bp间, ABRE元件在400~ 800 bp间; ZmNAC080308基因中含有2个DRE元件、2个LTRE元件和3个ABRE元件, ABRE元件分布在100~200 bp和700~800 bp之间, DRE元件在300~400 bp间, LTRE元件在600~700 bp之间(图2)。
 | 图2 玉米NAC基因启动子区域抗逆相关顺式作用元件的分布Fig. 2 Distribution of stress-related cis-elements in the promoters of NAC genes |
对突变后的4个SNAC基因启动子区逆境结合元件分析表明, 4个基因启动子区共48个多态性位点(29个单体型)分别检测到逆境结合元件, 发现3个基因ZmNAC030295、ZmNAC040359和ZmNAC 080308启动子区结合元件在数量上没有任何变化(与B73序列比较), 只有ZmNAC050302基因突变后ABRE元件数量减少2个, 说明SNAC亚族的这些基因启动子变异后对结合元件影响较小。
16个SNAC基因中除了4个基因(ZmNAC050302、ZmNAC060339、ZmNAC070359、ZmNAC070300)由于测序数据质量差被淘汰外, 共分析了12个SNAC基因的遗传变异。在启动子区和编码区总共检测到393个变异位点, 其中检测到SNP 237个, 插入缺失156个, 各个基因在玉米的10条染色体上位置, 变异位点分布在基因上的位置见图3。
在这12个基因中, 在启动子区检测到变异的有4个, 分别是ZmNAC030295、ZmNAC040359、ZmNAC050302和ZmNAC080308; 多态性位点超过30个的基因有4个, 分别是ZmNAC031467、ZmNAC080398、ZmNAC080308和ZmNAC100475; 检测到遗传变异以SNP位点为主的基因主要有ZmNAC010312、ZmNAC010373、ZmNAC050302和ZmNAC070395, 仅检测到3个插入缺失; 以插入缺失为主的基因有1个, 是ZmNAC031467, 达到基因总遗传变异的63.3%。
核苷酸多态性指标π 反映了不同材料之间碱基的差异情况, 用于衡量群体中的多态性程度, 是衡量遗传多样性的重要指标, 群体的变异程度越高, 其遗传多态性越丰富[21]。在本研究中, 检测到ZmNAC080308的核苷酸多态性最高(π = 0.00962)。另外, ZmNAC010312、ZmNAC030295、ZmNAC050302、ZmNAC080398、ZmNAC090241和ZmNAC100475基因π 值较高, 说明这7个基因群体的变异程度较高, 遗传多态性更丰富; ZmNAC010373、ZmNAC020303 (最低π = 0.00051)、ZmNAC031467、ZmNAC040359和ZmNAC070395基因π 值较低, 说明这个基因群体的变异程度较低(表3)。
| 表3 核苷酸多样性与中性检测 Table 3 Nucleotide diversity and neutrality test |
Tajima’ s D检验是鉴定目标DNA序列在进化过程中是否遵循中性进化模型。如果是负的显著结果, 那么这个结果可能是负选择造成的, 反之则受到平衡选择[22]。本研究利用有限的群体针对自然选择是否影响SNAC亚族基因初步分析, 通过使用Tajima’ s D值来检测群体是否符合中性进化模型(表3), 其中大多数SNAC基因差异不显著, 说明这9个基因在群体中未偏离中性进化, 基因突变受自然选择影响较小; ZmNAC020303、ZmNAC031467基因的Tajima’ s D值分别为-2.21101、-2.29433, 达到极显著水平, 说明这2个基因受负向选择压力较大, 突变受自然选择影响较大; 同时ZmNAC050302基因Tajima’ s D为2.14811, 达极显著水平, 说明这个基因受正向选择压力较大, 基因突变受自然选择影响较大。
t检验适用于总体标准差未知且样本容量小于30的小群体范围。本实验利用t检验, 对12个SNAC基因的393个多态性位点与耐旱选择指数(表4)进行差异显著性分析。结果在P-value < 0.01水平上检测到2个突变位点差异显著, 分别是位于第7染色体上的ZmNAC070395基因的553 bp位点和第8染色体上的ZmNAC080398基因的226 bp位点。初步推测这2个基因变异位点可能对耐旱性有着一定的影响, 需要进一步的验证(表4)。
| 表4 SNAC基因多态性位点与选择指数差异显著性分析 Table 4 Significant polymorphic variations in SNAC gene associated with drought-tolerance selection index |
在植物整个生育期中, NAC家族成员的功能具有多样性, 例如调控植物生长进程, 促进顶端分生组织的发育, 侧根形成, 次生细胞壁的合成等[23]。本研究通过对SNAC家族基因的进一步聚类分析得知, 由于不同物种蛋白质序列相似性较高, 相似的基因可能含有相同的功能性基序, 所以推测它们能与已知基因聚到一起和已知基因具有相似或相同的功能。
玉米中响应逆境的NAC基因有许多已经被证实, ZmNAC55在拟南芥中过表达同样增强了植株的耐逆性; ZmNAC111在拟南芥和玉米中的过表达都证明该基因使植株抗旱性增强; ZmSNAC1在拟南芥中过表达能够响应干旱[13]。水稻中相关的NAC基因SNAC1[24]同样能够响应干旱、高盐、冷和ABA; OsNAC4和ZmNAC080308基因的遗传进化距离较近, OsNAC4基因的功能是调控过敏性细胞死亡[25, 26], 因此ZmNAC080308基因极有可能参与植物响应逆境胁迫和调控植物生长发育中的过敏性细胞死亡; 通过RNA干扰和基因过表达2种手段对OsNAC5响应非生物胁迫研究发现, RNA干扰的植株对干旱、冷和盐胁迫比野生型的抗性降低, 然而OsNAC5在拟南芥和水稻中过表达能够增强对这些非生物胁迫的抗性[27]; 此外, OsNAC6基因能够被干旱、ABA、冷和盐所诱导[28]; SNAC2在水稻中受干旱、盐、冷和ABA诱导, 且在转基因植株过表达能够提高植物耐性, 特别是对干旱和ABA胁迫的耐性[29]; 另外, 在冷胁迫时, 野生型全部死亡, 而转基因植株仍然有50%的存活率。ANAC055、ANAC019、ANAC072 (RD26) 3个拟南芥NAC基因都能被干旱、高盐或低温所诱导[30]; ATAF1是拟南芥中第一个被发掘的NAC基因, 它被干旱和ABA强烈诱导, 对拟南芥幼苗进行控水试验发现, 正常浇水条件下ATAF1表达量在很低的水平, 干旱胁迫下ATAF1上调表达, 说明隶属于ATAF1一类的SNAC转录因子具有响应逆境胁迫的功能[31]。
这些胁迫相关的SNAC转录因子有一个共同点, 那就是在起始密码子前1500 bp区域内(通常认为的启动子区域)都包含有许多胁迫相关的顺式作用元件。本研究中对启动子区有变异的4个基因ZmNAC030295、ZmNAC040359、ZmNAC050302、ZmNAC080308的启动子区800 bp所含逆境相关顺式作用元件进行分析, 发现了3种元件: 干旱/脱水响应元件(DRE)、低温响应元件(LTRE)和脱落酸响应元件(ABRE)。并结合拟南芥及水稻的SNAC基因的聚类分析, 能够进一步推断SNAC转录因子的功能与其启动子区域所包含的这些逆境相关顺式作用元件有着紧密联系。顺式作用元件是由大约5~20 bp的核苷酸编码组成的保守基序, 决定了NAC转录本的启动能力[32]。研究发现, 寒冷与脱水响应元件(C repeat/DRE/CRT)是一种不依赖于ABA的调控元件, 在冷害、高盐胁迫下都能起一定的调控作用。当DRE/CRT结合蛋白DREB1/CBF在拟南芥过表达时, 改变了40多个胁迫诱导基因的表达, 从而对严寒、高盐、干旱的耐受性发生改变[33, 34, 35]。植物激素ABA在植物生长发育各个阶段及响应非生物胁迫中发挥着重要作用[36]。例如, 亮氨酸拉链蛋白与ABA响应元件结合后, 在依赖ABA的调控系统中成为一种转录激活因子[37, 38]。目前关于SNAC转录因子启动子顺式作用元件的研究已有许多证实。水稻SNAC基因OsNAC5、OsNAC6启动子区都含有ABA响应元件ABRE[39]; SNAC1和OsNAC3的启动子包含干旱响应元件(DRE)[40]。SNAC1启动子还包含LTRE、HSE、ABRE等低温、高热和ABA响应元件[41]。拟南芥SNAC基因ATAF1启动子同样含有非生物胁迫的顺式作用元件ABRE、DRE/CRT[31]。对拟南芥NAC转录因子ANAC072启动子1000 bp序列研究中发现了DRE元件[42]。另外, Mao等[14]在玉米ZmNAC55的启动子区段检测到一些胁迫相关顺式作用元件ABRE、CRT、LTRE、DRE等。而ZmNAC55又与SNAC1和ZmSNAC1具有很近的同源关系。
单核苷酸多态性是目前发现的最丰富和多样的遗传变异, 玉米中单核苷酸变异非常丰富, 研究发现单核苷酸变异与玉米的基因功能息息相关。因此, 发掘并解析玉米SNAC基因中的单核苷酸多态性对遗传研究非常重要, 玉米基因组中许多SNP或In/Del与基因调控和功能息息相关, 尤其是在逆境适应性方面起着决定性作用[43]。植物基因组序列变异是导致性状发生的关键, 如果发生在编码区, 一个碱基的变化有可能改变一个蛋白质的功能, 如果在植物的调节基因上发生等位变异, 则有可能影响整个基因的表达水平, 并可以导致定量突变。生物体中大多数自然发生的遗传变异是以单个碱基变化或小的插入/缺失表现的, 这种变异即是SNP或In/Del(单核苷酸多态性及插入缺失)。研究发现, 许多SNP或In/Del无论在结构基因还是在调节基因中都是表型变异的驱动者, 并决定了种质差异。
Mao等[14]对玉米第10染色体上SNAC基因ZmNAC111 (ZmNAC100475) ATG前572 bp处发现了一个82 bp的微型回文结构MITE的插入, 这导致了甲基化的发生, 并抑制ZmNAC111的表达。它们普遍存在于温带玉米种质中, 而热带玉米种质中缺失, 证明与玉米苗期耐旱性有关。Yang等[44]对大豆成熟果荚的散粒程度的研究中发现了一个NAC基因SHAT1-5能够调控果荚次生细胞壁的木质化纤维覆盖细胞, 这个机制就是由SHAT1-5引起了在第16染色体的一段约116 kb的选择性清除的结果。目前, 从遗传变异角度探索SNAC基因功能的研究较少, 而这几项研究可以作为玉米自然遗传变异的前车之鉴。在我们的研究当中一共检测了12个SNAC基因的遗传变异, 在启动子区域(ATG前800 bp)检测到变异的有4个, 通过测序分析对比发现, 在ZmNAC080308启动子内产生大量的核苷酸多态性, 其中包括SNP和InDel, 虽然结合耐旱选择指数没有发掘出更多的耐旱功能型变异, 但不可否认这些变异极可能会使作物具有响应非生物胁迫的能力, 我们可以进一步研究其结构及对基因功能的影响, 这为玉米SNAC转录因子的遗传变异研究指引了新方向。
12个玉米SNAC基因与拟南芥及水稻中已知的SNAC基因具有相似的功能基序, 推测参与类似的耐逆功能代谢途径。4个玉米SNAC基因在上游800 bp检测到遗传变异, 序列含有3种逆境相关的顺式作用元件。其中有7个SNAC基因核苷酸多态性较高, ZmNAC080308基因的多态性达到0.00962, 说明这些基因在遗传漂移过程中受自然选择影响较大。ZmNAC070395的553 bp位点和ZmNAC080398的226 bp位点与耐旱选择指数极显著相关, 推测这两个核苷酸多态性位点对玉米耐旱性有着一定影响, 可以被考虑作为耐旱标记辅助选择。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|