基因表达调控是水稻化感作用形成的重要基础。本研究对化感水稻PI312777 ( Oryza sativa L.)的 OsMYB ( CT829537)基因分别进行过表达(overexpression, OE)和RNA干扰(RNAi), 再与稗草( Echinochloa crusgalli, BYG)共培养, 以野生型PI312777为对照。结果发现, 与稗草共培养下 CT829537-OEPI312777的酚类代谢关键酶基因表达上调, 根系及其水培液中的总酚酸浓度增加, 抑草能力增强; 相同处理下 CT829537-RNAiPI312777则相反, 其酚类代谢关键酶基因较野生型植株表达下调, 总酚酸浓度降低, 抑草能力下降。这表明 OsMYB ( CT829537)通过调节化感水稻的酚酸类物质合成进而影响其抑草能力。
Regulation of gene expression is a vital process in the formation of rice allelopathy. In this study, allelopathic rice PI312777 ( Oryza sativa L.) was modified by RNA interference (RNAi) and overexpression(OE) technologies to inhibit or enhance gene expression of OsMYB ( CT829537) in PI312777 respectively. The results showed that up-regulation of phenolic synthesis related genes was found in CT829537-OE transgenic PI312777 co-cultured with barnyardgrass (BYG), compared with that in wild type. However, the reverse was true in the CT829537-RNAi transgenic PI312777. Up-regulation of the gene expression in CT829537-OE transgenic PI312777 increased phenolic acids contents in rice root and root exudates, which led to enhance allelopathic inhibition on barnyardgrass. In contrast, decreases of phenolic acids contents and weed inhibition were found in CT829537-RNAi transgenic PI312777. These results implied that CT829537 is responsible for regulating phenolic synthesis in allelopathic rice PI312777 and then enhances allelopathic inhibition on weeds.
自20世纪80年代美国农业部的遗传学家Dilday发现了水稻化感抑草现象后, 水稻化感作用成为研究热点之一。化感水稻品种的筛选、化感物质的分离、鉴定及作用机制等是过去几十年水稻化感作用的主要研究内容。Olofsdotter等[1]、Chou等[2, 3]、Fujii等[4]和Dilday等[5, 6]在实验室和田间筛选到多种具有强化感潜力的水稻品种, 如PI312777等。水稻化感作用属数量遗传性状, 由多基因控制, 易受环境影响[6, 7, 8, 9]; Jensen等[10]对化感水稻的QTL定位研究发现有4个相关QTL分布在3条染色体上, 并解释了35%的表型变异。Ebana等[11]也鉴定到若干个化感作用QTL, 其中以位于第6染色体上的1个QTL贡献最大, 可解释16.1%的表型变异。Xiong等[12]研究了水稻化感作用的基因上位性效应及其与环境的互作, 发现第2和第5染色体上各有1个加性QTL, 可分别解释6.95%和4.35%的表型变异。Lee等[13]、Zeng等[14]也在这些方面作过相关研究, 其结果都表明水稻的化感作用潜力与环境因素关系密切, 易受环境影响。逆境条件下, 化感水稻中与次生物质合成相关的基因表达增强且抑草能力提高[6, 7, 8, 9, 15, 16, 17]。
水稻化感物质的分离与鉴定是水稻化感作用研究的重点与难点。当前, 酚酸类、萜类、黄酮类物质均表现出化感效应, 但尚未见它们在水稻根际中的浓度以及存在时间的报道[18, 19, 20, 21]。最近, 日本研究人员发现momilactone具有较强的抑草潜力, 认为它是一类主要的化感物质[22]。但未见这些物质在土壤中行为的报道。总之, 何类物质是水稻的主要化感物质迄今尚存在着争议。
尽管如此, 逆境胁迫引起的化感水稻中与次生物质合成、调控相关基因的增强表达已被多数研究所报道。Fang等[23, 24]利用抑制消减杂交技术分离了强化感水稻PI312777和非化感水稻Lemont在稗草共培下差异表达的基因, 发现化感水稻体内与化感物质合成、植物生长调控、胁迫防御、蛋白降解与修复、信号转导相关的五大类基因均增强表达, 其中一个与信号转导相关的MYB (登录号为CT829537)转录因子在稗草共培下的化感水稻中上调表达。转录因子(transcription factor, TF)也被称为反式作用因子, 它能够与真核生物基因的顺式作用元件发生特异性相互作用, 并与转录有激活或抑制作用的DNA形成结合蛋白。在植物的生长发育中, 转录因子在转录水平上的调节作用可使细胞内基因的表达呈现时间和空间差异, 并最终调控细胞分化。转录因子也是植物逆境信号转导的关键因子, 胁迫刺激其不断合成, 并传递和放大信号, 以调控下游基因的表达, 从而引起植物生理生化的改变。因此, 研究这些转录因子的生物学功能可为揭示植物响应逆境胁迫的基因表达调控过程提供重要基础。本研究采用RNAi和过表达(overexpression)技术分别抑制和增强化感水稻PI312777的CT829537表达, 进而比较这些水稻与野生型植株之间在酚酸类合成相关基因表达和酚酸类化合物含量以及抑草能力上的差异。
以化感水稻品种PI312777 (Oryza sativaL.)为供体材料[9], 稗草(Echinochloa crusgalli, BYG)为受体材料。
供试化感水稻PI312777种子经42℃烘干2 h, 蒸馏水浸泡1 d后进行催芽并播于秧盘。三叶期时移到装有Hoagland完全营养液的塑料盆中, 每周更换营养液。14 d后剪取水稻根系, 采用TRIzol试剂提取根部总RNA, 并参照TIANGEN Quant cDNA合成试剂盒合成cDNA。
分别以CT829537转录因子的CDS序列以及5′ 列端的DNA结合位点序列为模板设计该基因的过表达(overexpression)引物和RNAi引物(表1), 以化感水稻PI312777的cDNA为模板进行扩增。将PCR产物克隆到质粒中, 进一步通过酶切将目的片段连接到pTCK303载体上, 分别构建CT829537的RNAi和过表达载体。
| 表1 本研究用于构建载体的引物序列 Table 1 Sequences of primers used for vector construction in this study |
重组质粒用冻融法转化农杆菌EHA105, 获得CT829537过表达和RNAi转基因水稻[25, 26]。以野生型PI312777为对照, 分别对水稻新根进行GUS染色。与此同时, 分别利用GUS基因和潮霉素基因(Hpt)的特异引物(表1)对转基因水稻的GUS和Hpt基因进行扩增。
分别提取转基因和野生型PI312777根系RNA, 逆转录成cDNA, 利用CT829537转录因子的特异引物, 并以β -Actin作为内参基因, 进行半定量RT-PCR分析。
进一步以半定量RT-PCR检测结果为阳性的转基因PI312777根系的cDNA为模板, 通过荧光定量PCR (qPCR)比较PI312777根系中的7个与酚酸类化合物合成相关的酶基因在转基因水稻和野生型植株中的表达变化。这些基因包括苯丙氨酸解氨酶基因(PAL)、肉桂酸-4-羟化酶基因(C4H)、羟化酶基因(HYL)、辅酶A连接酶基因(4CL)、O-甲基转移酶基因(OMT)、肉桂酰辅酶A基因(CCA)和肉桂酸脱氢酶基因(CAD), 以β -Actin为内参基因, 各基因的qPCR引物见表2[27]。qPCR程序为95℃预变性10 min; 95℃变性30 s, X℃退火30 s, 68℃延伸20 s检测荧光强度, 41个循环; 95℃变性15 s, 60℃退火15 s升温10 min至95℃, 95℃变性15 s。根据所生成的阈值(Ct值), 运用2-Δ Δ Ct法计算基因的相对表达变化量[28]。
| 表2 酚酸合成相关基因的荧光定量PCR引物序列 Table 2 Primer sequence for the qPCR of phenolic acid relative genes |
取CT829537-RNAi、CT829537-OE转基因水稻及野生型水稻各3株(三叶一心期), 于Hoagland完全营养液中恢复培养7 d后, 分别与等株数的稗草共培养, 并以单独培养的各供试水稻作为对照, 处理与对照均设3次重复。培养7 d后, 参照Folin-Ciocalteu比色法测定转基因水稻及其野生型水稻根系及根系分泌物中总酚的含量[29]。
分别收集转基因PI312777及其野生型植株的水培液, 经0.45 µ m滤膜过滤后取5 mL加至垫有滤纸的培养皿中, 以添加等体积双蒸水为空白对照, 分别放入20粒预萌发的稗草种子, 处理与对照均设4次重复。常温培养10 d后, 收集各处理稗草, 120℃杀青30 min, 80℃烘干至恒重。所得原始数据均转化为抑制率(inhibitory rates, IRs), 作为化感潜力评价指标。IRs = (1-TR/CK)× 100%。其中TR为不同处理下的稗草干物质重, CK为对照稗草的干物质重。IR < 0表示存在促进作用, IR > 0表示存在抑制作用。
采用农杆菌介导法将CT829537-OE和CT829537- RNAi载体分别转化到化感水稻PI312777中, 获得CT829537-OE和CT829537-RNAi的T0代的转基因PI312777。转基因水稻经GUS染色检验显示水稻根部被染成蓝色, 而相同处理的野生型水稻根系颜色未发生变化(图1), 说明OsMYB (CT829537)基因的RNAi和OE表达质粒载体成功转入供试水稻中。进一步以T1代转基因水稻根部cDNA为模板, 进行潮霉素基因(Hpt)和GUS基因的PCR检测, 电泳结果显示RNAi与OE载体有效遗传至T1代(图2)。
CT829537-OE转基因PI312777的CT829537基因表达量明显高于野生型植株, 而该基因在CT829537-RNAi转基因PI312777中的表达量则较野生型植株明显降低(图3)。
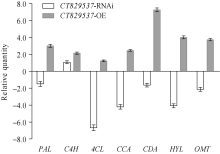
如图4所示, 与稗草共生7 d后, CT829537-OE转基因水稻中酚酸代谢途径7个关键酶基因的表达均高于对照。其中苯丙氨酸解氨酶基因(PAL)和羟化酶基因(HYL)上调了3倍和4倍, 肉桂酸-4-羟化酶基因(C4H)、辅酶A连接酶基因(4CL)、肉桂酰辅酶A基因(CCA)分别上调2.14、1.23和2.46倍, 肉桂酸脱氢酶基因(CDA)、O-甲基转移酶基因(OMT)基因表达量分别上调7.26倍和3.73倍。而在CT829537-RNAi转基因水稻中, 与对照植株相比, 除C4H表现微弱上调表达外, 其余6个基因表达均下调, 其中4CL、CCA、HYL下调了6.68、4.20和4.08倍, PAL、CDA、OMT分别下调1.50、1.64和2.14倍。稗草共生长处理后, 7个基因中只有C4H在RNA干扰和过表达转基因水稻中上调表达, 且在CT829537-OE转基因水稻中的表达量高于CT829537- RNAi转基因水稻。
与稗草共培养7 d后, 各水稻根系及其培养液中的总酚含量均高于相应的单一种植的对照水稻组(表3)。CT829537-RNAi的转基因水稻根部及其培养液中总酚含量均低于野生型水稻, CT829537-OE的转基因水稻则高于野生型水稻。通过检测3种水稻的根系和培养液中总酚含量显示, OsMYB (CT829537)能够调控化感水稻的总酚合成。
| 表3 稗草共培养处理对转基因水稻和野生型水稻的总酚含量影响 Table 3 Comparison of total phenol content between transgenic PI312777 and WT under rice/BYG co-cultured condition |
从图5可以看出, CT829537-OE转基因水稻根系分泌物对稗草干重的抑制率达36.7%, 显著高于野生型植株, 后者根系分泌物的抑草潜力为30.08%。而CT829537-RNAi转基因水稻的化感作用最弱, 仅为16.4%。
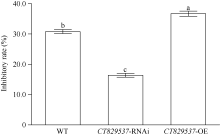 | 图5 转基因PI312777及野生型PI312777的根系分泌物对稗草干重的抑制率 缩写同图1。a、b、c表示不同处理之间存在显著差异(P< 0.05)。Fig. 5 Inhibitory rates of root exudates from transgenic PI312777and WT on dry weight of BYG Abbreviations are the same as those given in Fig. 1. Superscript letters indicate statistical groups that are significantly different (P< 0.05). |
已有研究表明, 逆境胁迫下, 水稻化感物质合成相关基因增强表达。Song等[30]应用抑制消减杂交技术(SSH)比较了低氮胁迫下化感水稻PI312777的基因表达变化, 发现参与酚酸类化感物质合成的PAL及O-甲基转移酶基因(OMT)均上调表达, 且其上调表达倍数高于相同处理下的非化感水稻Lemont, 同时PI312777也显示出更强的化感抑草潜力。Xiong等[31]利用荧光定量PCR系统比较了低氮胁迫下化感水稻PI312777和非化感水稻Lemont的7个酚酸合成相关基因的表达变化差异, 结果显示低氮胁迫下PI312777的7个酚酸类合成相关基因的上调倍数大部分高于Lemont, 且PI312777的酚酸含量也高于Lemont; 此外, 萜类合成相关基因在两种水稻中的表达差异不明显[32]。此外, 已有研究也表明, 田间旱育条件下, PI312777对田间杂草的抑制率高于土壤水分充足条件下的杂草抑制率, 并显著高于相同种植条件下非化感水稻Lemont的田间抑草率[33]。通过检测水稻根际土壤中的酚酸含量发现, 旱育条件下PI312777根际土壤中的酚酸类物质含量显著提高, 且明显高于Lemont的根际土壤, 表明了酚酸类化合物在水稻化感抑草过程中的重要作用[34]。
基因表达的转录受植物中最重要的一类调节基因(转录因子, TF)的调控。Junaedi等[35]发现了一些转录调控因子在稗草共培养下的化感水稻中增强表达。Fang等[23, 24]的研究结果也显示转录因子OsMYB (CT829537)在稗草共培养下的化感水稻PI312777中的表达增强。MYB转录因子是植物中最大的转录因子家族之一, 目前已有大量文献报道MYB转录因子生理作用非常广泛, 并参与植物次生代谢和生长发育的各个方面, 其中包括合成酚酸类物质、花色素苷、木质素、黄酮类的苯丙烷代谢途径[36, 37]。Moyano等[38]研究发现2个参与调控金鱼草苯丙烷代谢第一个酶PAL合成的转录因子AmMYB305与AmMYB340, 它们能够竞争性地结合在目的基因的启动子上, 并通过内部协调机制参与苯丙烷代谢。大豆中3个MYB基因ZmMYBZ2、ZmMYBJ6和ZmMYBJ7也均被证实可调控苯丙烷代谢途径部分关键酶的表达[39]。然而, MYB是否也调控水稻的化感作用, 尚未见相关报道。本研究结果显示化感水稻PI312777的OsMYB (CT829537)正调控其苯丙烷代谢途径中酚酸类合成相关基因的表达。过量表达PI312777中的CT829537基因后, 其酚酸类化合物合成相关的酶基因表达也增强, 酚酸类物质含量增加, 水稻化感抑草能力提高, 抑制PI312777中该基因的表达则呈现相反的趋势。该结果表明CT829537能够通过调控化感水稻的酚酸类代谢, 进而影响水稻的化感抑草能力。研究结果也初步揭示了水稻化感抑草作用的转录调控过程。
转录因子OsMYB (CT829537)正调控化感水稻PI312777的酚酸类物质代谢, 抑制PI312777的CT829537基因表达, 水稻的酚酸类合成相关基因表达下调, 酚酸类化感物质合成量减少, 抑草能力下降。过量表达该基因, 水稻的酚酸类合成相关基因表达增强, 酚酸类化感物质合成量增加, 抑草能力提高。然而, 这种调控作用究竟是通过转录因子与基因启动子直接互作实现的, 还是通过其他间接途径实现对基因表达的调控, 有待进一步研究阐明。
The authors have declared that no competing interests exist.
作者已声明无竞争性利益关系。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|