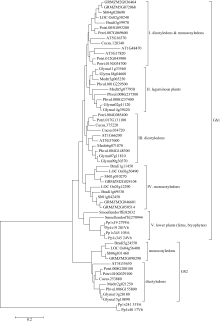
从Phytozome数据库中获得包括大豆在内的12种植物的谷氨酰胺合成酶(glutamine synthetase, GS)氨基酸序列, 利用MEGA5.10软件进行多序列比对、构建进化树。进化分析表明, 植物GS可以分成胞质型(GS1)和质体型(GS2)两大类, GS1可进一步分成分5个亚类, 包括双子叶植物为主的I、II和III亚类、低等植物类(IV)和单子叶植物类(V)。这5亚类中, 第II类是豆科植物特有的一类, 大豆的4个GS1 (GmGS1β1/2和GmGS1γ1/2)属于该亚类; 利用qPCR在大豆盛花期分析
The purpose of this study was to analyze the evolution, classification and tissue expression specificity of soybean cytosolic glutamine synthetase genes. Glutamine synthetase (GS) proteins from 12 species were downloaded from phytozome database, and used for phylogenetic analysis. The GSs were divided into two groups, cytosolic glutamine synthetase (GS1) and plastidic glutamine synthetase (GS2) based on the maximum likelihood of MEGA 5.10 software. The GS1 group was further divided into five subgroups, including three dicotyledons subgroups(I, II, and III), lower plants subgroup (IV) and monocotyledons subgroup (V). All GSs in subgroup II were mainly derived from leguminous plants including four GmGS1 proteins (GmGS1β1/2 and GmGS1γ1/2) in soybean. Real-time RT-PCR analysis of
氮是植物生长发育最主要的营养因子, 是构成生物体内核酸和蛋白的基本要素, 在农业生产中占据十分重要的地位。豆科植物通过共生固氮作用固定的氮素是自然和农田生态系统中氮素的重要来源。大豆是一种典型的根瘤固氮作物, 对氮素营养的需求量较高, 需氮总量是禾谷类作物的4~5倍。共生固氮对于满足大豆对氮素的高需求起到了至关重要的作用。据测定, 根瘤菌通过固氮作用每公顷可积累氮素45.0~52.5 kg, 相当于262.5 kg硫酸铵, 可满足大豆一半左右的需氮量。因此研究大豆根瘤氮素同化机制对于培育高产、优质、高效大豆新品种具有重要意义。
谷氨酰胺合成酶(glutamine synthetase, GS; EC 6.3.1.2)是植物N素同化途径中最为关键的催化酶之一, 被称为植物中无机态N转化为有机态N的“门户”, 对植物N素吸收、同化和利用效率有极重要的作用。GS可以催化NH4+和谷氨酸生成谷氨酰胺, 其中铵的来源可以是植物光呼吸或种子萌发过程中氨基酸的再氨基化所产生的次级氮, 也可以来自土壤或根瘤菌的固定[ 1, 2, 3]。高等植物中的GS全酶均为八聚体, 分子量为360 kD左右, 每个亚基分子量为38~45 kD, GS同工酶主要分为两类即胞质型GS (GS1)和质体型GS(GS2), GS1主要同化从土壤吸收的初级铵及再同化植物体内N循环途径释放的铵, 在根、茎、叶、根瘤等植物组织中广泛存在; GS2可以同化光呼吸过程所释放的氨, 主要存在于叶绿体等质体中[ 4]。Edwards等[ 5]对GS1和GS2的功能研究发现, 它们在植物体内起着非重叠作用, GS1主要同化植物根系统和维管组织中硝酸还原作用产生的氨, 负责对铵盐的积累、无机氮的吸收和循环; GS2与光合产物中氨的同化利用以及叶片衰老有关。Bernard等[ 6]研究表明植物 GS基因的表达受外界环境的影响, 是环境因子调节植物氮素积累和利用的重要分子靶点。目前, 关于植物 GS基因的命名尚没有统一标准, 拟南芥中胞质型GS可以写成 AtGLN1或 AtGSR1, 质体型 GS基因可以写成 AtGLN2或 AtGS2[ 2, 3], 玉米胞质型 GS多用 GLN表示[ 7], 根据3非翻译区的序列多样性, 大豆 GS1基因可以用α、β和γ表示, 即 GmGS1α、 GmGS1β和 GmGS1γ[ 8]。
农作物体内GS活性与许多经济性状有密切关系。王月福等[ 9]发现小麦开花后各器官的GS均具一定活性, 其中旗叶中GS活性最高, 后期旗叶和根系GS活性逐渐降低, 籽粒中GS活性先升高后降低, 与小麦籽粒的灌浆关系密切。Habash等[ 10]研究发现小麦GS1活性与籽粒和茎秆中的总氮量呈正相关; 在玉米第2染色体上存在同时控制细胞质GS活性和小穗数的QTL[ 11, 12, 13], 而水稻GS1活性与籽粒数目和籽粒大小显著相关[ 14, 15]。Li等[ 16]通过细致分析小麦 GS2基因单倍型的遗传效应进行了, 发现小麦不同染色体组上GS2单倍型与小麦氮素代谢及相关农艺性状显著关联。
虽然人们很早就已经明确GS是植物氮素同化代谢作用的关键限速酶, 但对于GS是否参与豆科植物的共生固氮作用还不清楚。直到2010年, Masalkar等[ 17]研究发现大豆GS1可以与位于根瘤共生膜上的主要蛋白NOD26的C端互作, 形成的蛋白复合体在植物细胞膜处快速将外界铵离子转化为中性分子谷氨酰胺, 这种发生在共生膜上的相互作用既可以避免过多铵离子进入细胞质对植物产生毒害, 也大大提高了根瘤共生固氮效率, 该研究为GS1参与大豆根瘤固氮作用提供了直接分子证据。在Masalkar等[ 17]研究的基础上, 本研究对大豆 GS基因家族的进化和分类进行研究, 并在大豆氮素代谢关键时期分析 GS1基因的组织表达特异性, 利用原核表达系统对主要在大豆根瘤中表达的 GS基因进行表达, 体外获得目标蛋白, 并分析其催化功能, 为大豆根瘤氮素同化代谢中分子机制的研究提供信息, 为大豆品种氮素代谢水平的遗传改良提供可调控位点。
大豆品种中品661来自中国农业科学院作物科学研究所, 2012年6月中旬播种, 常规田间管理。选择花期一致和生长状况相近的植株挂牌标记, 在大豆盛 花期取材, 选取大豆根、茎、叶、根瘤等组织, 快速清洗干净, 以锡箔纸包裹, 液氮速冷, -80℃保存备用。
从植物基因组数据库Phytozome中(http://phytozome.net/)下载12个物种共61条GS蛋白序列(入选蛋白都具有完整的GS beta-Grasp和GS catalytic功能域), 包括3种豆科植物(大豆、苜蓿和菜豆)、4种单子叶植物(水稻、玉米、高粱和二穗短柄草)、1个林木植物(杨树)、1种瓜果类植物(黄瓜)、2个低等植物(蕨类植物江南卷柏和藓类植物小立碗藓)和模式植物拟南芥。利用MEGA5.10[ 18]软件进行系统进化分析, 对聚类分析采用最大似然法(maximum likelihood), 氨基酸替换模型采用p-distance法。利用Z-test程序中的Kimura 2-para模型计算大豆 GS基因家族碱基替换速率。
采用TRIzol法从中品661根、茎、叶、根瘤等组织中提取RNA, 反转录成cDNA后-20℃保存备用。根据大豆6个 GS1基因和大豆 ACT11基因(内参)编码区序列利用Primer Premer 6.0设计荧光定量PCR引物(表1)。引物由上海生物工程技术服务有限公司合成。采用ABI SybrGreen PCR Master Mix(2X)试剂盒, 参照其说明书, 在ABI Stepone Plus型荧光定量PCR仪上进行荧光定量。采用2-∆∆Ct方法计算基因的相对表达量, 3次生物学重复取平均值。
| 表1 大豆 GmGS1基因qPCR引物序列 Table 1 qPCR primer sequences for GmGS1 genes |
根据大豆 GmGS1β2基因全长设计引物, 其上游引物GSF为5′-ATGTCGCTGCTCTCAGATCTC-3′, 下游引物GSR为5′-TCATGGCTTCCACAGAATGGTT-3′。
引物由上海生物工程技术服务有限公司合成。以中品661根瘤cDNA为模板利用GSF/GSR为引物进行PCR扩增。PCR反应体系20 μL, 含模板cDNA 40 ng、dNTPs 200 μmol L-1、10×buffer 2 μL、引物各2 μmol L-1、Ex Taq酶1 U。采用降落PCR扩增程序, 94℃预变性5 min; 94℃变性1 min, 62℃退火45 s, 72℃延伸50 s, 35个循环(每个循环退火温度下降0.3℃); 72℃延伸10 min。PCR扩增产物经1%琼脂糖凝胶电泳检测后, 回收目标片段进行TA克隆。提取阳性克隆质粒并进行酶切鉴定, 送上海生工生物工程技术服务有限公司测序。利用DNAMAN软件将碱基序列翻译成氨基酸序列, 利用ExPASy数据库在线程序Protparam和Scanprosit分析基因编码蛋白的理化特性。
根据目的基因的完整编码区, 设计1对用于构建原核表达载体的引物, 并在引物5端添加酶切位点, 引物序列为GS4T-F: 5′-CCCGGGTATGTCGCT GCTCTCAGATCTC-3′, GS4T-R: 5′-CTCGAGTCAT GGCTTCCACAGAATGGTT-3′(下画线处分别表示 SmaI和 XhoI酶切位点)。用GS4TF/GS4TR对中品661根瘤cDNA进行PCR扩增。反应体系和程序同1.3。利用 SmaI和 XhoI对经测序验证正确的pMD18-T- GmGS1β2和pGEX4T-1载体质粒分别进行双酶切, 纯化目标片段, T4连接酶连接。利用 Sma I和 Xho I对重组子进行双酶切鉴定, 并对重组质粒进行测序验证。重组质粒插入片段序列正确, 并且目的基因的阅读框与原核表达载体上的GST-Tag阅读框正确融合, 表明pGEX4T-1-GmGS1β2原核表达载体构建成功。
将重组质粒pGEX4T-1:GmGS1β2转化至宿主表达菌株BL21(DE3)中, 挑取重组单菌落振荡过夜培养, 培养物按1∶100接种于选择性LB液体培养基, 待培养至OD600≈ 0.6时, 加入IPTG(终浓度为0.5 mmol L-1), 30℃诱导表达融合蛋白。诱导5~7 h后4℃ 900× g离心20 min, 弃上清液, 用1×PBS悬浮沉淀, 4℃ 900× g离心15 min, 弃上清液, 再用1×PBS洗涤一次沉淀。用超声波破碎洗涤后的沉淀, 用4B-Beads蛋白纯化试剂盒(GE公司)纯化重组蛋白, 具体操作步骤参考说明书。纯化后的重组蛋白GST:GmGS1β2经SDS-PAGE (4%浓缩胶和12%分离胶)电泳检测, 考马斯亮蓝染色。
采用谷氨酰胺合成酶测定试剂盒(南京建成生化试剂有限公司), 以分光光度计法对纯化后的GmGS1β2重组蛋白GS活性进行分析。利用Bio-Rad蛋白定量试剂盒(5000112)对目标蛋白定量, 具体操作参考试剂盒说明书。
利用最大似然法对12种植物的61个GS聚类并构建进化树, 由图1可以看出除了2个小立碗藓GS (pp1s241 33V6和pp1s40 17V6)由于序列差异过大被聚在树外, 大部分植物GS可以分成胞质型GS (GS1)和质体型GS (GS2) 2大类, 每一类又可分为若干亚类, 其中GS1可以分为5个亚类, 包括主要由双子叶植物组成的I、II和III亚类, 其中第II亚类由豆科植物GS组成; 第IV亚类由单子叶植物组成, 第V亚类由低等植物(小立碗藓和江南卷柏)组成; GS2可以分为单子叶和双子叶两亚类。由此看出, 植物GS进化与植物的进化史相似, 每一类植物(低等植物、单子叶植物和双子叶植物)都有其特有的GS类型。由于GS1中也存在一个由单双子叶组成的混合类型(I), 说明单、双子叶GS1进化过程中也发生了趋同进化。2个低等植物(江南卷柏、小立碗藓)基因组中只有GS1没有GS2, 暗示GS2可能是从GS1演化而来。
大豆基因组中共有8个GS, 包含6个GS1和2个GS2, 每个GS都有2个拷贝, 说明大豆 GS基因在进化过程中经历了基因组复制事件。从聚类结果看, 大豆第2、第11、第14和第18染色体上的4个GS1距离较近, 与第7和第9染色体上的2个GS1距离较远, 并与豆科植物特有GS1同聚为一类(II), 暗示这4个基因可能与大豆共生固氮有关。
由图2可以看出大豆GS氨基酸序列的相似性(similarity)很高, 2个GmGS2的氨基酸序列相似性高达99.1%, 仅有4个氨基酸差异: 3个位于N端的信号肽内(23位T变成S, 52位L变为M, 60位T变成I), 1个位于GS的酶功能区内(beta-Grasp domain, 106位氨基酸D变为H)。这种序列高度相似性也存在于GmGS1s之间, GmGS1s氨基酸序列的平均相似性高达95.04%, 重复基因间的相似性均超过97%, 其中GmGSα1与GmGSα2氨基酸序列的相似性最高为98.9%, GmGS1β1与GmGS1β2的相似性为97.5%, GmGS1γ1与GmGS1γ2的相似性为98.3%, 序列上的高度相似性暗示进化产生的重复基因在功能上可能是重叠的。此外, 在氨基酸长度上, GS1和GS2存在明显差异, GS2的N端和C端分别比GS1多60个和16个氨基酸, N端60个氨基酸是GS2在质体等亚细胞器内定位所必须的信号肽, C端的16个氨基酸是否也与GS2在质体中的功能有关还不清楚。
碱基替换速率分析表明, GmGS1家族成员的同义替换速率( dS)大于非同义替换速率( dN)( dS- dN= 12.46, P<0.01), 说明 GmGS1s基因在进化过程中受到了纯化选择作用, 不利变异在进化时逐渐被淘汰。
由于GS2主要在叶绿体等质体中表达, GS1主要负责对土壤或根瘤共生固氮中氮素的同化利用[ 4], 本研究对6个大豆 GS1基因的组织表达特异性进行分析, 目的是发掘出在大豆根瘤氮素同化代谢中起关键作用的 GS基因。由图3可以看出, 4个豆科植物特有 GS1基因( GmGS1β1/2和 GmGS1γ1/2)在大豆根瘤中的相对表达量较高。6个 GmGS1基因的组织表达特点与基因的分类相似, 大体上也可以分为3类, 2个 GmGS1αs基因在各个组织中的表达量均较低; 2个 GmGS1γs基因特异地在大豆根瘤中表达; 2个 GmGS1βs基因在大豆各个组织中均高量表达, 表达丰度显著高于 GmGS1α和 GmGS1γ (图3), 但二者的 组织表达特异性存在一定差异, GmGS1β1基因主要在叶片中表达, GmGS1β2基因在根瘤中大量表达。
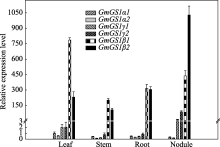 | 图3 大豆 GS1基因的组织表达特异性分析Fig. 3 Tissue-specific expression of GmGS1genes from soybean plants during flowering stage |
组织表达特异性的差别预示二者在功能上可能存在分化, GmGS1β2基因可能在大豆根瘤氮素同化代谢中有更重要作用。
利用RT-PCR从大豆品种中品661发育的根瘤cDNA中克隆出 GmGS1β2基因, 测序结果表明该基因CDS全长1071 bp, 编码356个氨基酸, 基因编码蛋白具有2个功能域(GS beta-Grasp和GS catalytic), 其中Beta-Grasp功能域由79个氨基酸组成(19~97氨基酸), Catalytic功能域由235个氨基酸组成(104~348氨基酸), 其中catalytic功能域内存在一个潜在的ATP结合位点(237~255氨基酸)。利用ExPASy数据库的在线分析程序Protparam和Scanprosit分析GmGS1β2蛋白的理化性质表明, GmGS1β2预测分子量为39.09 kD, 理论等电点为5.48, 原子组成为C1743H2679N479O526S10, C、H、N、O、S所占比例分别为32.06%、49.27%、8.81%、9.67%和0.18%。该蛋白稳定系数(instability index)为39.66, 是一个稳定蛋白(稳定系数>40为不稳定蛋白); 脂肪系数(aliphatic index)为75.90, 总平均亲水性(grand average of hydropathicity, GRAVY)为-0.414, 表明该蛋白是1个疏水蛋白。糖基化和磷酸化位点分析表明, 该蛋白具有3个潜在的 O-糖基化位点( O-glyco sylation), 磷酸化位点多达21个, 其中丝氨酸磷酸化位点11个, 苏氨酸磷酸化位点4个, 络氨酸磷酸化位点6个, 推测翻译后修饰可能是GmGS1β2的重要调控方式之一。
将重组质粒pGEX4T-1-GmGS1β2转化大肠杆菌BL21 (DE3)感受态细胞, 加入不同浓度的IPTG (0.2、0.5、1.0和1.5 mmol L-1), 在16℃、30℃和37℃下分别以不同时间诱导后, 提取大肠杆菌总蛋白经SDS-PAGE分析, 表明插入有外源片段的重组质粒经0.5 mmol L-1IPTG 30℃诱导5 h后重组蛋白表达效果较好。
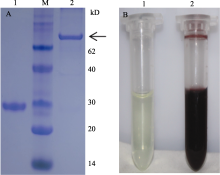
将诱导表达的菌体经超声波破碎后, 利用Glutathione sepharose 4B纯化目的蛋白。由图4-A可以看出, 纯化后的蛋白条带单一, 在蛋白分子量67 kD左右有1条蛋白条带, 而空载体转化子未出现这条蛋白带。除去GST-tag标签蛋白28 kD, 目的蛋白的分子量约为39 kD, 这与外源大豆谷氨酰胺合成酶基因 GmGS1β2编码区蛋白的分子量理论值相符, 表明重组质粒pGEX4T-1-GmGS1β2在大肠杆菌中诱导表达出了GmGS1β2蛋白。
对纯化后的GmGS1β2重组蛋白的GS活性进行分析, 由图4可以看出, 重组蛋白可以与底物发生催化反应生成红褐色产物(图4-B2), 而GST标签蛋白不能与底物发生反应(图4-B1), 说明重组蛋白具有GS催化活性。利用试剂盒进一步对重组蛋白的GS活性定量分析表明, GmGS1β2蛋白的GS活性为3028.27 U mg-1, 而标签蛋白GST未检测出GS活性, 说明GmGS1β2蛋白是一个有活性的功能蛋白, 在大豆根瘤谷氨酰胺的合成中具有重要作用。
在距今5900万年和1300万年前, 大豆基因组发生2次大规模复制, 大量的染色体重排, 产生了一个高度重复的基因组, 约75%的基因以多拷贝形式存在[ 19]。大豆 GS基因也是一个多拷贝基因家族, 从大豆GS基因的进化和分类的结果可以看出, 8个 GS1基因可以分为4类, 每一类基因都有2个拷贝, 表明大豆 GS基因由于基因组的大规模复制产生了重复基因。从氨基酸多序列比对结果可以看出, 大豆 GS重复基因间的序列具有很高的相似性(图2), 暗示 GS重复基因之间的功能可能是冗余的。从进化关系来看, 大豆 GS基因与同属豆科植物的苜蓿 GS基因亲缘关系最近, 苜蓿 GS基因也可以分为相同的4类, 但每类 GS基因只有1个拷贝, 说明苜蓿基因组可能没有经历过类似的大规模复制, 可以利用苜蓿GS基因是单拷贝的这一特性对豆科植物 GS基因家族功能进行研究。
大豆盛花期根瘤菌活动和繁殖最旺盛, 也是大豆固氮能力最强的时候, 我们在大豆盛花期对大豆 GS基因的组织表达特异性进行了分析, 目的是筛选出大豆根瘤氮素同化代谢关键 GS基因, 对重复基因之间的功能分化进行分析。从基因的表达丰度上看, 2个 GmGSαs基因在大豆各个组织中的表达量较低, 而4个豆科植物特有 GS1基因(subgroup II, GmGSβs和 GmGSγs)在大豆根瘤中的高量表达, 表明 GmGSβs和 GmGSγs基因在大豆根瘤氮素同化代谢中具有重要的作用。虽然 GmGS1β1和 GmGS1β2在大豆各个组织中的表达丰度都很高, 但基因主要表达部位不同, GmGS1β2基因在大豆根瘤中的表达量最高, 体外表达试验证明了该基因是有功能的, 暗示 GmGS1β2基因在大豆根瘤固氮方面可能发挥主要作用。
基因重复是生物遗传系统分化的重要推动力量[ 20], 基因复制后其中一个拷贝会保持原来的功能, 而另外一个拷贝可以不再受遗传选择的约束, 随机积累突变, 为产生新基因或新功能的产生提供了可能[ 21]。基因重复后2个拷贝在表达水平上的分化是重复基因功能分化的重要一步[ 20]。 GmGSαs和 GmGSγs的重复基因间有相似的组织表达部位和丰度, 说明这2类重复基因在进化过程中发生了趋同进化, 功能上可能是重叠的; 2个 GmGS1βs重复基因的在茎和根中的表达丰度相近, 但在叶片和根瘤中存在明显差异, GmGS1β2基因主要在根瘤中表达, GmGS1β1基因主要在大豆叶片中高量表达, 说明在进化过程中 GmGS1βs重复基因的功能发生了一定分化, GmGS1β1基因可能也同时参与了大豆叶绿体组织中光合产物的氮素同化代谢, 这与我们传统上认为GS1主要负责同化土壤中的氮素的观点有所不同[ 2, 5]。从蛋白结构上看, GmGS1β1蛋白的N端没有类似于GmGS2的信号肽, 因此GmGS1β1可能是通过一种新的途径参与大豆叶片中氮素的同化代谢。
大豆 GS1基因表达存在多种调控方式[ 22], 其中非翻译区(UTR)对基因的表达有较大影响。Ortega等[ 23]研究发现大豆 GS1基因3UTR可以通过影响转录本的稳定性, 抑制目的基因的表达; GmGS1β1基因5UTR可以显著增强目的基因的表达, 在转基因瞬时表达的烟草叶片中带有 GmGS1β1基因5UTR的报告基因的表达量是对照的20倍[ 24], 说明 GS1基因3UTR可能是基因表达的抑制子, 而5UTR则扮演着基因表达增强子的角色。对 GmGS1β1和 GmGS1β2两个基因启动子的序列比对发现, 二者的相似性超过45%, 暗示它们可能存在相似的调控原件。Morey等[ 6]和Miao等[ 25]研究表明 GmGS1β1和 GmGS1β2基因可以受外源NH3诱导表达。Marsolier等[ 26]通过启动子删除试验发现 GmGS1β1基因启动子-1.3 ~ -3.5 kb处存在受NH3诱导的作用区域, 但没能从 GmGS1β1基因启动子中鉴定出受NH3调控的顺式作用原件。
目前, 本项目组正在对 GmGS1β2基因启动子上组织特异表达及对外界环境氮素的响应的关键位点、基因的遗传多样性和生物学功能进行研究, 以阐明 GS参与大豆根瘤氮素同化代谢的机制, 为大豆氮素同化代谢水平的遗传改良提供依据。
大豆 GmGS1s重复基因间具有相似的表达模式, 大豆根瘤中存在4个豆科植物特有 GS1基因; 这4个基因中, 2个 GmGS1βs基因主要在大豆根瘤中表达, 且以 GmGS1β2基因表达丰度最高, 该基因编码蛋白具有谷氨酰胺合成酶活性, 参与大豆根瘤的氮素同化代谢, 可能在固氮中发挥主要作用, 是大豆氮素代谢遗传改良的关键调控位点。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|