蔗糖非酵解型蛋白激酶(SnRK)是植物体内ABA信号转导途径的关键调控酶, 在植物的抗逆境生长过程中发挥着重要的作用。本研究利用RT-PCR和RACE-PCR技术, 克隆出编码甘蔗SnRK2蛋白的基因
Sucrose non-fermenting 1-related protein kinase (SnRK) is a key enzyme in the ABA signal transduction pathways, which plays an important role in plant development under adverse environments. Reverse transcription-polymerase chain reaction (RT-PCR) and rapid amplification of cDNA ends (RACE) were applied to clone
甘蔗( Saccharum officinarumL .)是典型的亚热带作物。随着全球气候的多变性, 近年来干旱、低温、盐碱和氧化等非生物胁迫因子的影响已成为制约甘蔗产量和品质的关键因素。植物应答逆境胁迫的过程是一个涉及多基因、多信号转导和多基因表达产物的复杂过程[ 1], 其中蛋白质的可逆磷酸化是植物受渗透逆境诱导的主要信号转导机制之一。在蛋白质的磷酸化过程中, 蛋白激酶对植物的生长发育起着重要的调控作用。
蔗糖非酵解型蛋白激酶(sucrose non-fermenting 1-related protein kinase, SnRK)广泛存在于植物中, 是一类丝氨酸/苏氨酸蛋白激酶(serine/threonine protein kinase), 参与植物体内多种信号途径转导中的磷酸化。根据基因结构及蛋白序列同源性分析[ 2], 植物中SnRK相关蛋白激酶基因有SnRK1、SnRK2和 SnRK3共3个家族, 其中SnRK2和 SnRK3是植物特有的蛋白激酶。SnRK2家族基因在蛋白结构上均含有非常保守的Ser/Thr酶活催化结构域DFGY SKSSVLHSQPKSTVGTPAYIAPE[ 3]和ATP位点GXGXXGX, 且其C端序列具有较高的多态性[ 4, 5]。
通过对ABA信号转导通路中受体蛋白PYR/ PYL/RCAR、蛋白激酶PP2Cs及蛋白磷酸激酶SnRK2s的不断研究发现[ 6, 7, 8], 当植物细胞中不存在ABA时, 受体蛋白PYR/PYL/RCAR不能与PP2Cs相互作用, PP2Cs处于高活性状态并抑制其下游的SnRK2s的活性; 当植物受到外界的刺激或自身发育产生ABA后, 细胞中的ABA会与受体蛋白PYR/ PYL/RCAR结合, 使其构型发生变化进而使SnRK2s的活性得以释放, 处于激活状态的SnRK2s就可以通过磷酸化作用激活下游的转录因子, 正向调控ABA信号应答基因的表达[ 9, 10, 11]。
大量研究证明, 编码SnRK2蛋白激酶的基因是参与植物逆境胁迫以及糖代谢调控的一类家族基因[ 12, 13, 14], 主要受水分胁迫和ABA诱导[ 15, 16, 17]。在拟南芥中 SnRK2.2、 SnRK2.3和 SnRK2.6基因可以通过磷酸化激活bZIP类转录因子, 使其增强与ABA应答转录因子(ABA responsive transcription factors, ABF/AREB)的结合从而参与干旱和ABA的诱导过程[ 18, 12], Umezawa等[ 19]证实 SnRK2.8可以被渗透胁迫激活, 并且过量表达该基因的拟南芥抗旱性明显增强。小麦 TaSnRK2.8 参与干旱、高盐、低温及ABA等信号转导的过程, 其过量表达可引起转基因植株根部组织含水量、细胞膜稳定性、渗透势等抗逆境生理的一系列变化[ 20]。此外, 有研究发现, 大豆中的SnRK2成员 SPK1和 SPK2可以被高渗透逆境诱导激活[ 21], 而 SPK3和 SPK4可被脱水和高盐激活[ 22]。由此说明SnRK2家族基因与植物的抗逆性有密切的相关性。
近年来, 已有报道在多种植物中克隆得到SnRK2家族的相关基因, 但迄今未见甘蔗中ABA信
号转导途径中关键基因 SnRK2克隆及功能分析报道。本课题组前期应用基因芯片技术、RT-PCR和cDNA末端快速扩增技术 (rapid amplification of cDNA ends, RACE) 等方法筛选和克隆获得 SoSnRK2.1基因的cDNA全长, 同时构建原核表达载体pET-SoSnRK2.1, 并在大肠杆菌中进行IPTG的诱导表达。应用实时荧光定量技术对 SoSnRK2.1基因在ABA胁迫和PEG+ABA胁迫下的表达模式进行了研究, 以期为进一步研究该酶在甘蔗中的调控机制提供依据。
选用甘蔗品种桂糖21 (GT21), 取生长健壮、大小一致的蔗茎在沙床中培育, 待苗长至三至四叶期后, 一部分移栽至桶中水培; 一部分移栽至桶中土培。当苗长至五至六叶期时选取长势一致时进行处理, 水培处理如下: ABA胁迫(ABA stress, AS), 以叶面喷施100 μmol L-1 ABA; 干旱+ABA胁迫(water stress + AS, WAS), 于营养液中加入25% ( W/V)的PEG-6000及叶面喷施100 μmol L-1 ABA。土培处理如下: H2O2胁迫(H2O2stress, HS), 以叶面喷施 10 mmol L-1 H2O2; 低温胁迫(cold stress, CS), 于4℃低温冷库中处理, 干旱胁迫(PEG stress, PS)和NaCl胁迫(NaCl stress, NS)分别使用PEG-600015% ( W/V)、NaCl (100 mmol L-1)做浇淋根部处理。在水培处理后的0、3、6、9、12和24 h采集叶样(+1叶)和幼嫩根系, 在土培处理后的0、6、12 、24、48和72 h采集叶样(+1叶), 分装速冻于液氮中, 并于-80℃冰箱保存备用。
采用TRIzol试剂(Invitrogen) 提取甘蔗总RNA, 参照购自Clontech (634923)和Formentas (K1622)公司逆转录试剂盒说明合成cDNA第一链, 产物分别用于基因5′-RACE、3′-RACE和全长的克隆; 实时荧光定量PCR的cDNA第一链的合成则按照TaKaRa (SYBR Premix Ex TaqII)的说明书操作。
根据基因芯片中得到的EST片段序列设计3′-RACE和5′-RACE特异引物扩增该基因全长cDNA, 引物序列为: 3′-RACE: 5′-ACAGCAGGGCGATTCAG TG; 5′-RACE: 5′-TTCACGCGAGGTGTTGGA。3′-RACE- PCR结合接头引物18AP (5′-GGCCACGCGTCGA CTAGTAC-3′), 及 5′-RACE-PCR结合Clontech Race试剂盒提供的接头引物UPM (Universal Primer A Mix), 分别进行 SoSnRK2.1的3′和5′末端扩增, 反应体系含: 2× GoldStar Best MasterMix 25 μL、cDNA 2 μL、10 μmol L-1 Forward和Reverse引物各1 μL, 用ddH2O补齐至50 μL。扩增程序为95℃预变性10 min; 95℃变性30 s, 60℃退火30 s, 72℃延伸2 min, 36个循环; 72℃最后延伸10 min。PCR产物经1.5% 的琼脂凝胶电泳检测, 回收纯化目的条带, 连接pMDl8-T载体, 热激转化DH5α 感受态细胞, 通过蓝白斑筛选挑取白色菌落, 经PCR验证后挑选几个阳性克隆送生工生物工程(上海)公司测序, 3′-RACE和5′-RACE产物共有149 bp的重叠序列, 用于后续全长拼接。
用BioXM2.6预测基因氨基酸序列; ExPASy (http://expasy.org/tools/)分析SoSnRK2.1蛋白质的基本理化性质; 由ScanProsite (http://prosite.expasy. org/scanprosite/)预测蛋白质保守域; 由Protscale (http://web.expasy.org/protscale/)预测蛋白质的疏水性; 由NetPhos 2.0 Server (http://www.cbs.dtu.dk/ services/NetPhos/)预测蛋白质磷酸化位点; 用MEGA 5.0程序(http://www.megasoftware.net/)进行系统进化树分析。
以pET-30a(+) 作为表达载体, 根据 SoSnRK2.1基因的ORF (open reading frame)设计 SoSnRK2.1基因原核表达载体构建的引物, pETSn-F: 5'-CGG GGTACCATGGAGGAGAGGTACGAGGC-3'和pETSn-R: 5'-CCGCTCGAGTCAGTAGGTGTCATCAGCGTCT-3' (斜体加下画线部分分别为N端 Acc65 I和C端 Xho I)进行PCR扩增。PCR体系参见步骤1.3, PCR程序为95℃预变性10 min; 95℃变性50 s, 66℃退火50 s, 72℃延伸90 s, 共36个循环; 72℃延伸10 min。PCR产物经1.5% 的琼脂凝胶电泳检测回收纯化后连接pMDl8-T载体, 热激转化DH5α感受态细胞, 通过蓝白斑筛选挑取白色菌落, 经PCR验证后挑选几个阳性克隆送生工生物工程(上海)公司测序。选择测序正确的菌液提取大量质粒, 然后与 pET30a(+)载体质粒分别同时用 Acc65I和 XhoI双酶切, 回收片段、连接和转化。菌液检测和双酶切鉴定阳性单克隆, 获得重组质粒pET-SoSnRK2.1, 酶切鉴定正确的重组质粒需经测序验证后方可用于后续试验。
将构建好的重组质粒pET-SoSnRK2.1与空载体pET30a(+)质粒分别转化至表达菌株大肠杆菌BL21(DE3), 经Kan抗性筛选, 提取重组质粒进行双酶切鉴定和测序。将转化后的重组菌株接种至LB液体培养基中, 37℃培养振荡至OD值约0.4~0.6, 以终浓度为1.0 mmol L-1的IPTG在37℃条件下分别诱导表达2、4、6和8 h, 以不加IPTG诱导的pET-SoSnRK2.1和pET30a(+)空载体作为对照。诱导完成后以6000× g离心, 收集沉淀加150 μL 3× SDS-PAGE缓冲液裂解变性, 沸水浴10 min, 冰上冷却后取15 μL进行SDS-PAGE电泳分析(SDS- PAGE浓缩胶浓度为4%, 分离胶浓度为12%)。
根据获得的 SoSnRK2.1基因全长序列设计荧光定量PCR的特异性引物RT-SnF: 5′-GGTTTGTTTA ACACCCACAC-3′, RT-SnR: 5′-ACCTGGACTCATC TTCACTG-3′; 以已发表甘蔗看家基因 GAPDH(EF 189713)为内参[ 23], 设计其引物序列为GAPDH-F: 5′- TGGTGCTGACTATGTCGTGGA-3′, GAPDH-R: 5′- CATGGGTGCATCTTTGCTTG-3′。反应体系20 μL 中含10 μL 2×Premix Ex Taq II; 2 μL cDNA; 4 μmol L-1的正反向引物各1.0 μL、6.0 μL双蒸水; 扩增程序为95℃预变性10 min; 95℃变性10 s, 60℃退火20 s, 72℃延伸20 s, 共40个循环。反应完成后进行溶解曲线检验, 采用2-ΔΔCt法计算基因相对表达量。
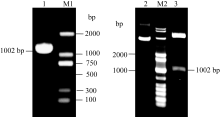
以逆转录合成的cDNA为模板, 经3′-RACE- PCR和5′-RACE-PCR分别扩增得到1007 bp (图1-A)和600 bp (图1-B)片段, 对所得片段回收、连接、克隆测序, 除去接头引物及重叠序列, 最后拼接得到 SoSnRK2.1基因全长cDNA序列为1385 bp, 基因登录号为JQ292841。序列分析显示5′端已经包含起始密码子且符合Kozak法则, 3′端有典型的poly (A)加尾信号, 包含完整的开放阅读框(ORF), 长度为1002 bp, 编码333个氨基酸, 5′端非编码区长125 bp, 起始密码子ATG位于序列126 bp处, 3′端非编码区长258 bp (图2)。
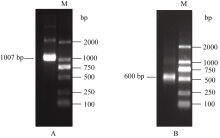 | 图1 SoSnRK2.1基因的扩增结果A: 3′-RACE产物; B: 5′-RACE产物。Fig. 1 Amplified products of SoSnRK2.1 geneA: 3′-RACE amplified results; B: 5′-RACE amplified results. |
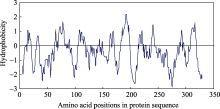
利用Expasy工具分析显示SoSnRK2.1编码蛋白等电点为5.54, 分子量为37.8 kD, 平均亲水系数(GRAVY)为-0.437。ScanProsite蛋白预测结果显示其在第5~261位氨基酸之间存在蛋白激酶保守催化域(图3), 其中第11~19位和第34位氨基酸为ATP结合位点, 第120~132位氨基酸为丝氨酸/苏氨酸蛋白激酶活化位点, 第124位氨基酸为质子受体位点。利用Protscale对SoSnRK2.1蛋白的疏水性预测发现, 蛋白质氨基酸链上第192位络氨酸(Tyr-192)、第193位缬氨酸(Val-193)疏水性最强, 分值为2.189; 分值最低的是第275位丙氨酸(Ala-275), 为-2.867。该蛋白的亲水区域多于疏水区域, 为亲水蛋白质(图4)。NetPhos 2.0 Server 分析结果表明, SoSnRK2.1蛋白有11处潜在丝氨酸磷酸化位点, 分别为第97、第101、第135、第159、第173、第231、第232、第247、第306、第322位氨基酸; 3处潜在苏氨酸磷酸化位点, 分别为第137、第213、第301位氨基酸; 8处潜在络氨酸磷酸化位点, 分别为第5、第35、第103、第147、第166、第227、第277、第333位氨基酸。
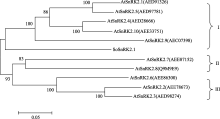
将获得的 SoSnRK2.1开放阅读框通过NCBI BlastX比对表明, 其与玉米(NP_001136496)、水稻(BAD17999)、短柄草(XP_003574349)、葡萄(XP_ 002262726)、大豆(XP_003549953)和高粱(XP_ 002461041)的一致性/相似性依次为93%/98%、93%/ 92%、91%/86%、92%/84%、91%/82%和90%/77%, 说明 SoSnRK2.1与其他作物SnRK2基因进化关系较近。通过与拟南芥SnRK2家族10个基因的氨基酸聚类分析发现(图5), SoSnRK2.1与拟南芥SnRK2.1、SnRK2.4、SnRK2.5、SnRK2.9和SnRK2.10先聚在一起, 同属于拟南芥SnRK2家族第一亚家族。
使用引物pETSn-F和pETSn-R进行扩增, 以逆转录成cDNA为模板, 扩增得到约为1002 bp的条带(图6-A)。对目的条带回收纯化, 连接PMD18-T后测序结果表明, 该产物为 SoSnRK2.1基因ORF, 长为1002 bp。重组质粒 pET-SoSnRK2.1双酶切结果(图6-B)显示, 可切出约1002 bp 左右片段, 所获片段大小与预期结果相符, 表明 SoSnRK2.1基因ORF已成功插入载体质粒中。将阳性克隆测序, 结果与获得的基因比对完全一致, 进一步证明 pET- SoSnRK2.1重组质粒构建正确, 表明 SoSnRK2.1基因的原核表达载体构建成功, 可进行下一步的表达分析。
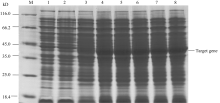
如图7所示, 经IPTG和不同时间诱导后均在38 kD 处出现pET-SoSnRK2.1融合蛋白, 与蛋白理论分子量37.8 kD结果一致, 而对照没有融合蛋白的表达。这说明 SoSnRK2.1基因在原核生物大肠杆菌中已被成功表达。
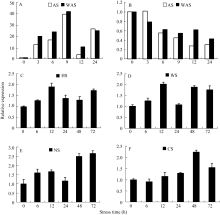
应用实时荧光定量PCR技术, 以甘蔗 GAPDH基因作为内参, 分析 SoSnRK2.1基因在不同栽培技术不同外源胁迫下不同时间内的表达量。由图8可知, SoSnRK2.1基因在甘蔗叶片和根系中的表达模式明显不同, 且水培条件下的表达量远远高于土培条件下的表达量。随着处理时间的延伸, 根系中 SoSnRK2.1基因在AS和WAS处理下均呈抑制表达(图8-B), 但在WAS处理下表达量的下降趋势明显慢于AS处理。在无论水培或土培条件下不同胁迫处理的叶片中, SoSnRK2.1基因均呈诱导表达, 但表达量存在明显差异。在水培条件AS和WAS处理下(图8-A), 甘蔗叶片中 SoSnRK2.1基因的表达量均显著提高, 且在9 h 达到最大值, 约为对照表达量的40倍, 随后出现表达低谷, 24 h 后表达量又出现回升。在土培条件下各个处理中 SoSnRK2.1基因表达量明显低于水培条件, 在HS处理中(图8-C), SoSnRK2.1基因表达量12 h达到最高值, 为对照的1.93倍, 随后表达量有些回落, 到72 h再次上升达到较高值, 为对照的1.78倍。在WS处理中(图8-D), SoSnRK2.1基因表达量在12 h出现第一次的高值, 为对照的2.00倍, 到24 h有小幅度的下降, 出现整个处理期内的最低值, 随后又回升, 48 h和72 h表达量均达到较高值, 分别为对照的1.87倍和1.75倍。在NS处理中(图8-E), SoSnRK2.1基因的表达量在12 h出现第一次高值然后有所下降, 后又再次逐渐增加, 到72 h达最高值, 为对照的2.65倍。在CS处理中(图8-F), SoSnRK2.1基因表达量缓慢增长到48 h出现最高值, 为对照的2.23倍, 随后72 h表达量略下降。
ABA作为植物体内重要的生长激素, 不仅可以调节植物的生长发育, 且在对逆境胁迫的响应中也有着重要的作用, 且逆境胁迫响应机制的实现离不开其复杂的信号转导途径的调节[ 24, 25]。参与ABA受体PYL/PYR/RCAR介导的信号转导途径的相关酶类很多, 蛋白激酶SnRK2就属于其中一类, 参与了ABA信号转导途径中激活下游转录因子或膜蛋白等作用因子的磷酸化过程[ 26, 27]。
本研究克隆得到甘蔗 SoSnRK2.1基因全长, 其氨基酸序列与其他物种如玉米、水稻等具有很高的同源性, 尤其是与水稻中起着ABA信号转导作用的 SAPK3同源性极高。这些结果说明 SoSnRK2.1基因可能与水稻[ 28, 29]、小麦[ 30]及拟南芥[ 14]等植物中已研
究的 SnRK2基因的功能相似, 可以响应水分胁迫、渗透胁迫、低温胁迫等一系列逆境胁迫过程。分析甘蔗 SoSnRK2.1基因功能结构域发现, 在其氨基酸序列上存在ATP结合位点和蛋白激酶保守催化域, 分别具有11、3和8处潜在的丝氨酸、苏氨酸、络氨酸磷酸化位点。因此可以推断, 甘蔗 SoSnRK2.1基因属于典型的Ser/Thr蛋白激酶, 而存在的磷酸化位点和ATP结合位点可能与ABA信号转导途径中的蛋白磷酸化相关。此外, 本研究将甘蔗 SoSnRK2.1基因与原核表达载体pET30a(+)重组, 转入大肠杆菌BL21(DE3)中进行融合蛋白的诱导。分析表明, 甘蔗 SoSnRK2.1基因可在原核表达体系中得到正确表达, 为制备单克隆抗体及后期的研究打下基础。
SnRK2基因是ABA信号转导途径中的关键调控酶, 它的激活在植物响应渗透逆境胁迫时发挥着重要的作用[ 3]。本研究结果分析表明, 在受到各种外源胁迫时甘蔗叶片中 SoSnRK2.1基因均得到诱导表达, 而在根中则受到抑制表达。随着处理时间的增加, 甘蔗叶片 SoSnRK2.1基因在受到ABA、PEG+ABA、H2O2、PEG、NaCl胁迫时表达量都先有一个大的增幅, 有所下降后又逐渐上升, 而在受到低温胁迫时表达量缓慢增加, 推测可能是甘蔗根部有土壤的保护, 使来自外界的胁迫缓慢作用于甘蔗根部, 植物体内启动抗逆境的过程也就比较缓慢, 具体原因还有待进一步研究。由于H2O2、PEG、NaCl和4℃低温对甘蔗的胁迫与甘蔗受氧化胁迫、干旱胁迫、盐胁迫和渗透胁迫的效应相似。因此, 通过 SoSnRK2.1基因表达调控的复杂性可以推测甘蔗 SoSnRK2.1基因可能受到甘蔗抗逆境胁迫多个途径调控。目前在玉米[ 31]、拟南芥[ 32]、小麦[ 15]和蚕豆[ 4]等植物上对ABA信号转导途径和SnRK2蛋白激酶的研究已越来越多, 但至今尚未见有关甘蔗SnRK2蛋白激酶的研究在国内的报道。对该基因的上述研究可以为进一步研究甘蔗 SoSnRK2.1基因的功能及其在逆境胁迫中的调控作用奠定一定的基础。
甘蔗 SoSnRK2.1基因的全长为1385 bp, 完整的阅读框长度为1002 bp, 编码333个氨基酸, 预测分子量为37.8 kD, 为亲水性非跨膜蛋白质。该基因以融合蛋白形式表达, 相对分子量约为38 kD。不同外源胁迫下不同时间甘蔗叶片中 SoSnRK2.1基因都上调表达, 推测该蛋白在甘蔗抗逆境胁迫机制中发挥一定作用, 可作为研究甘蔗抗逆境的待选基因。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|