CDPK是一类重要的钙信号感受蛋白和响应蛋白, 在植物非生物胁迫应答方面具有重要的作用。为探究耐旱作物谷子CDPK在抗逆胁迫中的应答机制, 利用RT-PCR技术从谷子幼苗cDNA中克隆到一个与逆境胁迫相关的
CDPK (calcium-dependent protein kinase) is a kind of essential calcium sensors and calcium responders, which plays important roles in response to various abiotic stresses in plants. In this research, we cloned the abiotic stress response related
钙离子是真核细胞内重要的第二信使, 介导复杂的信号转导调控网络, 在植物生长、发育、免疫防御、激素调节、生物胁迫、非生物胁迫等各个方面起着重要的作用。目前, 主要有CaM (calmodulin), CBL (calcineurin B-like protein), CDPK (calcium-dependent protein kinase) 3类钙离子识别蛋白。其中, CDPK还是钙离子响应蛋白。CDPK是一类典型的Ser/Thr蛋白激酶, 结构较保守, 其蛋白主要包括4部分, 从N端到C端依次为可变区、蛋白激酶
区、自抑连接区、钙离子结合区, N端含有保守的MGXXXSXX区域, 能够被豆蔻酰化修饰, 从而将CDPK蛋白定位在细胞内膜系统上起重要的作用[ 1, 3, 4, 5, 6, 7, 8]。除了豆蔻酰化, 有的CDPK还能被棕榈酰化[ 4, 9], 增强与膜质结合的能力。自抑连接区中有CDPK自抑制片段, 可作为CDPK的假底物。当假底物与蛋白激酶区结合时, CDPK处于未激活状态[ 2]。钙离子结合区含有保守的钙离子结合EF手性结构域, 当钙离子与此结构结合后可以引起CDPK蛋白结构变化, 解除自抑, 从而激活CDPK[ 2]。不同的CDPK蛋白具有不同的钙离子结合能力, 而对于单个CDPK而言, 钙离子是调节CDPK活性的重要因子[ 2, 10]。
CDPKs为多基因家族, 拟南芥中含有34个[ 11, 12, 13], 水稻中含有31个[ 14, 15], 小麦中至少含有20个[ 16], 玉米中含有40个[ 17], 其他物种均包含多个 CDPKs基因。CDPK可在植物所有的组织和器官中表达, 但有的CDPK组织特异性表达, 如AtCPK17和AtCPK34主要在成熟的花粉中表达并调节花粉管的伸长[ 18]。CDPK可以被定位于不同的亚细胞结构, 如细胞质基质、细胞核、细胞膜、内质网、液泡、线粒体、叶绿体、油体、过氧化物酶体等[ 3, 5, 7, 16, 18, 19, 20, 21, 22, 23, 24, 25]。
从1991年, Harper等[ 26]从大豆中克隆得到第一个 CDPK-α基因全序列开始, 不同物种、不同功能的 CDPK基因被不断研究。研究证实[ 15, 16, 27, 28], CDPK能够响应不同的外界刺激, 包括ABA、冷、干旱、高盐、热、病原菌入侵和机械损伤等。 CDPK启动子含有响应不同胁迫的顺式作用元件[ 28]。CDPK的作用底物具有多样性, 包括酶、离子通道、水通道、转录因子(ABF4、RSG和HsfB2a)[ 24, 29, 30, 31, 32, 33, 34, 35, 36, 37]等。CDPK参与植物细胞内其他重要的信号通路, 包括ABA信号转导通路、ROS信号通路、茉莉酸信号通路、乙烯信号通路、水杨酸合成途径、非生物胁迫应答等。近年来, CDPK在抗逆应答方面的研究取得了一定的进展[ 1, 32]。CDPK调节植物激素和非生物胁迫信号转导途径中的相关基因表达、代谢、离子通道、气孔运动等[ 1]。CDPK是植物发育、免疫防御和胁迫应答的中心因子, 并参与其他重要的信号转导通路。不同的信号通路相互交错、互补、连接, 形成植物细胞内复杂而有序的调控网络。
谷子具有根系发达, 叶片细窄, 水分利用率高等耐旱性特征。谷子与粮食作物亲缘关系较近且基因组较小, 已经逐步成为未来作物遗传改良育种理想的模式作物[ 38], 将对未来禾谷类作物的品种改良、能源作物的遗传解析产生深远影响。目前, 已完成谷子全基因组测序, 同时谷子分子标记(SSR、RFLP、AFLP、SRAP等)与功能基因组学研究[ 48]也取得了一定的进展, 必将带动谷子研究的高潮。国内, 在谷子抗锈基因、抗病基因、抗除草剂基因等方面取得了一定的进展, 如病程相关基因 SiSGT1、NBS类抗病基因、抗锈基因 RUS、抗锈基因 RLr1、抗除草剂基因 Srf[ 39]、丝/苏氨酸蛋白激酶类抗病基因[ 40]等。在抗旱方面, 国内学者对谷子 SiDnaJ基因[ 42]、干旱应答元件结合蛋白基因 SiDREB[ 42]、12-氧代植二烯酸还原酶基因 SiOPR1[ 43]、3-磷酸甘油醛脱氢酶基因 GAPDH[ 44]、磷脂酶D基因 SiPLDa1[ 45]、 CBL家族[ 46]、 PP2C家族[ 38]和 ABF3基因[ 47]等进行了研究。但目前未见关于谷子 CDPK基因家族研究的报道。谷子抗逆的分子机制的深入研究将为利用基因工程、分子生物学育种奠定良好的基础。
本研究以晋谷34为试验材料, 利用RT-PCR技术克隆得到耐旱作物谷子中第一个与植物非生物胁迫应答相关的钙依赖性蛋白激酶基因 SiCDPK1, 拟通过蛋白预测、系统发育分析、启动子调控元件分析等生物信息学分析和考察在ABA、高盐、PEG和自然干旱胁迫下的表达模式, 以期为揭示谷子 CDPK基因家族抗逆分子机制奠定基础。
晋谷34由山西省农业科学院作物遗传研究所提供, 将种子播种于苗床(草炭土、珍珠岩和蛭石的质量比为1∶1∶1)上, 控制适宜谷子生长的空气湿度、温度、光照等条件。幼苗长至三至四叶, 取长势一致的谷子幼苗分别设PEG模拟干旱处理(含20% PEG的1/2 Hoagland营养液, pH 7.0)、高盐处理(含100 mmol L-1 NaCl的1/2 Hoagland营养液, pH 7.0)、正常干旱处理(断水)和外部喷施ABA (100 μmol L-1)胁迫处理。取PEG和ABA胁迫处理0、0.5、1、2、4和8 h后的样品, 正常干旱处理断水0、1、2、3、4、5和6 d后每天9:00的样品, 每个样品10株, 3次重复。将样品于液氮速冻, -80℃冰箱保存待用。
1.2 SiCDPK1基因编码区ORF全长cDNA克隆
从谷子转录组数据库中, 筛选出 SiCDPK1基因片段, 使用Trinity (release-20130225)进行序列的组装(默认参数), 利用DNAMAN软件(http://www. lynnon.com/)进行ORF搜索(ORF>50), 得到 SiCDPK1基因编码序列。根据此序列, 设计并合成引物 SiCDPK1-F和 SiCDPK1-R (表1)。取不同时期正常干旱(断水)处理、高盐处理、外部喷施ABA处理、PEG模拟干旱处理的植物材料混合样品, 用RNAiso Plus (TaKaRa)提取mRNA, 使用PrimeScript First Strand cDNA Synthesis Kit (TaKaRa)反转录合成cDNA。利用SiCDPK1-F和SiCDPK1-R引物进行PCR扩增 SiCDPK1基因编码区ORF全长cDNA片段, 回收特异性PCR扩增产物, 连接到pMD-18T Simple载体(TaKaRa)上, 转化到大肠杆菌(DH5α)感受态细胞中, 挑取抗性培养基上生长的阳性单菌落, 经质粒酶切验证后送华大基因测序, 得到目的基因 SiCDPK1全长cDNA。
| 表1 SiCDPK1基因克隆及Real-time PCR引物 Table 1 Primers used in SiCDPK1 gene cloning and Real-time PCR analysis |
用ProParam (http://espasy.org/tools/protparam.html)分析SiCDPK1的物理和化学性质。用Predict Protein (http://www.predictprotein.org/)和ProScale (http://expasy.org/tools/protscale.html) 预测SiCDPK1的二级结构及疏水区域。利用TMpred (http://www.ch.embnet.org/software/TMPRED_form.html)和TMH MM (http://www.cbs.dtu.dk/services/TMHMM)分析SiCDPK1的跨膜结构域。通过PROSITE (http://www.expasy.ch/Prosite)预测该蛋白质的结构域和功能位点。利用CCD数据库和PFAM数据库分析该蛋白的保守结构域和家族。以拟南芥 CDPK基因家族序列为查询序列, 与谷子基因组比对预测谷子CDPK基因家族成员, 利用ClastalX (默认参数)进行多重序列比对, 使用MEGA (重复次数1000)构建Neighbor- Joinig系统进化树。使用在线预测工具PlantCARE (http://bioinformatics.psb.ugent.be/webtools/plantcare/ html/)预测 SiCDPK1的启动子结构。
根据克隆得到的 SiCDPK1序列, 设计并合成Real-time PCR引物SiCDPK1-RT-F和SiCDPK1- RT-R。以 SiActin基因作为Real-time PCR内参, 设计引物SiActin-F和SiActin-R (表1)。
利用RNAiso试剂盒(TaKaRa, 大连)分别提取PEG模拟干旱处理、高盐处理、正常干旱处理和外部喷施ABA胁迫处理后材料的根、茎、叶的总RNA, 分别以总RNA作为模板, 使用PrimerScript RT Master Mix (Perfect Real-time)试剂盒(TaKaRa, 大连)反转录合成cDNA。取总RNA约200 ng于200 μL离心管中, 加入5× PrimerScript RT Master Mix (for Real-time) 2 μL, 用RNase Free dH2O将反应体系补充至10 μL。将PCR仪(TaKaRa)设定为37℃保温15 min, 85℃保温5 s, 降温至4℃保存。使用One Step SYBR PrimerScript RT-PCR Kit (Perfect Real- time)试剂盒(TaKaRa, 大连)进行PCR, 反应体系为2×SYBR Premix Ex Taq II (含荧光染料) 12.5 μL、10 μmol L-1引物各1 μL、ddH2O 8.5 μL和cDNA模板2 μL。以谷子 SiActin基因作为内参基因, 采用两步法进行实时荧光定量PCR扩增 (ABI7300, Applied Biosystems)。反应条件为95℃ 30 s; 95℃ 5 s, 61℃ 31 s (搜集信号), 40个循环。使用SDS软件分析实时定量PCR结果。
利用生物信息学和PCR技术克隆得到一个谷子抗逆应答相关的钙依赖蛋白激酶基因 SiCDPK1(GenBank登录号为KC249975.1)。通过NCBI (http://www.ncbi.nlm.nih.gov/) Blastn比对结果可知, SiCDPK1与玉米( Zea mays)、水稻( Oryza sativa)、小麦( Triticum aestivum)、大麦( Hordeum vulgare)、二穗短柄草( Brachypodium distachyon)等单子叶植物部分 CDPK基因具有高度的同源性, 具有 CDPK基因家族的基本特性。
将测序得到的 SiCDPK1序列与谷子基因组数据库(http://www.phytozome.net/)比对, 发现该基因位于谷子基因组Scaffold_2区段的41 740 734~ 41 743 777。使用DNAMAN对该区域的序列进行结构化展示(图2), SiCDPK1是由7个内含子和8个外显子组成的。
ProParam预测结果显示SiCDPK1蛋白由531个氨基酸组成(图3), 分子量为59.5 kD, 等电点p I为5.94。SiCDPK1蛋白含谷氨酸(E, 8.3%)、丙氨酸(A, 7.3%)、甘氨酸(G, 7.3%)、亮氨酸(L, 7.2%)和赖氨酸(K, 7.0%), 这5种氨基酸所占比例达到了37.1%。
Predict Protein二级结构预测显示, SiCDPK1蛋白含有15个蛋白结合位点, 17个螺旋结构helix, 11个串strand, 主要以螺旋结构为主。疏水区主要分布在蛋白N端的蛋白激酶区。用ProScale进行疏水性分析显示, 150~300氨基酸部分疏水性较集中, 与Predict Protein预测结果相符。利用TMpred和TMHMM分析, 均未发现明显跨膜结构, 表明SiCDPK1不是跨膜蛋白。PROSITE分析结果显示SiCDPK1蛋白含有丰富的可磷酸化位点(14个丝氨酸S、11个苏氨酸T、6个酪氨酸Y)。通过Signal IP预测发现SiCDPK1蛋白不含有信号肽结构, 说明该蛋白不是分泌蛋白。
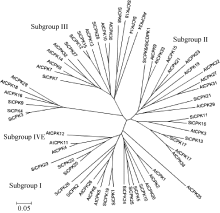
通过NCBI BlastP直接比对或利用CCD数据库的CD-search工具分析表明, SiCDPK1蛋白具有典型的CDPK保守结构, N端的蛋白激酶结构域属于PKc_like superfamily, C端含有4个EF hand结构域, 可形成2个钙离子结合loop。以已知的拟南芥 CDPK基因序列(TAIR,http://www.Arabidopsis.org/, release 10.0)为查询或搜索序列, 在谷子基因组数据库(http://www.phytozome.net/)中通过Blast找到预测的CDPK基因成员, 并通过谷子基因组数据库或NCBI数据库手动分析初步确定候选的谷子 CDPK基因家族成员有28个(表2)。从NCBI上下载拟南芥的34个 CDPK基因序列, 并结合预测到的谷子CDPK基因家族成员序列, 用ClastalX和MEGA4.0软件进行多重序列比对以及构建Neighbor-Joining系统进化树(图4)。从图4中, 我们看出, 谷子的 CDPK基因家族与拟南芥 CDPK基因家族一样, 可以分成4个亚类, 第IV亚类的基因数较少。 SiCDPK1/ SiCPK6属于 CDPK基因家族的第II亚类, 与 AtCPK9和 AtCPK33亲缘关系较近。目前, AtCPK9和 AtCPK33基因的功能并未被确定[ 1], 因此探讨 SiCDPK1基因的功能将推动 CDPK基因家族的研究进展。
| 表2 预测SiCDPK蛋白信息 Table 2 Information of predicted SiCDPKs protein |
从 SiCDPK1基因的ATG开始, 截取 SiCDPK1基因5°上游1969 bp的核苷酸序列, 使用PlantCARE在线预测 SiCDPK1基因的启动子及调控元件。ATG上游177 bp处发现TATA-box区域, 转录起始位点于上游212 bp处, 在上游210 bp至上游1487 bp区域预测到多种逆境胁迫相关的顺式作用元件(图5), 包括与光、温度、激素、厌氧、干旱、细胞周期及发育相关顺式调控元件。
2.2 SiCDPK1基因的表达模式分析
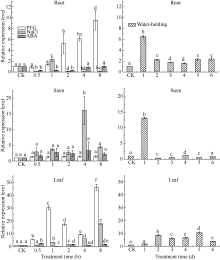
在用20%的PEG胁迫处理谷子幼苗后, SiCDPK1在谷子根和叶中表达量呈现显著上调的趋势, 分别在1 h和2 h时明显上调, 并有持续上调的趋势, 在8 h时表达量达到最大, 分别为对照组的9.5倍和46.2倍。而在茎部表达量则未出现明显的变化(图6)。高盐处理时, SiCDPK1在根中除了1 h出现上调外, 其余各点表达量下调。而在茎和叶中, 各个处理时间点 SiCDPK1表达量均呈现上调趋势。相对于PEG处理, 较晚响应高盐胁迫, 在茎和叶中分别在4 h和8 h表达量最高, 峰值分别为16.1倍和17.6倍(图6)。高盐处理时, SiCDPK1在不同组织中表达趋势的不同, 显示了 SiCDPK1复杂表达模式, 表达量的变化, 显示其在不同胁迫时期的响应。当用100 μmol L-1ABA处理后, SiCDPK1基因的表达量在根茎叶中变化不及PEG和高盐处理明显, 但仍出现变化, 显示其参与ABA信号通路(图6)。在正常干旱处理下, SiCDPK1基因在根茎叶中表达量先升高后下降。根和茎中, 均在第1天表达量达到最高, 分别为6.5倍和13.1倍。叶中, SiCDPK1基因在第2天表达量才明显上升, 于第5天达到最高值(图6)。
克隆得到的 SiCDPK1具有 CDPK家族的典型特征[ 13, 34], 谷子CDPK基因家族成员含有的EF hand结构域数量不同(表2中EF hand结构域数由谷子基因组数据库注释得到), 以3个EF hand结构域为主, 显示了不同SiCDPK活性受钙离子结合的调节[ 13, 35]。通过系统发育分析可知, 其属于 CDPK基因家族的第II亚类, 与其同源性较高的拟南芥CDPK家族成员有 AtCPK9、 AtCPK33、 AtCPK15、 AtCPK21、 AtCPK23等。其中 AtCPK9、 AtCPK33和 AtCPK15这3个基因的功能并未被确定, 而 AtCPK21已被证实参与植物渗透胁迫应答和气孔运动调控, AtCPK23被证实参与干旱和高盐胁迫应答、气孔运动调控。从本文实时定量数据分析可知, SiCDPK1/SiCPK6基因受PEG渗透胁迫的诱导, 对高盐胁迫产生应答, 从而表达量变化, 然而在不同的组织中其变化不同, 显示了 SiCDPK1基因具有组织特异性, 在不同组织中的功能也不尽相同。 SiCDPK1基因受ABA诱导表达, 与其启动子调控区域含有ABRE顺式作用元件相呼应。在自然干旱中, 我们可以看到 SiCDPK1表达量在最初2 d明显上升, 说明 SiCDPK1在谷子干旱胁迫应答中起一定作用。
AtCPK9、 AtCPK33、 AtCPK15、 AtCPK21和 AtCPK23基因, N端均能被豆蔻酰化和棕榈酰化[ 35], SiCDPK1蛋白N端同样具有豆蔻酰化和棕榈酰化的修饰位点(图3)。由于豆蔻酰化和棕榈酰化的修饰, AtCPK9、 AtCPK21和 AtCPK23被定位于细胞膜上, 推测 SiCDPK1也被定位于细胞膜上。
从 SiCDPK1启动子调控元件的分析可知, SiCDPK1基因很可能参与激素调节(GA、ABA)、光感应、厌氧应答、病原菌免疫、周期调控、胁迫应答和调控植物生长发育(胚乳、分生组织)等。推测 SiCDPK1与bZIP、WRKY等转录因子相关, 调控下游基因表达。由此可见, SiCDPK1在谷子的生长发育及逆境应答中具有重要的功能。
目前, 对谷子抗逆基因研究还处于初步阶段, SiCDPK1基因为证实的一个谷子抗逆相关的钙离子依赖蛋白激酶基因。植物的生长发育、对外界刺激的响应是一个复杂而综合的网络, 是CDPK、MAPK、CBL-CIPK、SnRK等通路[ 36, 37]相互作用、相互联系的结果。对CDPK生理功能和底物的研究, 有利于揭示植物抗逆胁迫的应答机制, 为提高植物的抗逆性奠定坚实的理论基础。目前, 较常用的研究方法和技术有基因敲除、酵母双杂、Microarray和BiFC等技术。同一物种中的不同CDPK具有不同的生理功能, 然而它们之间又存在着功能冗余的现象, 使得单独敲除或干扰单个CDPK并不能取得理想的试验效果, 双缺或三缺突变体成为研究的必要, 如 cpk4cpk11、 cpk3cpk6、 cpk7cpk8cpk32[ 33]。CDPK新的作用底物不断被发现, 然而大多数都是体外实验得到的[ 13], 而在体内筛选CDPK作用底物目前还不是很成熟。发现更多的CDPK特异性底物能够加速CDPK功能的鉴定, 系统和全面地对 CDPK基因家族进行分析、研究和整理, 合理综合使用生物化学、细胞生物学、遗传学、生物信息学等方法, 必将更好地推动CDPK信号转导系统的研究。
从谷子中克隆到一个与逆境胁迫应答相关的钙依赖蛋白激酶基因 SiCDPK1, 含有 CDPK基因家族典型的结构特征, N端的蛋白激酶结构域属于PKc_like superfamily, C端含有4个EF hand结构域。N端含有豆蔻酰化和棕榈酰化修饰位点, 推测 SiCDPK1定位于细胞膜上。 SiCDPK1含有多个与植物逆境胁迫相关和响应激素的调控元件, 如ABRE、ARE、Box-W1等, 推测SiCDPK1可能作用于bZIP、WRKY等转录因子, 从而调控下游基因表达。 SiCDPK1受PEG、ABA、高盐、自然干旱的诱导表达, 在谷子逆境胁迫应答中起重要作用。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|
| [26] |
|
| [27] |
|
| [28] |
|
| [29] |
|
| [30] |
|
| [31] |
|
| [32] |
|
| [33] |
|
| [34] |
|
| [35] |
|
| [36] |
|
| [37] |
|
| [38] |
|
| [39] |
|
| [40] |
|
| [41] |
|
| [42] |
|
| [43] |
|
| [44] |
|
| [45] |
|
| [46] |
|
| [47] |
|
| [48] |
|