RUB1 (related to ubiquitin 1)是植物和酵母中一种泛素类似蛋白质, 是Cullin家族的一个成员。为阐释津田芜菁 BrRUB1基因的表达特性, 本研究克隆了津田芜菁 RUB1基因的全长cDNA序列, 命名为 BrRUB1, GenBank登录号为KF501173。其ORF全长471 bp, 编码156个氨基酸; 亚细胞定位结果显示, BrRUB1-GFP定位于细胞核, 表明BrRUB1蛋白可能在细胞核中发挥其功能。qRT-PCR检测 BrRUB1的表达表明, 该基因表达量在花蕾中最高, 花瓣中次之, 具有组织特异性。而且 Br RUB1在芜菁根皮中的表达受长波紫外线(UV-A)诱导。
RUB1 (related to ubiquitin 1) is a ubiquitin-like protein in plant and yeast, belong to Cullin family. To elucidate the expression feature of RUB1 in Tsuda, we isolated cDNA of RUB1gene from this plant and named BrRUB1 (GenBank accession No. KF501173). The cDNA of BrRUBIwas 670 bp in full length and its open reading frame (ORF) was 471 bp in full length, encoding 157 amino acids. BrRUB1-GFP was localized to nucleus, indicating that BrRUB1 may play an important role in the nucleus. qRT-PCR analysis showed that BrRUB1 expressed highly in bud and less in petal, showing tissue specificity. Furthermore, the expression of BrRUB1 was induced by UV-A light in the swollen hypocotyls.
RUB1 (related to ubiquitin 1)是植物和酵母中一种类似泛素分子的蛋白质, 在哺乳动物中又称为NEDD8蛋白(neural precursor cell-expressed, developmentally down-regulated protein)。RUB1是泛素化E3连接酶家族Cullin家族的一个成员, 它可以对蛋白质肽链进行修饰, 也可以被参与调控植物体感受激素信号的蛋白AXR1 (auxin resistant1)和ECR1 (erythrocyte complement receptor type1)以异源二聚体的形式构成RUB激活酶, 与Cullin蛋白内保守的赖氨酸残基结合, 正调控Cullin蛋白组成的E3复合体, 加强E3复合体和Ub-E2复合体的稳定性, 影响生物体内多种调节蛋白的功能, 从而调控植物体生长发育与形态建成[1, 2, 3]。
RUB1作为泛素调控途径的一员, 除了参与植物生长发育和形态建成外, 还在包括植物膜受体的内在化、染色质异构、转录激活以及DNA修复、植物应对干旱和冷害等非生物胁迫反应中发挥功能[4, 5, 6, 7]。津田芜菁(Brassica rapaL. cv. Tsuda)膨大的肉质根不见光部分呈白色, 无花青素的合成; 而见光部分呈紫色, 有花青素的合成。单色光中只有在UV-A诱导下可合成花青素[8]。本文根据已经报道的拟南芥序列, 通过RT-PCR结合RACE技术克隆了芜菁BrRUB1基因, 并对该基因的定位和表达进行研究, 以期为深入探讨RUB1在芜菁中的功能提供参考依据。
样品为温室中生长至60 d的津田芜菁(Brassica rapaL. cv. Tsuda)膨大根部的白色根皮(不见光)、红色根皮(自然光下)、叶片, 以及长至5个月的津田芜菁花瓣和花蕾(0.8 cm), 另取60 d的津田芜菁白色根皮用UV-A光(352 nm, 15 W m-2)处理1、3、6和24 h。将样品放在液氮中速冻后于-80℃超低温冰箱贮存待用。将UV-A光照处理的根皮(1 cm× 1 cm)置色素提取液(含1%盐酸的甲醇溶液)中, 4℃放置24 h[9]。测定样品在530 nm的光吸收。以OD值表示花色素苷的含量, 重复测定3次, 生物学重复3次。
以芜菁根白色根皮、UV-A处理过的白色根皮, 红色根皮、叶片、花瓣和花蕾为试验材料, 采用CTAB法提取各组织总RNA, 检测RNA质量和浓度后, 取红色根皮1 µ g的总RNA用于cDNA第1链的合成。
参照TaKaRa公司的RNA PCR Kit (AMV) Ver. 3.0试剂盒说明书合成cDNA第1链。依据GenBank中亲缘关系相近的物种拟南芥AtRUB1基因的核苷酸序列, 应用PrimerPremier 5.0设计引物BrRUB1-F: 5′ -GAGAATTTAAGCAAGAAAAG-3′ ; BrRUB1-R: 5′ -ACAGCAAAGCATCAGACC-3′ , 由华大生物公司合成。以cDNA第一链为模板。PCR条件为94℃ 3 min 预变性, 94℃ 30 s, 52℃ 30 s, 72℃ 1 min, 30个扩增循环, 最后72℃延伸10 min。PCR扩增产物经0.8%琼脂糖凝胶检测、回收目的条带, 与pGEM-T Vector (Promega)链接, 16℃过夜, 连接产物转化到大肠杆菌TOP10, 在X-gal/IPTG琼脂糖平板上挑选阳性克隆, 并进行PCR和酶切鉴定, 采用Amersham phamacia Biotech Inc.提供的MegaBACE 500毛细管自动测序仪测序。测序引物分别为pGEM-T载体引物T7和SP6, 完成测序。
在已获得ORF序列的基础上利用cDNA末端快速扩增(RACE)法克隆3'-DNA序列[10], 以红色根皮1 μ g总RNA为模板, 以oligo(dT)17接头引物5′ -GACTCGAGTGCACATCG(T)17-3′ 及反转录酶III (Invitrogen)合成第一链cDNA。参考已获得的ORF序列设计的5′ 引物BrRUB1-F: 5′ -ATTGATCGCA TCAAGGAGCG-3′ 和接头引物5′ -GACTCGAGTGC ACATCG-3′ 进行PCR扩增。扩增条件为94℃预变性5 min, 随后30个循环, 每循环94℃变性30 s、54℃退火扩增30 s、72℃延伸90 s, 完成最后一个循环后, 72℃延伸10 min。PCR扩增产物经1%琼脂糖凝胶检测, 回收纯化、克隆、质粒提取、测序同cDNA克隆。
根据获得的基因序列设计拼接引物BrRUB1- cDNA-F: 5′ -GAGAATTTAAGCAAGAAAAG-3′ 和接头引物克隆该基因全长cDNA。PCR产物的回收纯化、克隆、质粒提取、测序同cDNA克隆。
BrRUB1基因序列同源性比对由NCBI的nucleotide BlastN程序进行(http://www.ncbi.nlm. nih.gov/blast/)。BrRUB1保守性功能域分析由NCBI的protein BlastP程序进行(http://www.ncbi.nlm. nih.gov/blast/)。核定位信号分析由SignalP4.1程序预测(http://www.cbs.dtu.dk/services/SignalP/)。进化树的构建由MEGA5程序完成。
取津田芜菁新鲜的嫩叶, 用CTAB法提取基因组DNA。根据所得的BrRUB1全长cDNA序列, 分2段设计特异性引物BrRUB1-D1-F: 5′ -ATGCAGAT CTTCGTCAAAACCCTGAC-3′ 、BrRUB1-D1-R: 5′ -T CCCCACCAAAACCAGAAGAGATC-3′ , 用LA Taq DNA聚合酶(TaKaRa)进行PCR扩增。反应条件为94℃变性30 s, 55℃退火30 s, 72℃延伸2 min, 35个循环后, 72℃延伸7 min。PCR产物经回收, 插入peasy-T1载体, 转化到大肠杆菌Top10中, 选择阳性克隆测序。
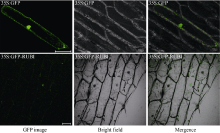
用高保真酶和加有酶切位点的引物对测序验证过的含有目的基因的质粒DNA进行扩增, 凝胶电泳回收目的基因片段。用Sal I和Spe I双酶切PCR产物和pA7-GFP vector, 经回收、连接、转化、提取质粒DNA鉴定。利用真空渗透的方法将构建好的重组质粒(瞬时表达载体)转入洋葱表皮细胞, 取1.5 cm× 1.5 cm该洋葱表皮, 在培养基上面铺一层滤纸, 培养洋葱表皮细胞时, 培养16 h, 用Zeiss 510 nete激光共聚焦扫描显微镜观察。以激发波长为488 nm和505~550 nm扫描图像, 使用Zeiss LSM软件将其抓获, 并使用Adobe Photoshop软件处理系统, Mountain View, CA[11]。
用7500 Real time PCR System (Applied Biosystems, 美国)检测BrRUB1基因在芜菁膨大根的白色根皮、红色根皮、叶片、花瓣、花蕾以及UV-A处理不同时间芜菁根白皮中的表达。所用BrRUB1引物为BrRUB1-F: 5′ -ATTATCCCTTCAAGCCACC-3′ ; BrRUB1-R: 5′ -AACGAGCAGATCGACAGC-3′ 。内标BrActin引物为BrActin-F: 5′ -GCTCAGTCCAAGAG AGGTATTC-3′ ; BrActin-R: 5′ -GCTCGTTGTAGAAA GTGTGATC-3′ 。所选用的试剂为ABI公司的High- Capacity cDNA Reverse Transcription Kits及Power SYBR Green PCR Master Mix。20 µ L反应体系中包括10 µ L Power SYBR Green PCR Master Mix, 8.4 µ L cDNA(稀释后), 正向引物和反向引物各0.8 µ L (10 µ mol L-1)。采用的Real-time PCR程序为95℃ 30 s预变性, 40个扩增循环(95℃ 10 s, 60℃ 34 s), 从60℃加热到90℃的熔解过程。以BrActin的转录表达水平为内参, 目的基因相对转录表达水平为2-Δ Ct, Δ Ct=Ct (目的基因) - Ct (Actin)。每个样品设3个试验重复, 生物学重复3次。
克隆到的BrRUB1基因的cDNA全长为650 bp, ORF区为471 bp, 编码156个氨基酸, 基因组全长945 bp, 含有3个外显子和2个内含子, 内含子为174 bp (intron1)和121 bp (intron2) (图1)。蛋白质基本性质分析工具(ProtParam)分析显示, BrRUBI蛋白质的分子量为17.413 kD; 等电点(pI)为5.77, 其中带负电荷的氨基酸残基为25个, 带正电荷的氨基酸残基有23个, 分子式为C771H1283N211O240S2, 原子总数是2507; 不稳定系数为38.15, 属于稳定蛋白; 代表亲水性的GRAVY值约为-0.383; 表明BrRUBI为亲水性蛋白; 蛋白质结构域的预测显示, BrRUB1具有UBQ-E2泛素结合酶位点, 与泛素、NEDD8高度相似, 属UBQ超家族。
 | 图1 津田芜菁BrRUB1基因核苷酸序列及其推导的氨基酸序列(框内为内含子)Fig. 1 cDNA and deduced protein sequence of BrRUB1 from Tsuda (introns are boxed) |
从NCBI数据可搜索拟南芥、玉山筷子芥和小麦等9个物种的10个RUB基因, 氨基酸序列同源性分析表明, BrRUB1与这10个RUB的同源性都很高, 达到90%以上。其中BrRUB1与同为双子叶、十字花科植物拟南芥的At RUB1(NP_564379.2)同源性最高达96%, 与玉山筷子芥的Al RUB1 (XP_002891151.1)同源性为95%, 与拟南芥的At RUB2(NP_565812.1)同源性为94%, 它们4个RUB1在进化上属于同一个进化分支。BrRUB1与其他科植物的RUB1同源性也很高, 与野生稻(XP_006660632.1)、谷子(XP_ 004965723.1)、大豆(NP_001276242.1)、番茄(XP_ 004248366.1)的同源性都为95%, 与小麦(EMS64626.1)、玉米(NP_001148606.1)的同源性都为94%, 这6个物种的RUB1则属于另一个进化分支(图2)。
对BrRUB1氨基酸序列进行核定位信号预测, 结果显示该蛋白不具有核定位信号。我们构建了BrRUB1-GFP瞬时表达载体(图3), 将其转入洋葱表皮细胞中, 并获得了瞬间表达。利用激光共聚焦显微镜检测表明, 绿色荧光集中分布在细胞核中, 说明BrRUB1蛋白定位于细胞核(图4)。
2.3.1 津田芜菁BrRUB1基因组织特异性表达
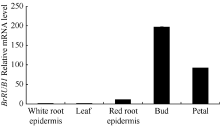
提取芜菁不同组织来源的总RNA, 以芜菁的Actin基因作为内参照, 对BrRUB1基因qRT-PCR分析。显示(图5), 津田芜菁BrRUB1在花蕾中表达量最高, 在花瓣中次之。在有花青素合成的红色根皮中有少量表达, 在叶和白色根皮中表达量最低。说明BrRUB1基因在芜菁中的表达有较高的组织表达特异性。
2.3.2 UV-A光诱芜菁根皮中花青素含量检测
温室中长至60 d的津田芜菁, 取白色根皮进行UV-A光照诱导花青素合成, 分别取照射0、1、3、6和24 h后的样品检测。在UV-A光照诱导1 h后花青素含量未见明显升高, 3 h后有微量花青素开始合成, 并且在6 h和24 h后花青含量持续增高(图6)。
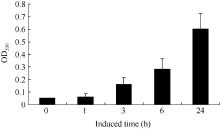 | 图6 UV-A光照诱导芜菁白色根皮花青素合成含量.Fig. 6 Content of anthocyanin in white root epidermis of Tsuda induced by UVA |
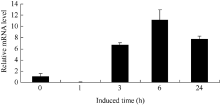
2.3.3 UV-A光诱导BrRUB1在津田芜菁白根皮中的表达 津田芜菁根皮花青素合成受UV-A光的诱导(图6)。我们提取UV-A处理1、3、6和24 h后的津田芜菁肉质根皮总RNA, 以芜菁的Actin基因作为内参照, 对Br RUB1基因qRT-PCR分析显示(图7)白皮中BrRUB1的表达受UV-A光诱导, 长于1 h的使BrRUB1表达量增加, 处理6 h后表达量最高, 而处理24 h后BrRUB1的表达量较处理6 h明显下降。表明UV-A诱导BrRUB1的表达与UV-A光诱导花青素合成含量相一致, 花青素合成和BrRUB基因的表达趋势相比存在滞后, 这可能与启动花青素合成相关基因表达需要一定时间有关。
本研究克隆了芜菁泛素化相关基因BrRUB1基因, BrRUB1基因含有3个外显子和2个内含子。内含子的边界并不符合GT-AG规则, GT-AG规则主要适用于真核生物基因的剪接位点, 但是也有例外, 比如甘蓝中硫氧还蛋白编码基因BoTHL1, 沙冬青低温诱导基因AmCIP等基因的内含子也不符合[12, 13]。此外, 这种保守性也不适用于线粒体、叶绿体和酵母tRNA基因转录后的加工。该基因的cDNA序列含有一个471 bp的开放读码框, 编码一个156个氨基酸的蛋白质。氨基酸序列分析表明, BrRUB1是泛素(UBQ)类似物, 是RUB/Nedd8家族的成员, BrRUB1氨基酸序列中具有保守的泛素结合酶作用位点。氨基酸同源性分析结果表明, 芜菁BrRUB1与拟南芥AtBrRUB1的同源性最高, 与拟南芥AtBrRUB1、玉山筷子芥AlBrRUB1、拟南芥的AtRUB2在进化上属于同一个分支。虽然在BrRUB1氨基酸序列中没有预测到核定位信号, 但是研究结果表明BrRUB1蛋白定位于细胞核, 可能在细胞核中发挥功能。就像拟南芥的MAX2 (more axillary growth2)蛋白一样, 也属于泛素代谢途径蛋白, 其氨基酸序列中也没有预测到核定位信号, 但它定位在细胞核, 在细胞核中发挥功能[14], 它们可能都通过核定位信号以外的机制进入细胞核。
BrRUB1基因在芜菁中的表达具有较高的组织表达特异性, 在花蕾中表达量最高, 在花瓣中次之, 在叶和白色根皮中表达量最低。RUB1蛋白的活力可以影响CUL4-DDB1复合体形成, 进而影响COP1 (constitutively photomorphogenic 1, COP1)的功能[15]。泛素化相关蛋白COP1可以直接调控与开花相关的重要转录因子CONSTANS(CO)发挥功能, CO可以接受长日照信号启动开花。黑暗条件下COP1可以降解CO, 使植物在日照长度不足的条件下不启动开花进程[16]。RUB1在开花过程中起到了一定的调控作用, 这也可能是BrRUB1在花蕾和花瓣的表达量相对高的原因。
在有花青素合成的根皮中, RUB1基因也有少量的表达。且在UV-A光诱导下花青素合成, RUB1基因的表达也被诱导表达, 说明自然光和UV-A光诱导下RUB1蛋白都参与了花青素合成的调控。苯基苯乙烯酮合酶(chalcone synthase, CHS)是花青素合成途径中的第一个关键的酶, 津田芜菁根皮花青素合成中CHS基因的表达量增加了[8], 而RUB1蛋白可能通过其参与调控的途径影响着CHS基因的表达, 进而影响花青素的合成和积累。已经有研究表明, RUB1蛋白的活力能够影响CRL (CULLIN-RING E3 ligases)的活性与稳定性, 而CRL是一类依赖于Cullin蛋白的E3连接酶[17], 可以介导包括HY5 (ELONGATED HYPOCOTYL 5)在内的多种与植物光信号通路有关正调控因子的降解[18]。而且COP1作用于目的基因需要HY5作为桥梁。黑暗中, COP1蛋白进入细胞核, COP1蛋白与转录因子HY5等结合, 使这些转录因子失活或离开其作用区即CHS、叶绿素a/b结合蛋白CAB等基因启动子区, 使CHS、CAB等基因不能表达。在光下, COP1蛋白脱离结合的转录因子, 转录因子活化, 基因开始表达[19]。这是否是在光下RUB1蛋白参与调控花青素合成的原因, 有待进一步验证。
克隆了津田芜菁BrRUB1基因, 该基因基因组全长945 bp, 含有3个外显子和2个内含子, ORF区为471 bp, 编码156个氨基酸。BrRUB1蛋白定位于细胞核。BrRUB1基因表达具有组织特异性, 且在UV-A光诱导的花青素合成中表达量增加。
The authors have declared that no competing interests exist.
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|