碱性亮氨酸拉链(basic leucine zipper, bZIP)是真核生物特有的转录因子家族, 在高等植物基因表达与调控中起重要作用。本文通过对玉米基因组数据库中收录的全长cDNA序列全基因组范围的bZIP分析, 发现其中25个序列编码D亚族bZIP转录因子。针对这些序列进行生物信息学分析、染色体分布和直系同源组的分类分析, 发现部分序列可能是由同一基因座编码, 但不同方式剪切形成的。对部分基因克隆和测序发现了更多的剪切形式。定量检测其中3个基因在ABA和干旱胁迫条件下的表达发现, 其中1个受ABA诱导, 2个受ABA抑制, 但均不受干旱胁迫影响。表明玉米中D亚族基因可能参与ABA响应。
Basic leucine zipper (bZIP) is unique transcription factor family among eukaryotes, playing important roles in gene expression and regulation in higher plants. Here we reported the finding of 25 full length cDNA sequences encoding D subfamily of bZIP factors in maize from a database mining on whole genome level. The analysis of bioinformatics, chromosome distribution and classification of Possible Groups of Orthologous (PoGO) suggested the existence of diverse alternative splicing in some of these genes, which was supported by the sequencing results of the cloned cDNAs. The expression pattern of three representative genes in response to ABA and drought stress were examined by quantitative RT-PCR, and the results revealed different regulations of ABA on the expression of these genes. Our results suggest that D subfamily of bZIP genes in maize might involve in ABA signal pathway.
碱性亮氨酸拉链(basic region/leucine zipper motif, bZIP)类的转录因子普遍存在于动植物及微生物中, 参与ABA信号转导和各种环境胁迫的响应等, 是一类重要的转录因子[ 1]。它由一个二聚体化作用的亮氨酸拉链区域和一个DNA结合的碱性结构域组成。亮氨酸拉链结构域的典型特征是每7氨基酸的第7位含有一个亮氨酸, 以及其他疏水残基位于第3和第4位。DNA结合区域是由大约20个氨基酸组成的碱性结构域, 用于识别并结合G-box等顺式作用元件[ 2]。bZIP类转录因子在未结合DNA之前以未折叠的单体形式存在, 受到逆境胁迫后以二聚 体的形式结合DNA, 识别核心序列为ACGT的顺式作用元件[ 3]。Jakoby等[ 4]根据碱性亮氨酸区域的同源性和其他保守结构域的特性, 将拟南芥基因组中所有的bZIP类转录因子分成A、B、C、D、E、F、G、H、I和S类10个亚族, 认为在同一亚族的bZIP转录因子往往有相似的功能。拟南芥中D亚族有10个成员[ 4], 水稻中有16个[ 5]。目前对不同植物中D亚族bZIP转录因子的研究表明, 该亚族基因主要参与病害防御和生理生长, 还参与生长发育进程[ 4]。Correa等[ 5]进一步提出了直系同源组(Possible Groups of Orthologues, PoGO)的分类, 其中将植物D亚族bZIP又分为5个组, 即D1~D5。其中D2和D4组主要参与生长发育进程, 如拟南芥中AtbZIP46/ Perianthia控制植物器官数目[ 6], 玉米的 Liguleless2 ( lg2)定位叶和鞘的边界[ 7]; D3和D5组主要在植物防卫反应过程中起正向和负向双重调控作用[ 5], 如拟南芥中TGA4/OBF4/AtbZIP57可能通过AtEBP与PR启动子上的乙烯应答元件相结合起抗病作用[ 8, 9], 而TGA类型的bZIP转录因子与响应水杨酸(salicylic acid, SA)的顺式元件相结合, 在SA信号途径中起负调控作用[ 10]。此外, D3和D5组基因还参与茉莉酸(jasmonic acid, JA)信号转导以及生长激素和磷缺陷的响应等[ 11]。D1类的基因功能目前尚不清楚。
对拟南芥D亚族bZIP转录因子的研究表明, 该亚族的氨基酸序列具有极高的同源性, 如TGA4和TGA1氨基酸相似性达92%, TGA5、TGA6和TGA2的相似性分别达92.8%和95.3%, TGA4和TGA5有53%的氨基酸相同[ 8, 12]。玉米中分离出2个bZIP蛋白基因OBF3.1和OBF3.2, 其氨基酸相似度达95.8%[ 13]。不同植物中的该类基因保守性也极高, 拟南芥TGA1和烟草TGA1a有63%氨基酸相同[ 14]。
进化研究表明, D亚族的基因与A、G亚族可能有共同的祖先[ 5]。但几乎所有A亚族的基因都参与ABA信号转导, 并耐受抗旱等多种非生物胁迫[ 4], 而D亚族是否参与ABA信号和干旱等非生物胁迫响应尚无报道。本文利用基因组数据挖掘方法分析玉米D亚族bZIP基因, 克隆并研究其在ABA和干旱胁迫条件下的表达, 为进一步阐明bZIP转录因子D亚族的功能提供基础。
将玉米( Zea maysL .) B73自交系种子置于湿润纱布上, 28℃暗培养2 d后, 种植于搅拌均匀的花土和蛭石的小花盆中, 在28℃、12 h/12 h光周期条件培养。取三叶一心期的幼苗, 将根部浸没在 100 μmol L-1 ABA中进行处理, 并以溶剂作为对照, 于0、2、6和12 h处理后取叶片。同时, 三叶一心期时开始停止浇水, 进行干旱处理, 用土壤水分探测仪测定土壤水分含量。水分饱和时土壤水分含量在40%~50%之间, 15%~20%为轻度干旱(feebly arid, FA), 10%左右为中度干旱(moderate drought, MD), 5%左右为重度干旱(serious drought, SD), 并以同期生长的正常浇水的玉米幼苗作为对照(control check, CK), 按各处理分别取样, 立即用液氮处理并置-70℃保存。
用总RNA提取试剂盒(TRIzol Reagent, TaKaRa)提取样品的总RNA, 并用DNase I (TaKaRa)纯化。按照SuperScript III反转录酶(Invitrogen)说明书合成第一链cDNA, 作为基因扩增及荧光定量PCR(Real time quantitative PCR, qRT-PCR)的模板。
用特异引物和高保真的Phusion High-Fidelity DNA聚合酶(NEB)进行基因扩增, Touchdown程序反应程序为98℃ 30 s; 98℃ 10 s, 62℃ 20 s, 72℃ 45 s, 退火温度每隔一个循环降低1℃, 至49℃; 最后延伸72℃ 10 min。PCR产物经1%琼脂糖凝胶电泳检测, 回收目的条带。连接Gateway载体pENTR/ D-TOPO (Invitrogen), 转化大肠杆菌感受态TOP10, 经菌液PCR筛选阳性克隆, 测序验证。
采用Toyobo公司SYBR Green Master Mix进行qRT-PCR, 反应体系含cDNA 1 μL、7.5 μL 2×SYBR Green Master Mix、引物(10 pmol μL-1)各0.3 μL、ddH2O 5.9 μL。以玉米β-tubulin为内参, 反应程序为94℃ 30 s; 94℃ 5 s, 54℃ 30 s, 72℃ 20 s, 共40个循环。采用基因相对表达分析2-∆∆Ct法分析基因相对表达量[ 15]。
利用ZmGDB (http://www.plantgdb.org/ZmGDB/)和MaizeGDB (http://www.maizegdb.org/)数据库进行玉米bZIP基因全长cDNA和基因组序列搜索以及染色体位置和外显子-内含子结构分析。bZIP蛋白多重序列比对由ClustalX 1.83构建, 使用软件默认参数[ 16], 根据比较结果, 用Genedoc进行保守序列分析, 用MEGA4构建系统进化树[ 17]。以上述玉米基因组数据库为依据, 应用MEME (http://meme.nbcr.net/meme/ cgi-bin/meme.cgi)软件进行保守结构域分析。
将ZmGDB和MaizeGDB基因组数据库中收录的可能编码bZIP转录因子的全长cDNA序列与拟南芥D亚族基因进行序列比对分析, 发现25个与拟南芥D亚族基因同源性比较高的玉米bZIP序列。对这
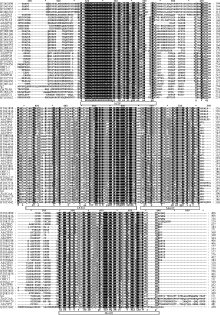
25个基因编码产物分析发现它们与拟南芥D亚族bZIP转录因子相似, 除了共有bZIP结构域外, 紧跟着bZIP基元还有3个保守的motif (图1)。其中第一个motif是DOG1 (Delay of Germination 1)保守结构域[ 18], 该结构域曾在一个种子休眠关键基因 DELAY OF GERMINATION 1 ( DOG1)中发现, 但具体功能未知。
很多报道指出D亚族基因氨基酸相似度较高。通过序列比对, 发现25个D亚族bZIP转录因子两两之间的氨基酸序列相似度在55%~99%之间, 蛋白的亮氨酸拉链区的下游至C末端的氨基酸序列高度保守(图2)。然而, 蛋白的N末端氨基酸序列差异很大, 可能与这些蛋白的不同功能或调节性质相关。
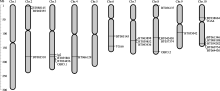
在序列比对中发现一些玉米D亚族bZIP基因具有完全相同或高度相似的编码序列。为了解这些基因之间的关系, 分析了这25个cDNA所对应的染色体位置, 发现除了第1和第5染色体外, 它们分布于其他8条染色体, 第10染色体上最多, 有7个基因; 第4和第9染色体上最少, 只有1个基因; 其余的5条染色体各有2~4个(图3)。同时我们根据基因组序列和cDNA序列比对, 分析相应基因的内含子-外显子结构, 发现D亚族基因内含子较多, 在5~12个之间(图4)。
发现有6组基因具有完全相同或高度相似的编码序列, 并位于同一个染色体的同一个基因座上(表1), 分别为: (1)BT088188、BT061346、BT034282、BT060754与BT064430, (2)BT043436、BT039852与BT063898, (3)BT037374与BT042450, (4)BT088145与BT069355, (5)BT038664与TGA4, (6)BT035801、BT034456与OBF3.1。其中第一组基因都位于第10染色体的144 522 640~144 530 848之间, 位置很接近。进一步分析这些基因的内含子-外显子结构发现, BT060754、BT064430的内含子-外显子结构也一样, 仅5-UTR和3-UTR序列不同; 二者氨基酸序列与BT034282相似性达到100%, 但内含子-外显子结构不同; BT088188和BT061346内含子-外显子结构相同, 但与其他3个不同。表明这5个基因可能是同一基因座编码的不同剪切方式造成的不同转录本。BT088145和BT069355都位于第2染色体6 717 439~ 6724101之间, 仅前者比后者多出N端的27个氨基酸, 其他序列完全相同, 但二者的内含子外显子结构差异很大; 都位于第7染色体133 128 880~ 133 137 073之间的BT039852比BT043436和BT063898多了N端50个氨基酸和1个内含子; 都位于第3染色体187 322 690~1 887 336 485之间的BT034456、BT035801和OBF3.1的外显子内含子结构相同, 但后者5-UTR短。另外2组基因分别位于第8染色体120 135 342~120 139 090区段和第10染色体49 981 532~49 984 883区段, 组内基因内含子外显子结构完全一样, 仅5-UTR和3-UTR序列不同。表明这5组内的不同基因很可能是预测得到的多种可能的转录本, 也有可能是不同剪切方式造成的。
另外, 位于第8染色体上的OBF3.2与位于第3染色体上的OBF3.1、BT035801和BT034456有较高的同源性, 第3染色体上的lg2与第6染色体上的TGA6有较高的同源性, 表明玉米基因组中的bZIP基因可能发生过复制。
| 表1 玉米D亚族bZIP基因的PoGO分组和染色体分布 Table 1 PoGO group and chromosomal distribution of D subfamily bZIP genes in maize |
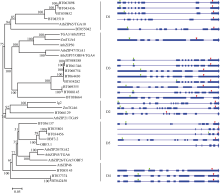
为了解玉米D亚族bZIPs的进化特征, 对玉米和拟南芥的D亚族bZIP转录因子构建了进化树(图4)。结果表明, 这些转录因子在进化树上的分布符合Correa等的PoGO分组[ 5]。其中, BT063898等3个基因座编码的5个bZIP序列与AtbZIP65构成D1组分支; 另外ZmTGA6等3个玉米bZIP因子与AtbZIP21组成D2分支; BT088188等3个基因座编码的8个bZIP序列组成一个小分支, 与AtbZIP22等4个拟南芥bZIP因子和玉米TGA4的小分支共同形成D3组分支; BT085145等2个基因座编码的3个玉米bZIP序列与AtbZIP46组成D4分支; OBF3.1等3个基因座编码的5个bZIP因子组成一小支, 与AtbZIP20等3个拟南芥基因组成的小分支共同组成D5组分支。表明拟南芥和玉米的5个组分别具有共同祖先, 但在2种植物中平行进化, 在玉米中大量复制进化形成多个拷贝, 而在拟南芥中除D3组外都维持较低的拷贝数。
对玉米D亚族5个PoGO组的代表性bZIP基因进行扩增和克隆测序。获得的5个基因扩增产物中D3组的BT034282和D5组的BT034456和BT036137基因序列与预测的完全相同, D5组BT035801测序结果比预测长63个核苷酸(编码21个氨基酸的序列), 即预测的第1个内含子存在于转录本中; 而D2组BT066129核酸序列与公布序列相比有8个碱基差异, 导致其中1个氨基酸不同。因基因克隆时选用高保真的DNA聚合酶进行扩增, 错配率非常低; 而且多个克隆测序发现此差异是所有克隆中共同存在的, 因此可以确定差异是真实存在的, 而不是PCR和克隆过程造成的。D5组的BT034456和BT035801存在于第10染色体的同一基因座, PCR与测序结果表明该基因确实存在不同剪切形式的转录本。上述结果表明这些基因在玉米幼苗中是转录的。然而D1组和D4组的基因没有得到扩增产物, 可能与这些基因在三叶一心期的幼苗中不表达, 或者表达水平低有关, 也可能是其所在区段的染色体结构特殊或GC含量较高所致。
由于BT034282可能是BT088188的不同剪切形式, BT036137与BT035801相似度极高, 无法获得特异性扩增, 故仅分析了D5组的BT036137、D3组的BT088188和D2组的BT066129这3个 bZIP基因应答ABA和干旱胁迫的表达谱。结果如图5所示, ABA处理后BT036137被诱导表达, 且表达量随着时间的延长有逐渐升高的趋势, 12 h可达到0 h的2倍以上; 而BT088188和BT066129的转录表达受ABA胁迫的抑制, 且12 h表达量最低(图5-A)。干旱处理后, 这3个基因的表达均受到抑制, 随着干旱的加强, 表达量逐渐降低, 甚至几乎不表达(图5-B)。
bZIP转录因子在ABA和植物逆境反应中扮演着重要角色, 干旱、高盐及其他逆境条件下, 植物细胞内源ABA含量迅速增加, 促使大量ABA诱导基因表达, bZIP转录因子可以与ABA诱导基因的启动子的ABRE(ABA-responsive element)结合来调节下游靶基因的表达[ 19]。A亚族基因在ABA和胁迫下的表达和在逆境基因表达调控方面的功能已被证实, 其中代表性的基因包括 ABI5及 ABI5-like、 ABF1- ABF4和 AREB3等。D亚族是除A亚族外唯一具有44个氨基酸组成的bZIP domain的亚族, 与A亚族可能从共同的祖先进化而来[ 5]。但对D亚族 bZIP基因的研究主要限于生物胁迫、植物发育以及参与JA和SA信号转导等[ 11], 对ABA和干旱等非生物胁迫的响应和所具有的功能尚无报道。
对拟南芥、水稻和毛果杨bZIPs基因的进化研究表明, bZIPs基因家族的祖先在单双子叶植物分化之前就已经存在[ 5]。本研究通过对玉米bZIP转录因子的全长cDNA序列分析, 得到25个D亚族玉米bZIP基因; 进化树分析结果表明, 这25个玉米D亚族bZIP转录因子与拟南芥D亚族基因聚到一起, 推测玉米与拟南芥bZIP转录因子很可能存在共同的祖先。这25个玉米D亚族bZIP基因分布于第8条染色体上, 且有6组为同一基因座编码的基因, 另外两组基因高度同源但分布于不同染色体, 每组组内基因序列甚至内含子-外显子结构都完全一样, 猜测可能是一个基因座编码的不同剪切方式的转录本。这可能与玉米bZIPs基因在进化过程中经历了整个基因组复制事件、染色体区域内的大片段复制事件以及接下来的基因缺失、小范围内的复制、局部重排、转座子跳跃、染色体倒置等事件有关, 使玉米D亚族的基因呈一定程度的重复性和冗余性。这些都表明D亚族bZIP基因在植物中是高度保守的。对拟南芥和玉米家族的保守结构域分析发现, 除典型bZIP motif外, 所有D亚族bZIPs中都含有1个DOG1保守结构域, 该结构域曾在一个种子休眠关键基因 DELAY OF GERMINATION 1 ( DOG1)中发现, 但具体功能未知。DOG1是与种子休眠相关的关键基因, 其受ABA调控[ 18, 20]。最新研究表明DOG1不仅影响萌发还影响开花时间, 进而影响植物整个生命过程和适应能力[ 21]。外显子捕获、重排和融合是新基因产生的进化机制之一[ 22]。该亚族基因是否通过进化获得DOG1 motif进而获得新功能, 有待进一步研究。
目前的研究表明D2和D4组主要参与生长发育进程, D3和D5组bZIP转录因子在植物防卫反应中起调控作用, 参与JA、SA信号转导以及生长激素的响应等[ 5], 本文报道ABA处理可诱导D5组BT036137表达, 而抑制D3组BT088188和D2组BT066129的表达, 表明玉米中D亚族基因也参与ABA响应, 但是调控方式不同, 也表明D亚族的基因尽管相似度极高, 但可能在ABA途径中的作用可能不同。
得到25个D亚族玉米bZIP基因, 其序列存在较大的重复性和冗余性, 均含有1个DOG1保守基序, 暗示该亚族基因与种子休眠、开花时间等可能有一定的关系。该亚族中BT036137基因受ABA诱导表达, 表明其可能参与ABA信号转导。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|





 50 are not described in the literature. Using common domains, the AtbZIP family can be subdivided into ten groups. Here, we review the available data on bZIP functions in the context of subgroup membership and discuss the interacting proteins. This integration is essential for a complete functional characterization of bZIP transcription factors in plants, and to identify functional redundancies among AtbZIP factors.
50 are not described in the literature. Using common domains, the AtbZIP family can be subdivided into ten groups. Here, we review the available data on bZIP functions in the context of subgroup membership and discuss the interacting proteins. This integration is essential for a complete functional characterization of bZIP transcription factors in plants, and to identify functional redundancies among AtbZIP factors.