叶绿体在植物碳水化合物代谢中起着至关重要的作用, 然而有关碳水化合物代谢在叶绿体发育过程中的功能却知之甚少。从水稻中克隆了一个
Chloroplast plays an important role in plant carbohydrate metabolism; however the function of carbohydrate metabolism in the chloroplast development is poorly understood. The
近年来, 越来越多的证据表明碳水化合物代谢在很大程度上影响着植物的生长发育和对环境因子的响应[ 1, 2, 3, 4, 5, 6, 7]。光合作用的最初产物磷酸丙糖在果糖- 1,6-二磷酸酶等的催化下可以转变成蔗糖。当水稻中的果糖-1,6-二磷酸酶功能缺失时, 植物光合速率下降, 生长发育缓慢, 并且在四叶期停止发育[ 5]。叶绿体膜上的麦芽糖转运子(MEX1)是叶绿体中淀粉降解产物转运的主要通道。 MEX1基因突变后, 叶绿体中的麦芽糖不能被及时运出, 导致麦芽糖大量积累。 mex1突变体淀粉合成和降解的速度都变慢, 同时植株生长缓慢, 叶绿素含量降低[ 6]。与上述可溶性碳水化合物不同, 淀粉在植物中主要作为能量的储存形式。然而, 近来的研究表明淀粉的生物合成和代谢也调控着植物的生长发育。当拟南芥中2个编码淀粉分支酶的基因 AtSBEIIa和 AtSBEIIb同时丧失功能时, 植物淀粉含量显著降低、生长发育缓慢、植株矮小, 并且花序萎蔫[ 8]。在植物体内, 淀粉大多以磷酸化的形式存在, α-葡聚糖水合激酶( α-glucan waterdikinase, GWD)是淀粉磷酸化的关键酶。番茄 legwd突变体由于丧失了GWD活性, 致使花粉中的淀粉粒不能正常磷酸化。这些非磷酸化的淀粉在花粉粒发育后期无法被降解利用, 导致花粉败育死亡[ 4]。此外, 淀粉的合成和代谢与植物的生物产量密切相关, 是植物生长发育的调控枢纽[ 7]。蔗糖合酶是蔗糖代谢途径中的关键酶, 花生蔗糖合酶基因 AhSuSy的表达受到干旱胁迫的诱导, 可能在花生干旱胁迫中起一定的作用[ 1]。
糖苷水解酶(EC3.2.1.-)是一类广泛存在于生物体内可以水解糖苷或寡糖糖苷键的酶。根据其氨基酸序列, 糖苷水解酶被分为133个家族(http://www. cazy.org/fam/acc_GH.html)。其中, 糖苷水解酶13家族是一个比较常见的家族, 它包括淀粉分支酶和糖原分支酶等[ 9]。我们曾研究了糖苷水解酶13家族的AtBE1的功能[ 2]。与野生型相比, 拟南芥 AtBE1部分丧失功能的突变体 atbe1-3主根变短、侧根数目变少、子叶变绿延迟、叶片和花畸形; 而该基因完全丧失功能导致胚胎在心形胚时期发育停滞[ 2]。进一步的研究表明, 蔗糖、葡萄糖和果糖等游离糖类可以部分恢复 atbe1-3的表型, 暗示糖苷水解酶AtBE1可能通过碳水化合物代谢调控拟南芥胚胎发生和胚后发育[ 2]。此外, 该基因在植物离体器官再生过程中也起着极其重要的作用, atbe1-3突变体不定芽再生和体细胞胚胎发生的能力严重受阻[ 3]。序列分析表明, AtBE1与拟南芥淀粉分支酶AtSBE2.1和AtSBE2.2的一致性仅为40%左右, 而与水稻OsBE1的一致性为65%。拟南芥为双子叶植物而水稻为单子叶植物, 单双子叶植物中 BE1基因的功能可能不尽相同。本文对单子叶植物水稻 OsBE1基因的功能研究表明水稻 OsBE1基因功能缺失后导致叶绿体发育异常, 水稻幼苗白化, 在三叶期死亡。本研究为深入解析 OsBE1基因的功能奠定了基础。
突变体 osbe1 (PFG_1C_15429)来自韩国庆熙大学植物系统生物技术系(Department of Plant Systems Biotech, Kyung Hee University)的T-DNA插入突变体库[ 10, 11, 12]。从种子播种发芽到三叶期, 均在25℃、16 h/8 h (光照/黑暗)的条件下培养。在中国水稻研究所富阳实验基地进行水稻大田种植。
采用康为世纪生物科技有限公司的RNA提取试剂盒(货号CW0559, 含有DNase I)提取水稻总RNA。利用总RNA和宝生物(大连)有限公司的第1链合成试剂盒(PrimeScript II 1st Strand cDNA Synthesis Kit, 货号6210A)合成cDNA。利用半定量RT-PCR检测突变体 osbe1中 OsBE1基因的表达情况, 检测所用的引物为OsBE1F2: 5'-GGGAAGGG CAAGAACCAGAT-3'和OsBE1B2: 5'-CCTCATCGA CGTATTGATTG-3'。
由于 OsBE1基因非常大(32 kb左右), 很难进行分子操作, 固采用 OsBE1基因的cDNA。首先, 利用KOD-Plus Ver.2高保真DNA聚合酶(Toyobo Life Science, 货号KOD-211)和引物OsBE1F: 5'-tcccccggg TTCCCGTTCCTCCTTCCTCC-3'、OsBE1B: 5'-gctctaga GCTTGGTCTATCAACAATGC-3'扩增 OsBE1基因。将PCR产物用 Sma I和 Xba I双酶切后连接到pCAMBIA1300-35S-GFP-OCS载体(中国科学院遗传与发育生物学研究所储成才研究员馈赠)的相应酶切位点。构建成pCAMBIA1300-35S-OsBE1-OCS载体用于水稻的遗传转化。所有操作都按照试剂盒提供的方法。
运用Lasergene 11.0的MegAlign软件分析序列的亲缘关系, 采用MEGA 6.0软件分析保守氨基酸, 将结果拷贝到Photoshop进行编辑。
采用PCR的方法分离和鉴定 osbe1突变体。其中, pGA2707载体T-DNA引物PGAL序列为5'-AAAGTGGCACGTGGTGAATGG-3'。 OsBE1基因T-DNA侧翼序列的引物分别为LL (5'-TGCGCTCTC TTCCTCTCTAC-3')和LR (5'-CCACTCCATGAAATC CACAC-3')。野生型中引物LL和LR可以扩增出条带, 引物PGAL和LR扩增不出条带; 杂合突变体中LL和LR以及PGAL和LR都可以扩增出条带; 而纯合 osbe1突变体中引物LL和LR不能扩增出条带, 引物PGAL和LR可以扩增出条带。
采用分光光度计法测定叶绿素含量: 取二叶期幼苗的地上部分, 称重(W)后加入3 mL提取液(丙酮和无水乙醇等量混合液), 利用宁波新芝Scientz-48高通量组织研磨器充分研磨; 10 000× g离心5 min, 上清液转移至新的离心管中; 加入1 mL提取液, 震荡混匀后离心; 将2次提取的上清液混合后定容至4 mL(V); 利用分光光度计测定645 nm和663 nm的吸光度。叶绿素 a的含量(mg g-1) = (12.70 OD663- 2.690 OD645)×W×(1000×V)-1, 叶绿素b的含量 = (22.90 OD645- 4.86 OD663) × W × (1000 × V)-1, 叶绿素a、b的总含量 = (8.02 OD663 + 20.20 OD645) × W × (1000 × V)-1。
切片材料取自二叶期野生型和突变体幼苗叶片的相同部位。水稻叶片以2.5%的戊二醛固定液4℃固定过夜, PBS漂洗; 1%锇酸固定液固定4 h左右, PBS漂洗; 乙醇梯度脱水, 环氧丙烷置换, 环氧丙烷和Epon812树脂的混合液浸透2 h, 纯Epon812树脂浸透过夜、包埋, 干燥箱中聚合48 h; 利用LEICA EM UC6超薄切片机切片, 切片厚度为70 nm; 醋酸双氧铀和柠檬酸铅双染色; JEM-1400透射电镜观察拍照。
将上述构建好的质粒通过电击转化到农杆菌EHA105中, PCR检测阳性的克隆用于水稻转化。参照Wang等[ 13]的方法进行农杆菌介导的水稻基因转化。
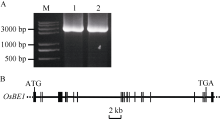
为了获得水稻 OsBE1基因的序列, 利用拟南芥AtBE1 (登录号为NP_001154629)与GenBank中的序列比对分析, 结果表明AtBE1与水稻Os06g0367100基因编码的蛋白质(登录号为NP_001057611.1)同源, 其一致性(Identities)为65%, 因此将该基因命名为 OsBE1 ( Oryza sativa Branching Enzyme 1)。以OsBE1F和OsBE1B为引物, 籼稻9311的cDNA为模板, 利用KOD-Plus Ver.2高保真酶进行PCR, 可以扩增出一条2~3 kb的条带( 图1-A)。随后对该条带测序分析发现, 该条带全长为2797 bp, 编码903个氨基酸。将该序列与水稻基因组序列比对分析表明, OsBE1基因含有22个外显子, 21个内含子( 图1-B)。但与一般基因不同, 该基因的终止密码子TGA不是在最后一个外显子上, 而是在倒数第2个外显子上( 图1-B), 这与拟南芥中调控开花的 FCA基因(AT4G16280)类似。
利用Pfam在线基序分析软件(http://pfam.xfam.org/)[ 14]分析OsBE1的功能表明, 该蛋白的127~220氨基酸残基为Carbohydrate-binding module 48结构域(E = 8.9e-8), 该结构域具有与碳水化合物结合的活性, 存在于碳水化合物相关活性的酶中; 425~557氨基酸残基为α-淀粉酶结构域(E = 0.0035), 该结构域可以水解淀粉等多糖中的α键; 802~899为α-淀粉酶C结构域(E = 3e-17)。这些结果暗示 OsBE1可能编码了一个糖苷水解酶13家族的蛋白。ChloroP 1.1[ 15]预测结果表明, 该蛋白N端为41个氨基酸残基的信号肽, 定位于叶绿体中。
进一步的序列比对分析表明, OsBE1与拟南芥AtBE1 (NP_001154629)、玉米ZmBE1 (AFW83197)、谷子SiSBEI (XP_004966509)、黄瓜CsBE1 (XP_ 004139870)和番茄SlBE1 (XP_004244099)等的一致性都在60%以上。然而, OsBE1与水稻典型淀粉分支酶OsSBEI (AAP68993, 820)、OsSBEIIa (BAA82828)和OsSBEIIb (BAA03738)的一致性较低, 仅为40%左右。为了深入了解BE1与典型淀粉分支酶SBEI和SBEII的亲缘关系, 从GenBank中获得了水稻、玉米、谷子、拟南芥、黄瓜和番茄中的典型淀粉分支酶。其GenBank登录号分别为: 水稻OsSBEI (AAP68993)、OsSBEIIa (BAA82828)、OsSBEIIb (BAA03738); 玉米ZmSBEI (ABQ15209)、ZmSBEIIa(DAA37957)、ZmSBEIIb (AAC33764); 谷子SiBE1 (XP_004965411)、SiSBEI(XP_004966509)、SiSBEIIa (XP_004975543)、SiSBEIIb (XP_004952629); 黄瓜CsSBEI (XP_ 004144236)、CsSBEII (XP_004137878); 番茄SlSBEI (XP_004238426)、SlSBEII (XP_004246561); 拟南芥AtSBE2.1 (NP_181180)和AtSBE2.2 (NP_195985)。利用Laser Gene11.0的Megalign软件包对上述序列进行亲缘关系分析, 结果表明BE1与典型淀粉分支酶SBEI和SBEII属于不同的家族, 而且单双子叶植物的BE1分别属于两个不同的亚类( 图2)。这暗示BE1家族可能具有不同于典型淀粉分支酶的功能, 单双子叶植物中 BE1基因的功能也可能不完全相同。
 | 图2 不同物种BE1和典型淀粉分支酶的系统进化树Fig. 2 Phylogentic relationship between BE1 and the clasical starch branching enzymes in different species |
为了深入了解BE1蛋白家族的功能, 我们对水稻、玉米、谷子、拟南芥、黄瓜和番茄中的BE1蛋白进行了多序列比对分析。如 图3箭头所示, 共有52个保守的氨基酸在单双子叶植物BE1之间存在差异, 这与上面系统进化树分析结果是一致的。此外, 还发现在淀粉分支酶SBEI和SBEII中都保守的105个氨基酸与BE1中的相应氨基酸不同( 图3), 暗示BE1可能具有不同于淀粉分支酶的新功能。
为阐明 OsBE1基因在水稻生长发育过程的功能, 从韩国庆熙大学植物系统生物技术系的T-DNA插入突变体库(http://cbi.khu.ac.kr/RISD_DB.html)中索要了 OsBE1的T-DNA插入突变体(PFG_1C_15429)[ 10, 11, 12]。T-DNA插入在该基因的第1个内含子中( 图4-A)。从索要的部分株系中分离到纯合的 osbe1突变体( 图4-B)。利用半定量RT-PCR检测了 osbe1纯合突变体中 OsBE1基因的表达情况, 结果表明在该突变体中检测不到 OsBE1基因的转录本( 图4-C), 暗示T-DNA的插入完全破坏了 OsBE1基因的功能。
从韩国庆熙大学植物系统生物技术系获得了12个株系的可能的 OsBE1基因的T-DNA插入突变体种子。将其播种后, 有4个株系出现白化幼苗( 图5-A)。叶绿素含量分析表明, 突变体中叶绿素 a、叶绿素 b和总叶绿素的含量都较低, 分别为野生型的3.67%、11.85%和5.87% ( 图5-B)。进一步的分析表明, 野生型和 osbe1突变体中叶绿素 a和叶绿素 b的比值分别为2.71和0.84, 暗示 OsBE1基因的突变对叶绿素 a的合成影响更大。 osbe1突变体在三叶期后即停止发育, 最终死亡( 图5-C)。由于外源可溶性糖类可以部分恢复拟南芥 atbe1-3突变体生长发育缺陷的表型[ 2], 那么外源糖类是否可以拯救 osbe1突变体幼苗白化或致死的表型呢?为了回答这一问题, 将这些杂合株系的种子播种在含有2%蔗糖的MS培养基上, 观察 osbe1突变体的生长发育情况。但结果表明, 蔗糖不能拯救突变体幼苗白化或者致死的表型( 图5-D)。
为了阐明 osbe1突变体白化苗的表型是否是 OsBE1基因突变引起的, 利用单株收获的种子中含有白化苗的株系连锁分析表明, 所分析的62株幼苗中, 白化苗与正常幼苗的比例为1∶3 (14株白化∶48正常); 14株白化幼苗全部为 OsBE1基因T-DNA插入的纯合体, 而48株正常株系中18株为无T-DNA插入的野生型植株, 30株为含有T-DNA插入的杂合植株。这些结果表明 OsBE1基因的突变导致了水稻幼苗白化。生物信息学分析表明, 与OsBE1一致性最高的已知活性的酶是淀粉分支酶( 图1), 该酶活性的丧失有可能导致淀粉含量的变化。但碘/碘化钾染色表明, osbe1萌发的幼苗中淀粉含量与野生型无显著差异( 图5-E)。
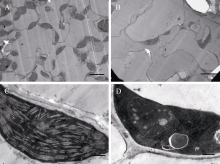
osbe1突变体幼苗白化, 在三叶期死亡, 暗示该突变体叶绿体的发育可能受到影响。为验证这一猜测, 利用透射电镜观察了 osbe1突变体叶绿体的发育情况。与野生型相比, 突变体中叶绿体的数目明显减少, 平均每个视野为5个叶绿体, 而野生型为25个( 图6-A, B)。另外, 尽管 osbe1的叶绿体轮廓完好, 但内部结构简单, 缺乏丰富、高度组织化的内膜系统, 且无基粒和基粒片层的分化( 图6-C, D)。
碳水化合物代谢是植物体内最重要最基础的代谢之一, 在植物生长发育过程起着至关重要的作用。我们曾对拟南芥 AtBE1基因进行了深入研究, 该基因编码一个糖苷水解酶13家族蛋白, 通过碳水化合物代谢调控了植物胚胎发生和离体器官再生[ 2, 3], 但具体机制尚不清晰。本文从水稻中克隆了一个 AtBE1的同源基因 OsBE1, OsBE1与AtBE1的一致性为66%, 而与水稻中淀粉分支酶的一致性仅为40%左右。系统进化分析表明, OsBE1与谷子、玉米、番茄和黄瓜等的BE1亲缘关系较近, 而与经典的淀粉分支酶SBEI和SBEII较远( 图1), 暗示OsBE1可能是不同于淀粉分支酶的一个新的糖苷水解酶13家族蛋白。这与Han等[ 16]的研究结果是一致的。
系统进化树表明, 单子叶植物水稻OsBE1、玉米ZmBE1和谷子SiBE1序列聚为一个亚类; 而双子叶植物拟南芥AtBE1、黄瓜CsBE1和番茄SlBE1聚为一个亚类( 图2)。多序列比对分析也表明单双子叶植物BE1序列之间存在一定的差异( 图3)。上述结果暗示单双子叶植物中 BE1基因的功能可能存在一定的差异。这一推论在随后的功能研究中得到证实: 双子叶植物拟南芥中 AtBE1完全丧失功能后导致胚胎败育, 部分丧失功能突变体虽然叶片发育畸形, 但叶色基本正常[ 2]; 而单子叶植物水稻 OsBE1完全丧失功能后, 叶绿体发育异常, 幼苗白化( 图5和 图6)。另外, 外源糖类可以部分恢复拟南芥 AtBE1基因部分功能丧失突变体 atbe1-3的表型[ 2], 但却无法拯救水稻 osbe1突变体( 图5)。进一步研究表明, 过量表达水稻 OsBE1导致幼苗黄化( 图7-B), 暗示水稻 OsBE1基因的表达量受到严格的调控, 过高过低都会导致植物生长发育异常。当该基因功能缺失时, 叶绿素合成完全受阻, 幼苗白化; 而当该基因过量表达时, 叶绿素的合成部分受阻, 幼苗黄化。但具体原因尚待进一步研究。
我们对拟南芥AtBE1初步的活性分析表明, AtBE1可能不具有典型的淀粉分支酶活性[ 2]。此外, 在植物体内淀粉主要作为储藏物质而存在, 因此淀粉的合成和代谢相关酶类的缺失一般不会导致植物的死亡。拟南芥基因组中编码两个典型的淀粉分支酶即BE2.1和BE2.2 (又称为BE2和BE3), 即便是这两个编码基因全部突变, 支链淀粉的合成完全被阻断, 双突变体也可存活, 只是生长发育缓慢[ 8]。除了淀粉分支酶, 支链淀粉的生物合成还需要可溶性淀粉合酶(soluble starch synthases, SSs)和淀粉脱分支酶(debranching enzymes, DBEs)的共同参与[ 17]。水稻淀粉脱分支酶ISA3功能缺失后导致造粉体和叶绿体发育异常, 表明 ISA3基因参与了质体发育的调控[ 18]。但与水稻 ISA3基因相比, 拟南芥和水稻中 BE1基因对于叶绿体的发育可能更为重要。
从水稻中克隆了一个 OsBE1基因, 该基因编码一个糖苷水解酶13家族蛋白。 OsBE1基因功能缺失突变体 osbe1幼苗白化, 叶绿体发育异常, 暗示了该基因通过碳水化合物代谢调控了叶绿体的发育, 但具体机制还待进一步的生化功能研究。
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|